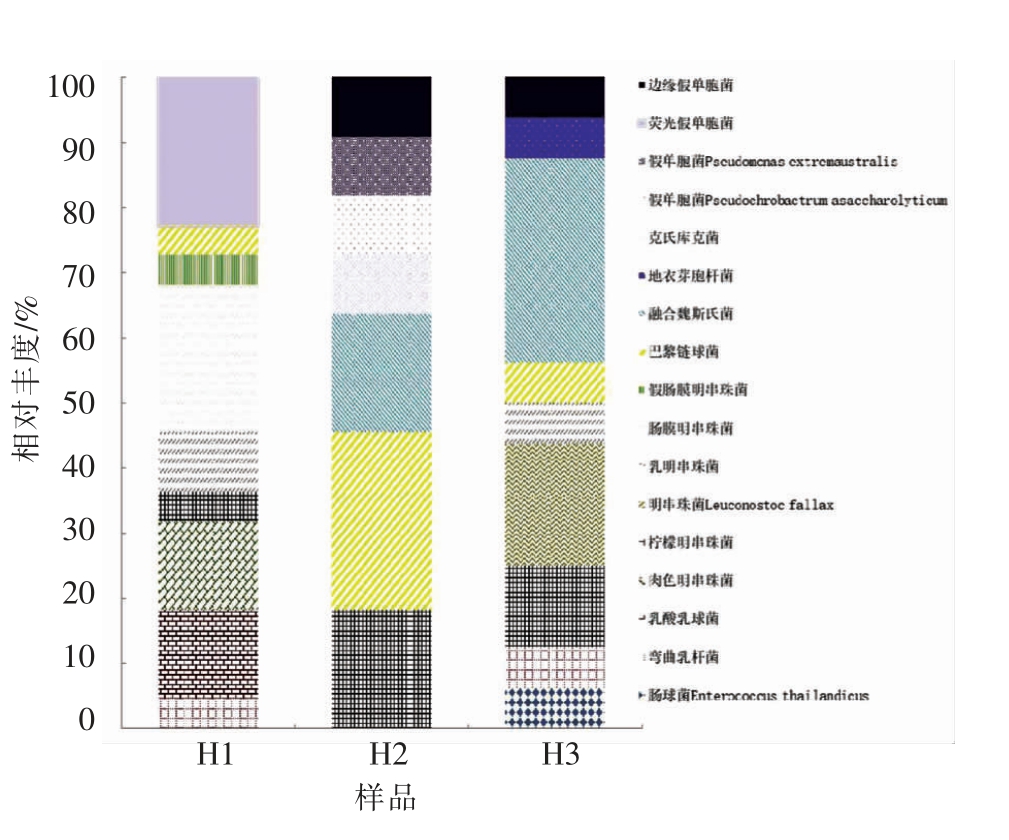
图1 基于细菌16S rRNA 测序分析的相对丰度
Fig.1 Relative abundance,based on bacterial 16S rRNA sequencing
臭豆腐是中国传统的发酵豆制品,具有悠久的历史。臭豆腐的制造过程因地区而不同,以杭州为代表的南方地区的臭豆腐先要制作臭卤水[1]。在水中添加蔬菜、肉类或草药混合,经过自然发酵而成,即利用自然界存在的菌种进行发酵,也叫作“敞口发酵”[2-3]。待卤水成熟后,将豆腐浸泡在其中数小时至数天不等,以获得富含“臭味”的成品。
目前对于臭豆腐中的微生物的多样性研究,主要还是应用传统的分离培养的方法。然而,这种分离培养的方法在研究复杂的微生物菌群时,不能很好地反映其中所有微生物的组成,导致低估了菌群数量和多样性[4]。高通量测序技术与传统的分离培养方法相比具有较为明显的优势,在微生物群落结构的研究方面定量准确、实时检测等[5-7]。目前,高通量测序技术广泛应用于微生物群落结构的探索方面,为探索与发酵食品相关的微生物种群多样性提供了巨大的帮助[7-9]。
到目前为止,对于臭豆腐中的微生物资源的研究相对较少,这种传统小吃中的微生物组成尚不清楚。本研究的目的是将高通量测序技术与传统的分离培养方法结合,分析臭豆腐中的乳酸菌的多样性,了解其微生态系统,为获取乳酸菌资源以及规范臭豆腐的生产奠定基础。
样品取自杭州市不同地点的臭豆腐;MRS 培养基、 营养琼脂培养基,杭州微生物试剂有限公司;Axygen 细菌基因组DNA 提取试剂盒,杭州爱思进生物技术有限公司。
YQX-Ⅱ型厌氧培养箱,上海新苗医疗器械制造有限公司;Powerpac Basic 电泳仪、 ALS-1296 PCR 仪,BioRad 生命医学产品有限公司;5417R冷冻离心机,德国Eppendorf 公司;ChamGel 5000凝胶成像仪,北京赛智创业科技有限公司。
1.3.1 细菌的分离纯化 无菌条件下称取臭豆腐样品10 g,加入到 90 mL 灭菌后的生理盐水(0.85%,m/V)中,搅拌至样品彻底均匀悬浮,取1 mL 悬浮液10 倍梯度稀释至10-7。取梯度10-5~10-7,从每个梯度吸取100 μL 样品稀释液均匀涂布于MRS 和营养琼脂(NA)培养基上,分别在30 ℃下培养48~72 h。选取合适的稀释度(菌落数在30~300 之间),挑选疑似不同的菌株,经3~5 次划线纯化后获得纯菌落。对得到的纯培养物进行过氧化氢酶试验和革兰氏染色,并镜检观察。将过氧化氢酶阴性,且革兰氏染色阳性的菌株初步视为乳酸菌;反之,为其他种类细菌。
1.3.2 DNA 提取与PCR 采用试剂盒提取细菌的基因组DNA,并进行PCR 扩增。其中PCR 25 μL 反应体系为:10×buffer(Mg2+)2.5 μL,dNTPs(2.5 mmol/L)2.0 μL,正、 反向引物各1.5 μL,ExTaq DNA 聚合酶 (5 U/μL)0.25 μL,DNA 模板2 μL,无菌去离子水16.75 μL。扩增引物为16S rDNA通用引物27F 和1492R。PCR 条件为:94 ℃预变性5 min;94 ℃变性30 s,56 ℃退火30 s,72 ℃延伸60 s,30 个循环;72 ℃终延伸10 min。
1.3.3 琼脂糖凝胶电泳检测与DNA 测序 PCR产物经0.8%(m/V)琼脂糖凝胶电泳检测后,送生工生物工程(上海)股份有限公司测序。后与NCBI数据库进行序列比对。
1.3.4 高通量测序 根据Axygen DNA 提取试剂盒说明直接从臭豆腐样品中提取总基因组DNA。通过通用引物 (F:5’-CCTACGGGAGGCAGCAG-3’,R:5’-TCCTCCGCTTATTGATATGC-3’)扩增16S rRNA 基因的V3-V5 高变区,用以下程序以25 μL 体系PCR:98 ℃预变性4 min,98 ℃变性30 s,55 ℃退火40 s,72 ℃延伸60 s,27 个循环,72℃终延伸7 min。使用454 GS FLX+系统(Roche 454 Life Sciences,USA)基因测序和生物信息学分析均委托派森诺生物科技有限公司完成。比对数据库为SILVA 和NCBI。

图1 基于细菌16S rRNA 测序分析的相对丰度
Fig.1 Relative abundance,based on bacterial 16S rRNA sequencing
在NA 与MRS 培养基中,共分离到49 株菌(图1)。通过16S rRNA 基因测序,最终分离到了11 种乳酸菌,分别是肠球菌 (Enterococcus thailandicus),弯曲乳杆菌(Lactobacillus curvatus),乳酸乳球菌 (Lactococcus lactis),肉色明串珠菌(Leuconostoc carnosum),柠檬明串珠菌(Leuconostoc citreum),明串珠菌(Leuconostoc fallax),乳明串珠菌 (Leuconostoc lactis),肠膜明串珠菌(Leuconostoc mesenteroides),假肠膜明串珠菌(Leuconostoc pseudomesenteroides),巴黎链球菌(Streptococcus lutetiensis)和融合魏斯氏菌(Weissella confusa)。此次样品中,分离到的乳酸菌大部分属于明串珠菌属(Leuconostoc)。这与之前的文献中报道乳杆菌属 (Lactobacillus)和肠球菌属(Enterococcus)是臭豆腐中常见的细菌菌群[3,10-11]不同。这很有可能是不同地区臭豆腐卤水在原材料的选择、 制作工艺、 环境差异等多种因素造成的。
卢义伯等[2]采用定性、定量培养的方法,从臭豆腐的卤水中分离出的优势菌株荧光假单胞菌(Pseudomonas fluorescens),在本试验中也分离得到。此外,本试验还分离到了Pseudomonas extremaustraliss 和边缘假单胞菌(Pseudomonas marginalis)两株假单胞菌。假单胞菌能分解蛋白质,是造成食物腐败的菌种之一,这可能是臭豆腐特殊气味的形成原因[2,12-13]。文献中提到的芽孢杆菌(Bacillus)属[14-16],此次也分离到了一株地衣芽胞杆菌(Bacillus licheniformis)。目前虽无报道详细说明芽孢杆菌与乳酸菌的相互作用及其对臭豆腐风味和营养有影响,但是芽孢杆菌属的一些菌种可以产生淀粉酶、透明质酸和纤维蛋白溶酶,在一些大豆发酵食品中有重要的作用,对人类健康有益[15,17]。此外,还分离到了一株克氏库克菌(Kocuria kristinae),这种菌主要来自于自然环境,是一种极罕见的条件致病菌[18]。
2.2.1 稀释曲线 稀释曲线 (Rarefaction curve)是每个样本中随机抽取一定量的序列,统计这些序列所代表的OTU 数目,并以序列数与OUT 数来构建曲线。它可以用来说明样本的测序数据量是否足以反映环境中的物种多样性,也可以用来比较不同样本中的物种多样性情况。图2横坐标为OTU 数目,纵坐标是样本序列数,而样品的曲线趋于平坦,说明测序数据足以覆盖所有的微生物。对3 个样品的丰度进行比较可知,样品中微生物的多样性丰富程度依次为H1、H3、H2。
2.2.2 丰度分布曲线 丰度分布曲线(Rank abundance curve)通常取物种的序数为横坐标,丰度值为纵坐标,它可以反映样品中物种的分布规律,即物种丰度和物种均匀度。图3中横坐标表示OTU 等级,纵坐标代表OTU 的丰度值。H1 样品的曲线比其他样品的曲线稍缓,说明H1 中的微生物分布更均一。
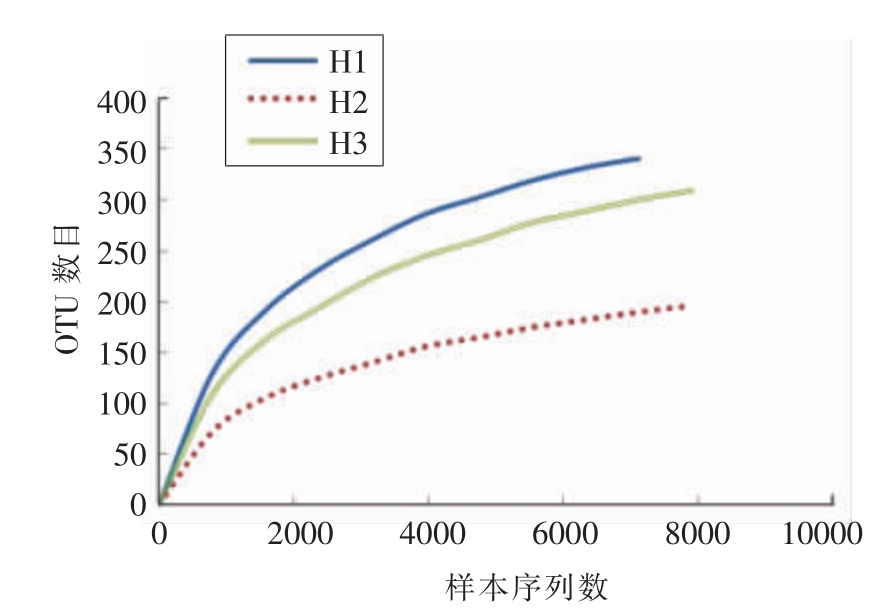
图2 样品稀释曲线
Fig.2 Rarefaction curves from stinky tofu samples
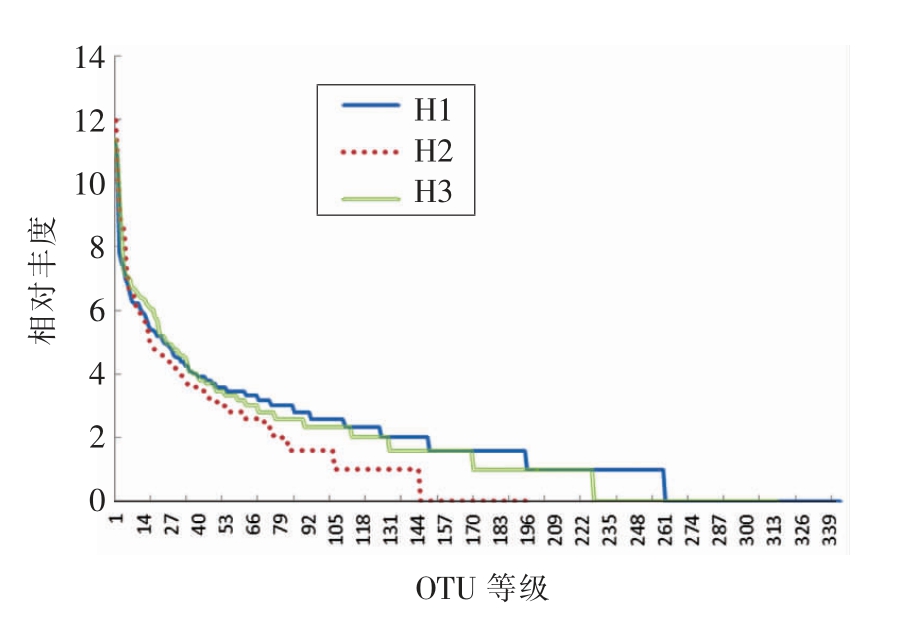
图3 样品丰度分布曲线
Fig.3 Rank abundance from stinky tofu samples
2.2.3 Alpha 多样性分析 Alpha 多样性是指一个特定地区或生态系统内的多样性,而多样性指数是反映丰富度和均匀度的综合指标。主要包括反应群落丰富度的Chao1 指数和ACE 指数,以及反应群落多样性的Shannon 指数和Simpson 指数(表1)。表1中的Chao1 指数和ACE 指数显示H1样品中群落丰富度最高,其次是H3。另一方面,Shannon 和Simpson 指数值越大,说明群落多样性越高。有表1可以看出H1 和H3 的群落多样性高于H2。
表1 Alpha 多样性
Table 1 Alpha diversity

chao1 指数 ACE 指数 simpson 指数 Shannon 指数H1 261 316.410599 0.828972 4.394096 H2 145 183.848774 0.728756 3.319686 H3 227 267.832288 0.846758 4.274834
2.2.4 PCA 分析 PCA 分析可以直观的反应数据相似性或差异性。如图4所示,成分1(PC1)和主成分2 (PC2)是造成样品间的两个最大差异特征,贡献率分别为44.45%和14.07%。这个图中的两个样品越接近,它们之间的属组成就越相似,反之亦然。这表明,H1 和H3 在组成上更相似。而这两个样品同H2 呈现明显的分离状态且距离比较远,可以推断这两个样品与H2 样品的菌落结构差异比较大。这种现象说明,原料来源不同,微生物的情况差异显著[19]。
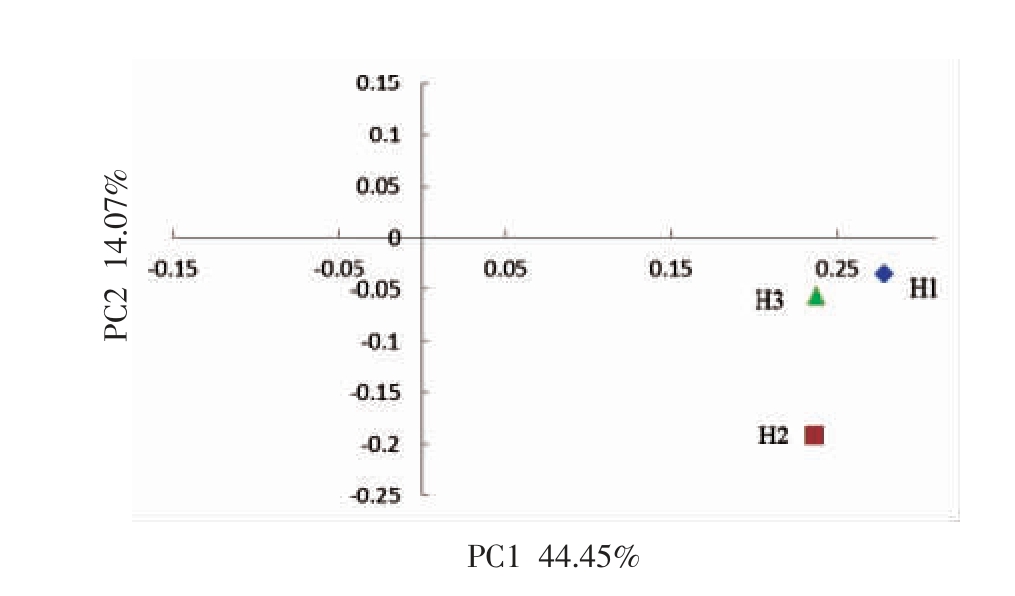
图4 主成分分析图
Fig.4 PCA analysis from stinky tofu samples
2.2.5 微生物在属和种的水平进行分类比较 通过高通量测序分析进一步说明样品的细菌分布情况,结果分别在图5A和图5B以属和种水平上呈现。如图5A 所示,样品具有相对较高的乳酸菌丰度,主要涉及8 个属,分别是乳杆菌(Lactobacillus)、乳球菌(Lactococcus)、明串珠菌(Leuconostoc)、肠球菌(Enterococcus)、链球菌(Streptococcus)、魏斯氏菌(Weissella)、库特菌(Kurthia)、漫游球菌(Vagococcus)。漫游球菌属在H2 样品中丰度值较高,赵国忠等[14]在臭豆腐卤水样品也分离到该属,但在种水平上不同。目前,采用高通量方法研究臭豆腐的菌群构成的文献相对较少。贺静等[19]通过454 平台从两个样品中分离到了24 个菌属,其中的优势菌并不是乳酸菌,而分别是厌氧球菌(Anaerococcus)、卟啉单胞菌(Porphyromonas)和氨基杆菌属(Aminobacterium)。在本试验中,卟啉单胞菌属在H1 样品中占主导地位,比例达到41.19%,其次是乳杆菌属(37.40%),而乳杆菌属在H3 中所占比例达到38.99%。而在H2 中拟杆菌门Odoribacter 菌属居于主导地位,所占比例高达一半。造成这种差异性的原因很大程度上是由于在进行自然发酵的过程中发酵环境的差异性,因此温度、pH 值以及周围环境菌群对臭豆腐发酵过程的影响,需要进一步的研究。在H1 和H3 中乳酸菌所占比例超过50%,H2 中的乳酸菌含量也达到约三分之一。因此,乳酸菌对臭豆腐菌种的构成具有重要作用[3,20-21]。其他属在某些样品中具有相对高的丰度,然而,他们在发酵过程中的作用仍然不清楚,需进一步调查。
在种水平上的分析(图5B)显示,在H1 和H3 样品中乳杆菌的相对丰度较高,其中沙克乳杆菌(Lactobacillus sakei)相对丰度最高,其次是乳酸乳球菌与婴儿链球菌 (Streptococcus infantarius)。此外,还有一些不可培养的属于乳杆菌属细菌也具有较高的相对丰度。乳酸菌在H2 中相对丰度较低,各乳酸菌种的丰度也有所差别。虽然在该样品中也检测到沙克乳杆菌,但其相对丰度并不高,河流漫游球菌(Vagococcus fluvialis)是H2样品中相对丰度值最高的乳酸菌。之前有高通量方法检测同一地区的不同臭豆腐卤水样品,菌种差异性同样也很显著[19]。造成不同样品间差异的原因,这很有可能与卤水制备中采用自然发酵,样品中的菌落构成会受到周围环境变化的影响,具有一定的偶然性有关[22]。
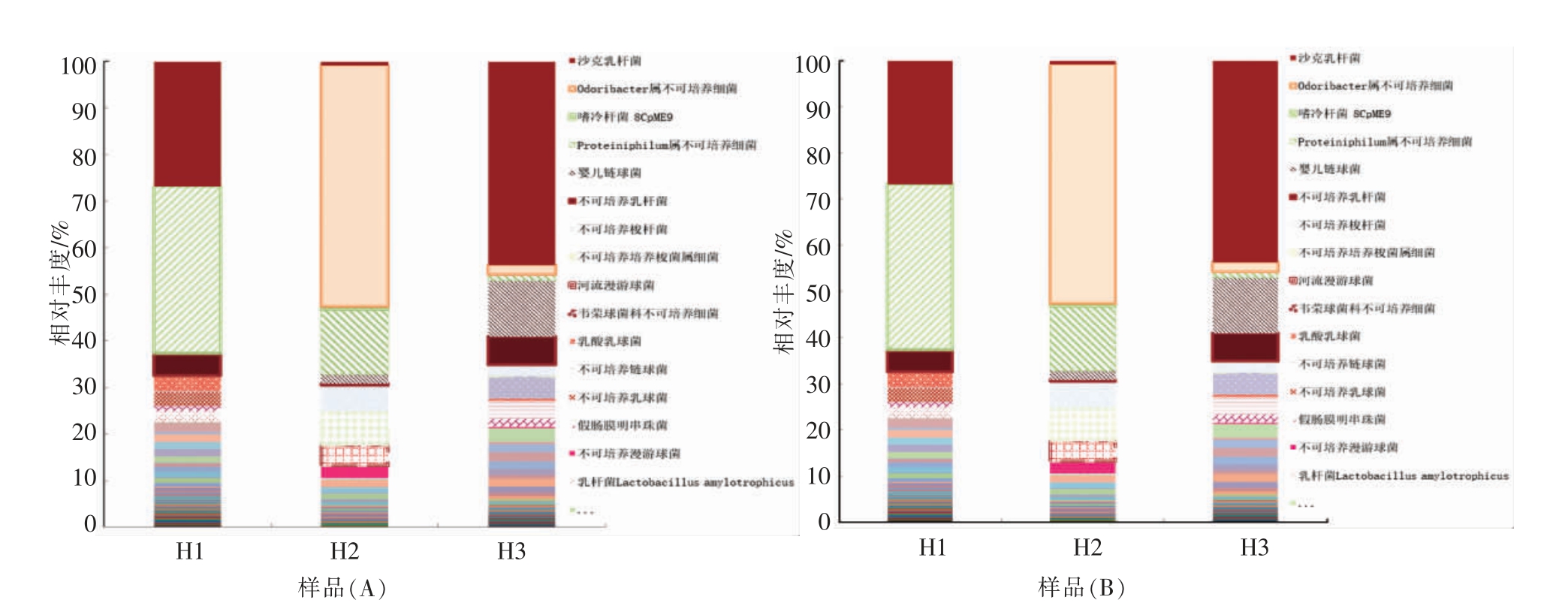
图5 基于细菌16S V3-V5 编码区高通量测序分析的相对丰度,臭豆腐样品的细菌属(A)和种(B)
Fig.5 Relative abundance,based on bacterial 16S V3-V5 tag-encoded high-throughput sequencing analysis,of bacterial genera (A)and species (B)of stinky tofu samples
通过两种鉴定方法分到离乳酸菌共71 种,其中高通量检测到的菌种多达66 种,远多于传统分离方法得到的菌种。不仅如此,通过高通量方法还发现了涉及12 个属20 种不可培养的乳酸菌。沙克乳杆菌、 河流漫游球菌等菌种在高通量测序的臭豆腐中显示出巨大的丰度,但是却没有通过传统方法检测到,这很有可能是因为培养基不适合所有乳酸菌,或者在进行培养基培养时,它们在样品中已经死亡。但在传统培养中分离出的3 种乳酸菌,分别是肠球菌(Enterococcus thailandicus)、肉色明串珠菌和乳明串珠菌,在高通量测序并没能检测到。可能由两个原因造成:首先,这3 个物种可能在臭豆腐样品中的数量非常少,因此没有足够的DNA 被提取用于高通量测序;第二,高通量测序无法区分特定的物种与其近亲和有限的DNA 长度。目前被报道的文献中,不同样品的菌群的构成存在差异性,可见敞口发酵的不稳定性使每个臭豆腐都是独特的。
本研究采用高通量测序与传统培养方法,考察了臭豆腐样品中细菌的多样性。本试验采用两种方法共检测71 种乳酸菌,臭豆腐中具有丰富的乳酸菌资源,乳酸菌很有可能在臭豆腐发酵系统中占有优势;高通量测序可以很好地反应出臭豆腐样品中细菌种类以及各种细菌所占丰度的不同,但仍需从多地区采集大量样本,扩大样本量进行验证。
在本次研究中,通过高通量测序方法鉴定出212 种菌,比传统分离培养方法检测到的菌种更丰富全面,甚至包括一些不可培养的菌种,能够更加全面地反映菌群的轮廓。高通量测序法在应用中虽受测序深度,阅读长度以及可用于测序后数据分析的算法和数据库的限制[6-7],但对于深入研究各种生态系统中微生物组成具有重要意义。
传统臭豆腐卤水制作工艺是利用微生物进行自然发酵,这使得发酵过程中存在的偶然因素对样品影响较大,从而导致了同类别的发酵食品品质指标的差异。因此有必要对臭豆腐的原料在发酵过程中微生物群变化规律、 风味物质和生理生化的变化进行深入的研究,从而能为传统产业升级提供理论依据。
[1]GU J S,LIU T J,SADIQ F A,et al.Biogenic amines content and assessment of bacterial and fungal diversity in stinky tofu - A traditional fermented soy curd[J].LWT - Food Science and Technology,2018,88(Supplement C):26-34.
[2]卢义伯,潘超,祝义亮.臭豆腐发酵菌种的筛选与鉴定[J].食品科学,2007,28(6):246-249.
[3]CHAO,S H,TOMII Y,WATANABE K,et al.Diversity of lactic acid bacteria in fermented brines used to make stinky tofu[J].International Journal of Food Microbiology,2008,123(1/2):134-141.
[4]LIU T J,LI Y,SADIQ F A,et al.Predominant yeasts in Chinese traditional sourdough and their influence on aroma formation in Chinese steamed bread[J].Food Chemistry,2018,242:404-411.
[5]曲巍,张智,马建章,等.高通量测序研究益生菌对小鼠肠道菌群的影响[J].食品科学,2017,38(1):214-219.
[6]叶雷,闫亚丽,陈庆森,等.高通量测序技术在肠道微生物宏基因组学研究中的应用[J].中国食品学报,2016,16(7):216-223.
[7]张和平,郑艺.高通量测序技术在乳制品研究中的应用[J].中国食品学报,2015,15(3):1-7.
[8]LIU T J,LI Y,CHEN Y Y,et al.Prevalence and diversity of lactic acid bacteria in Chinese traditional sourdough revealed by culture dependent and pyrosequencing approaches[J].LWT - Food Science and Technology,2016,68:91-97.
[9]陈泽斌,李冰,徐胜光,等.高通量测序分析食用玫瑰花瓣内生细菌多样性[J].中国食品学报,2017,17(9):227-235.
[10]LEE S F,HUANG W H,LIU Y C,et al.Alkaline fermented foods - stinky tofu:screening and identification of fermenting microflora for stinky brine[J].Food Science,Taiwan,1999,26(4):384-394.
[11]CHAO,S H,TOMII Y,SASAMOTO M,et al.Lactobacillus capillatus sp nov.,a motile bacterium isolated from stinky tofu brine[J].International Journal of Systematic and Evolutionary Microbiology,2008,58(11):2555-2559.
[12]CUEVA C,MORENO -ARRIBAS M V,BARTOLOME B,et al,Antibiosis of vineyard ecosystem fungi against food-borne microorganisms[J].Research In Microbiology,2011,162(10):1043-1051.
[13]FRANZETTI L,SCARPELLINI M.Characterisation of Pseudomonas spp.isolated from foods[J].Annals of Microbiology,2007,57(1):39-47.
[14]赵国忠,谢小芳,邸海波,等.臭豆腐卤水微生物群落及其接种发 酵[J].中 国酿造,2017,36(2):21-24.
[15]CAI X H,MA J,WEI D Z,et al.Functional expression of a novel alkaline-adapted lipase of Bacillus amyloliquefaciens from stinky tofu brine and development of immobilized enzyme for biodiesel production[J].Antonie Van Leeuwenhoek International Journal of General And Molecular Microbiology,2014,106(5):1049-1060.
[16]何理,吴晖.市售臭豆腐中菌种的分离及生化鉴定[J].现代食品科技,2012,28(8):952-955,958.
[17]HE S H,MA Y,ZHOU S J,et al.Antioxidant activities of fermented soybean prepared with Bacillus subtilis[J].Asian Journal of Chemistry,2013,25(18):10565-10568.
[18]伍蕊,沈宁,朱红,等.克氏库克菌血流感染1 例并文献复习[J].中国感染与化疗杂志,2015,15(4):380-382.
[19]贺静,谢靓,曾玉伦,等.454 高通量技术比较两种不同臭豆腐卤水中微生物多样性[J].微生物学杂志,2017,37(4):46-52.
[20]LIU S N,HAN Y,ZHOU Z J.Lactic acid bacteria in traditional fermented Chinese foods[J].Food Research International,2011,44(3):643-651.
[21]SUN G P,ZHANG X J,WANG Y,et al.The investigation of bacteria diversity in stinky tofu brine[J].Modern Food Science and Technology,2010,26(10):1087-1091.
[22]LIU C J,GONG F M,LI X R,et al.Natural populations of lactic acid bacteria in douchi from Yunnan Province,China[J].Journal of Zhejiang University-Science B,2012,13(4):298-306.
Diversity of Bacteria in Stinky Tofu by High-throughput Sequencing