骆驼乳是营养价值很高的饮品,因富含维生素、矿物质、微量元素,低过敏原以及耐贮藏等特性,而受到人们的喜爱。骆驼乳还富含乳铁蛋白、溶菌酶、免疫球蛋白等,具有抗微生物的作用[1]。近年来,有文献报道,与其它乳类相比,骆驼乳中的微生物含量最高,达到105 CFU/mL[2],并且其具有改善肠道内微生物平衡,促进营养物质吸收的作用[3]。XU 等[4]研究发现蒙古国骆驼乳中分离出的副干酪乳杆菌亚种具有成为益生菌的潜力,推测该菌可作为肝损伤抑制剂,用于基于代谢异常和炎症应答的肝病干预,说明骆驼乳可作为调节肠道菌群的功能性乳制品。
酒精性肝病(Alcoholic liver disease,ALD)是全世界较常见的可预防疾病之一,每年诊断为ALD 患者有上百万人,并且全球死亡人数中约6%是由ALD 引起的[5]。戒酒是ALD 患者的首要治疗措施,可防止ALD 患者发生进一步的病变[6]。有研究表明,长期大量饮酒会造成肠道屏障功能障碍[7],增加肠道通透性,使微生物产物脂多糖(Lipopolysaccharide,LPS)易位到血液和肝脏中[8],引发炎症反应,并与乙醇诱导的肝毒性产生协同作用,从而导致脂肪变性,造成ALD[9]。Ming 等[10]研究表明骆驼乳通过调节ALD 小鼠肠道菌群来减轻肝脏炎症应答,发挥其保肝作用。以上研究表明,肠道菌群与ALD 发病机制密切相关,通过靶向肠道菌群的治疗可能对ALD 有效。表征ALD共生微生物组的结构,鉴定饮酒后产生的微生物组的变化,以及探讨骆驼乳对ALD 微生物多样性及物种结构的影响具有重大意义。
本实验以骆驼乳为原料,使用NIAAA 模型诱发慢性ALD 小鼠为研究对象,研究骆驼乳对慢性酒精性肝损伤小鼠肠道菌群多样性及其结构的影响,以期为骆驼乳益生菌的开发提供理论依据。
1 材料与方法
1.1 动物与材料、试剂
SPF 级雄性C57BL/6NCr 小鼠,北京维通利华实验动物技术有限公司,许可证号:SCXK (京)2016-0006。饲养于IVC 动物实验系统内,室温20~25 ℃,相对湿度50%~60%,昼夜交替周期为12 h。
骆驼乳,采自内蒙古自治区巴彦淖尔市牧区;Lieber-DeCarli(Lieber-DeCarli,LDC)液体饲料,南通特洛菲饲料科技有限公司,生产许可证号:苏饲证(2014)06092。
小鼠脂多糖(Lipopolysaccharide,LPS)ELISA试剂盒,上海酶联生物科技有限公司;无水CH3CH2OH(GR),国药集团化学试剂有限公司;E.Z.N.A.® Soil DNA 抽提试剂盒,美国Omega Bio-Tek 公司;MiSeq 测序试剂盒,美国Illumina 公司;FastPfu Polymerase AxyPrep DNA Gel Extraction试剂盒,美国Axygen 公司;建库试剂盒,美国Bio Scientific 公司。
1.2 仪器与设备
真空冷冻干燥机(2014-18Ab),上海东Omega Bio-Tek 富龙科技股份有限公司;台式高速冷冻离心机(5810R),美国Eppendorf 公司;多功能微孔板检测仪(Synergy H1),美国BioTek 公司;Richter喂养管,南通特洛菲饲料科技有限公司;医用低温保存箱(DW-88L388J),青岛海尔特种电器有限公司;电热鼓风干燥箱(DHG-9030A)、电热恒温水槽(DK-8AXX),上海一恒科学仪器有限公司;立式压力蒸汽灭菌器(SQ510C),上海申安医疗器械厂;洁净工作台(SW-CJ-2FD),北京东联哈尔仪器制造厂;超微量分光光度计(NanoDrop2000),美国Thermo Fisher Scientific 公司;电泳仪(DYY-6C),北京市六一仪器厂;PCR 仪 (ABI GeneAmp R 9700),美国ABI 公司;测序仪(Illumina Miseq),美国Illumina 公司。
1.3 方法
1.3.1 分组与取样
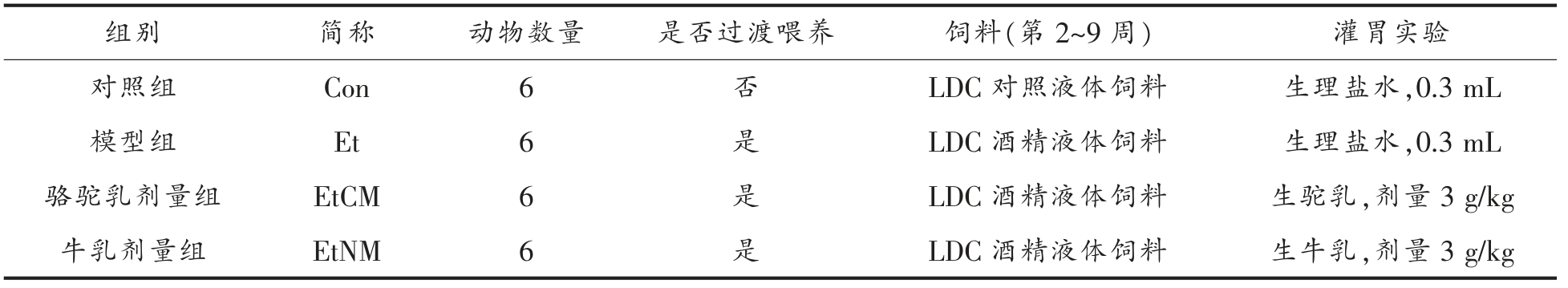
1) 动物实验 小鼠适应性饲喂1 周后,将24 只小鼠随机分为4 组,对照组(Con,n=6)、模型组(Et,n=6)、骆驼乳剂量组(EtCM,n=6)和牛乳剂量组(EtNM,n=6)。每组所饲喂饲料和实验处理如表1所示。分组后单笼饲养,专用Richter 喂养管给食,饲喂量30 mL/只·d。开始实验后,除液体饲料外,不再单独给水。实验共进行8 周,前4 周仅饲喂专用饲料,不进行灌胃操作;后4 周饲喂方式不变,灌胃给乳或生理盐水,每日灌胃1 次,灌胃体积0.3 mL。
表1 每组所饲喂饲料和实验处理
Table 1 Feed and experimental treatments for each group
?
2) 造模 参考Bertola 等[11]的研究方法,建立NIAAA 模型(图1)。第1 周采用过渡饲喂法[12],第2~9 周开始,造模组连续接受8 周的4%LDC 酒精液体饲料饲喂,对照组喂等热量LDC 对照液体饲料。造模组在结束液体饲料饲喂后的第2 天,按照5 g/kg 剂量一次性灌胃体积分数31.5%的酒精溶液,对照组灌胃等热量的麦芽糖糊精溶液。

图1 NIAAA 造模图
Fig.1 NIAAA modeling diagram
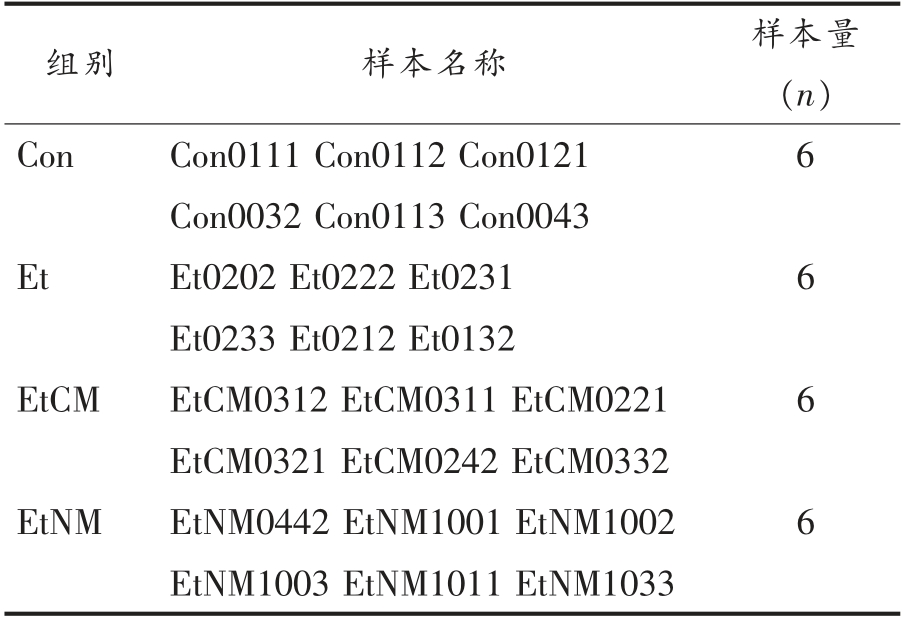
造模结束后,异氟烷麻醉小鼠,眼眶采血,颈椎脱臼法处死小鼠,并在无菌操作台上收集小鼠结肠部粪便,保存于灭菌后的EP 管中,液氮速冻后保存于-80 ℃冰箱。样本分组情况如表2所示。
表2 小鼠结肠粪便样本信息
Table 2 Mice colon stool sample information
?
1.3.2 血清指标检测 血液常温下静置4 h 后,低温离心(4 ℃,3 000 r/min,25 min)分离血清,按照ELISA 试剂盒说明书的方法检测血清中LPS的含量(EU/L)。
1.3.3 小鼠肠道菌群分析
1.3.3.1 小鼠微生物群落多样性测序实验过程将小鼠粪便从-80 ℃冰箱取出后,快速放入干冰内送至上海美吉生物医药科技有限公司测序。实验过程如下:
1) E.Z.N.A.® Soil DNA 抽提试剂盒随机提取部分小鼠粪便中的DNA,以338F(5′-ACTCC TACGGGAGGCAGCAG-3′)和806R(5′-GCAC TACHVGGGTWTCTAAT-3′)为引物,进行PCR 预实验,扩增16S rRNA 基因的V3-V4 区域。并用NanoDrop2000 超微量分光光度计和2%琼脂糖凝胶电泳检测PCR 产物的质量和浓度以及完整性。扩增出浓度合适的产物后,进行正式实验。
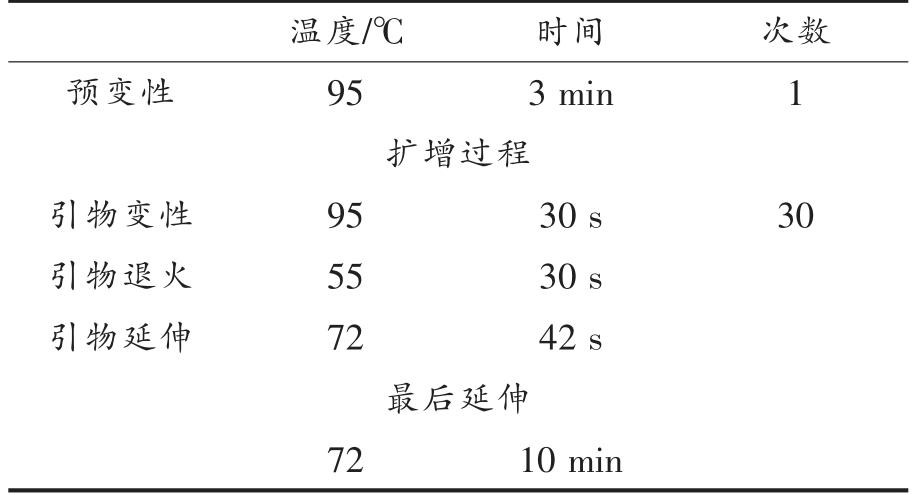
2) PCR 正式实验采用TransGen AP221-02:TransStart FastPfu DNA Polymerase,20 μL 反应体系:4 μL 的5×FastPfu Buffer,2 μL 2.5 mmol/L dNTPs,上、下引物各0.8 μL,0.4 μL FastPfu Polymerase,0.2 μL BSA,10 μL Template DNA,最后加ddH2O 至20 μL。PCR 反应参数如表3所示。
表3 PCR 反应参数
Table 3 PCR reaction parameters
温度/℃ 时间 次数预变性 95 3 min 1扩增过程引物变性 95 30 s 30引物退火 55 30 s引物延伸 72 42 s最后延伸72 10 min
3) 按照1.3.3.1(1)节的方法检测PCR 产物的质量和浓度及完整性,并用FastPfu Polymerase AxyPrep DNA Gel Extraction 试剂盒回收PCR 产物。用酶标仪检测 PCR 产物定量。用NEXTFLEX Rapid DNA-Seq 试剂盒构建MiSeq PE 文库。最后用Illumina Miseq PE 300 平台进行高通量测序。
1.3.3.2 生物学信息分析 MiSeq 测序得到的是双端序列数据,并优化数据。首先通过PE reads和overlap 的关系,将成对reads 拼接(merge)成一条序列,同时对reads 的质量和merge 的效果进行质控过滤;其次,根据序列首尾两端的barcode 和引物序列区分样品,最后得到优化序列。基于优化序列进行OTU 聚类之后与Silva 16S rRNA 数据库(v132)进行序列对比,并采用RDP classifier 贝叶斯算法对97%相似水平的OTU 代表序列进行物种鉴定和注释。根据分类学统计结果,实验一方面评估本次测序样本量是否足够,另一方面通过Alpha 多样性统计各OTU 注释结果在每个样本中对应的丰度信息;之后进行Beta 多样性分析,得到不同样本间菌群结构差异及各类水平上的物种结构信息。
1.4 数据处理
Alpha 多样性指数数据使用Wilcoxon 秩和检验进行分析之后,用Graphpad prism 7 软件进行绘图。与正常组比较,*.P<0.05,**.P<001;与模型组比较,#.P<0.05,##.P<0.01。
2 结果与分析
2.1 骆驼乳对慢性酒精性肝损伤小鼠血清LPS含量的影响
由图2所示,与Con 组相比较,Et 组小鼠血清中LPS 含量显著升高(P<0.01),说明酒精导致小鼠肠道粘膜损伤,使肠道菌群发生定量和定性改变(即小肠细菌过度生长和营养不良),肠道通透性增加,从而导致细菌LPS 易位至门脉血流[13]。与Et 组相比较,EtCM 和EtNM 组小鼠血清中LPS 的含量显著降低(P<0.01),其中EtCM 组小鼠血清中LPS 含量小于Con 组小鼠。实验结果表明骆驼乳可以预防酒精引起的结肠功能障碍,能够抑制血清中LPS 的含量的升高。
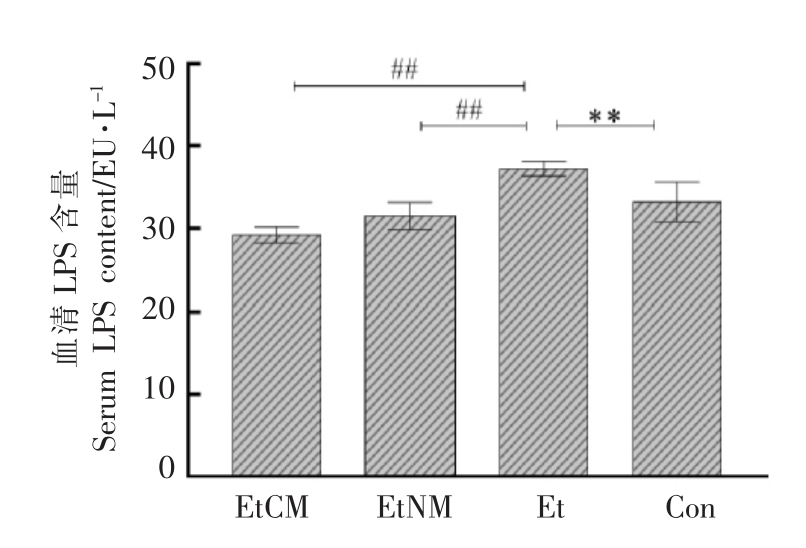
图2 骆驼乳对慢性酒精性肝损伤引起的小鼠血清中LPS 含量的影响
Fig.2 Effect of camel milk on changes in LPS content in serum of mice caused with chronic alcoholic liver injury
2.2 α 多样性分析
长期大量饮酒会降低细菌多样性。通过α 多样性来检查OTU 数量[14],α 多样性是度量单个样本中微生物群落的丰度和均匀度的指标[15]。本实验 采 用ACE 指 数、Chao 指 数、Shannon 指 数、Simpson 指数、Coverage 指数和稀释曲线来评估单个样本的α 多样性。
采用对有效序列进行随机抽样的方法绘制稀释曲线,以抽到的序列数与它们对应的OTU 数目构建稀释曲线,并用mothur 计算不同随机抽样下的α 多样性指数,利用R 语言工具制作曲线图[16]。如图3结果所示,在OTU 水平上,小鼠肠道菌群的多样性指数Sobs 稀释曲线趋于平坦(图3a),说明本次测序数据量合理,增加数据量只会产生少量新的OTU。并且小鼠肠道菌群的多样性指数Shannon 稀释曲线也趋于平坦(图3b),说明测序数据量足够大,可以反映样本中绝大多数的微生物多样性信息。
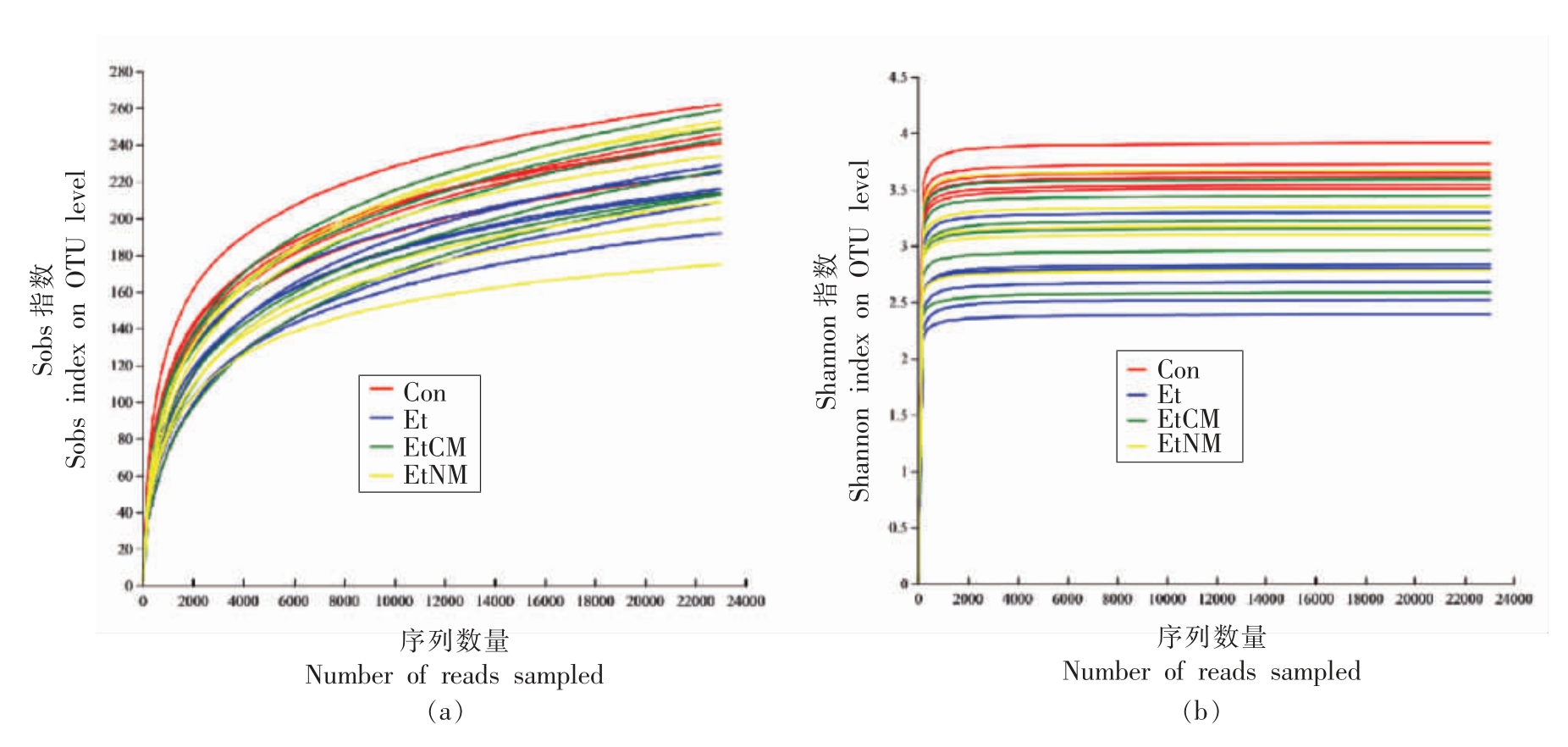
图3 基于Sobs 指数与Shannon 指数的稀释曲线图
Fig.3 Dilution curve based on Shannon index and Sobs index
ACE 和Chao 指数反映样本中所含微生物群落丰度。而Shannon 和Simpson 指数反映样本中所含微生物群落丰度和均匀度两方面评估[15]。Coverage 指数反映样本中所含微生物群落的覆盖度[17]。由图4结果可知,每个样本的Coverage 值均为0.997 以上,表明测序深度良好,覆盖率高,可以真实反映样本中微生物群落状况(图4a)。根据ACE(图4b)和Chao(图4c)指数结果可知,Et 组小鼠粪便微生物丰度显著降低,而EtCM 组小鼠粪便微生物丰度显著升高。并且根据Shannon(图4d)和Simpson(图4e)指数结果显示,Et 组小鼠粪便微生物群落均匀度显著降低,而EtCM 组小鼠粪便微生物群落均匀度呈上上升趋势。以上结果表明,虽然EtCM 组小鼠粪便微生物群落均匀度略低,但是有更高的丰度,表现出比其它组较高的多样性。
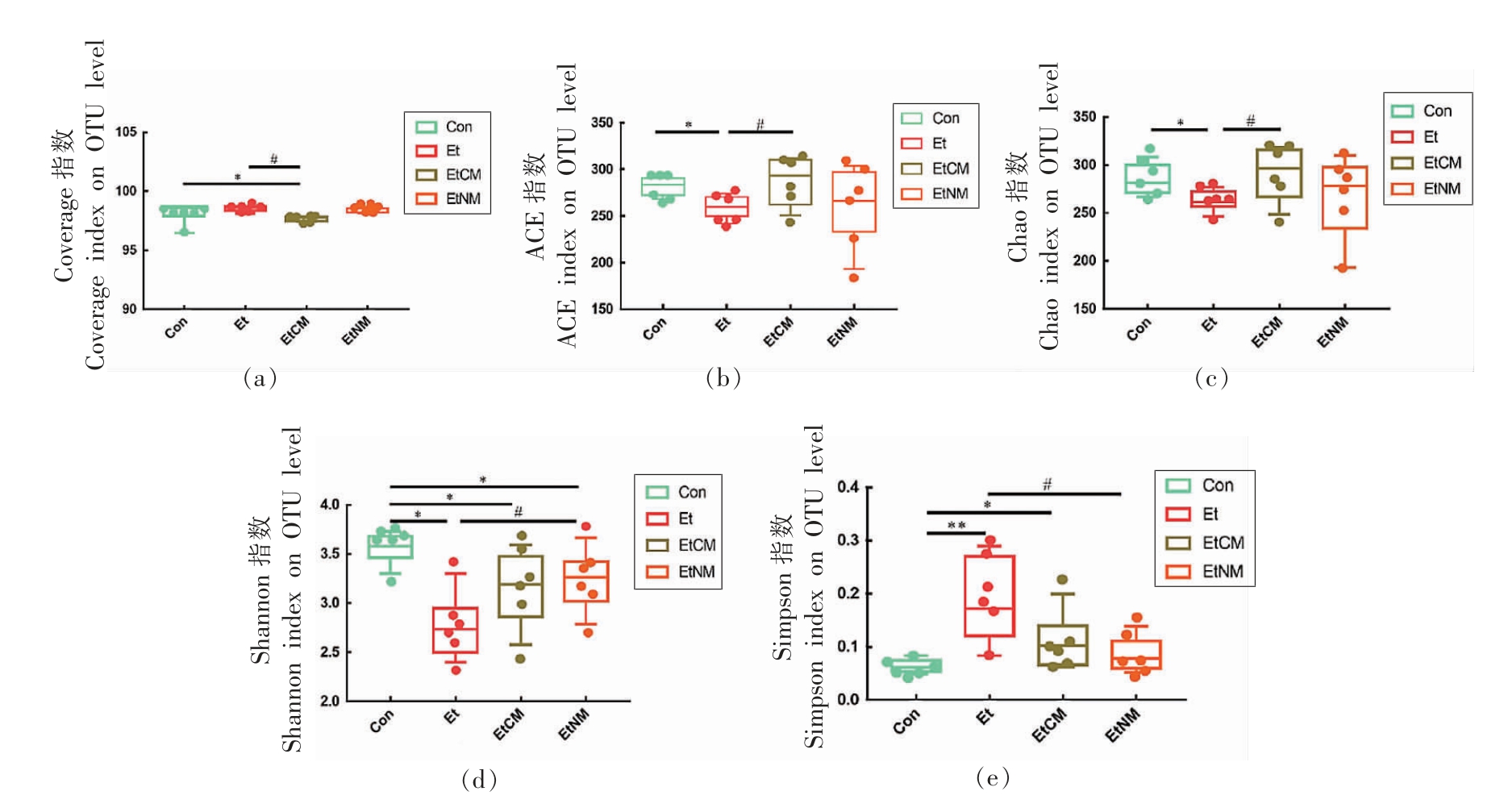
图4 基于OTU 总数的α 多样性指数图
Fig.4 α-Diversity indexes based on the total number of OTUs
2.3 各组在OTU 水平上的Venn 图分析
Venn 图可用于统计多组或多个样本中所共有和独有的物种(如OTU)数目,可以直观展现不同组样本中物种(如OTU)结构相似性及重叠情况[18]。由图5可知,4 组样本中共检验出1 413 个OTU。EtCM 组样本OTU 数量最多,达到370 个,其次是Con 组,OUT 数为357 个;EtNM 组和Et 组OUT 数分别为345 个和341 个。长期大量饮酒,导致小鼠OTU 数量有所降低,这可能由于饮酒使小鼠肠道菌群失调,影响肠道菌群结构[14]。经骆驼乳干预处理后,小鼠OTU 数量显著增加,并且较Con组小鼠OTU 数量高出13 个。表明骆驼乳可以显著提高小鼠粪便微生物群落的多样性。该结果与α 多样性结果相一致。从维恩图(图5)进一步得出,4 组共有OTU 数为258 个,Con、Et、EtCM 和EtNM 组独有的OTU 数分别为99,83,112,87 个,分别占各组总OTU 数的27.73%,24.34%,30.27%和25.22%。
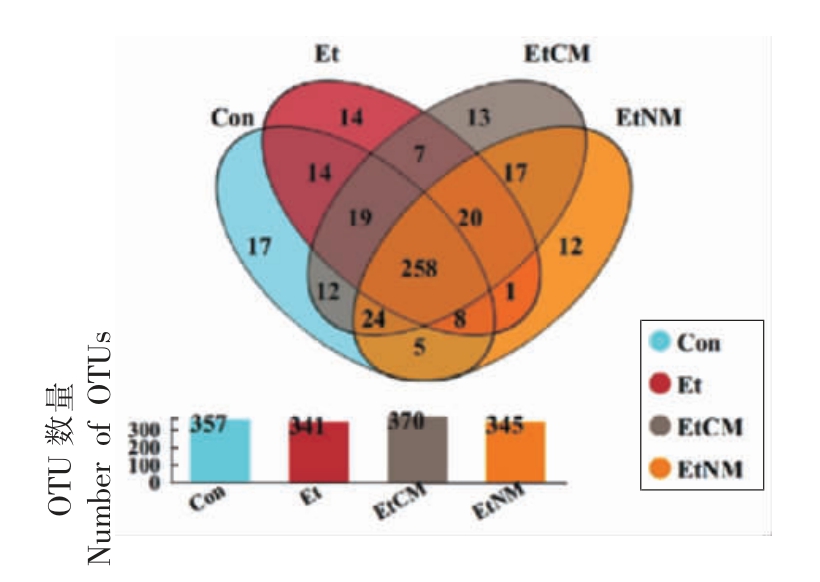
图5 各组微生物群落OTU 水平的Venn 图
Fig.5 Venn diagram of OTU level of microbial communities in each group
2.4 β 多样性分析
β 多样性是度量样本间微生物群落结构相似度的指标。PCoA 分析法用来考察和区分样本间的菌群结构差异,是β 多样性分析方法之一。通过β多样性分析可以确定不同组间微生物群落是否存在差异[19]。由图6所示,每组样本点分散区域比较密集,表明组内差异较小,微生物群落结构相似。根据P<0.001,表明组间差异很大,Con、Et、EtCM和EtNM 组能很好地区分开来(图6a),说明酒精和骆驼乳以及牛乳对小鼠的微生物群落有显著影响。而EtCM 组和EtNM 组的组间差异很小,两组小鼠肠道菌群结构上无显著差异 (P>0.05)(图6b)。
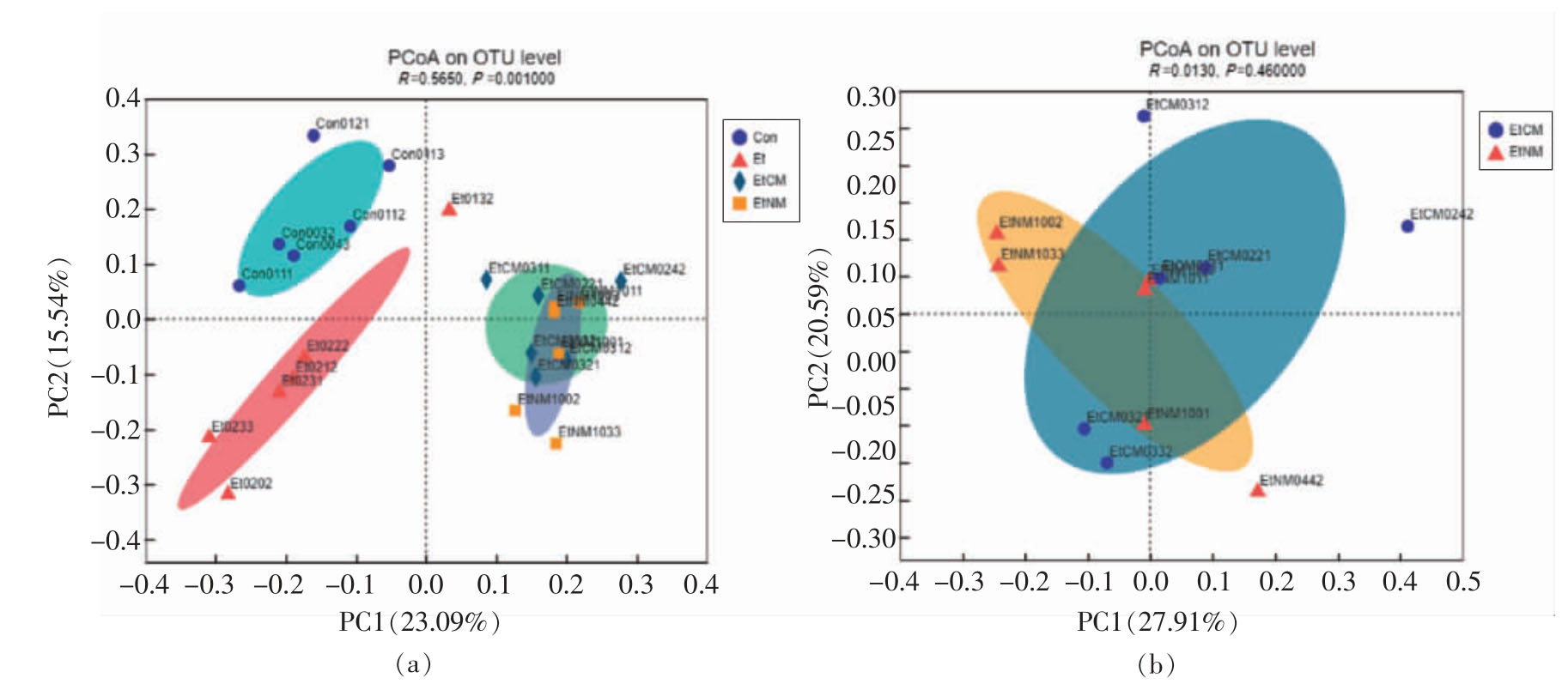
图6 小鼠肠道菌群与PCoA 分析图
Fig.6 Analysis of mouse intestinal community and PCoA
根据α 多样性和β 多样性分析,可以确定本次实验测序数据量足够大,可以反映样本中绝大多数微生物多样性信息;其中EtCM 组小鼠粪便微生物群落α 多样性最高,而Et 组小鼠粪便微生物群落α 多样性最低。
通过PCoA 分析,发现Con 与Et 组小鼠肠道菌群结构上有显著差异,表明酒精使小鼠肠道菌群结构发生了明显变化。经骆驼乳和牛乳干预处理后,发现Et 与EtCM 组和EtNM 组小鼠肠道菌群结构上有显著差异,说明骆驼乳和牛乳能在一定程度上改变小鼠肠道菌群环境,调节小鼠肠道菌群结构。此外,研究发现EtCM 组和EtNM 组小鼠肠道菌群结构上无显著差异。Morrin 等[20]研究发现牛乳可以直接调节肠道细胞表面,并可能改变细胞的糖基化状态,进而促进不同细菌群落,如双歧杆菌(Bifidobacterium)的黏附。Morrin 等[21]研究发现牛乳中的免疫球蛋白G 刺激肠道上皮细胞表面来增加双歧杆菌对肠上皮细胞的粘附。Zhao等[22]研究发现新鲜的骆驼乳具有较高的细菌多样性,并且是分离新型乳酸菌的宝贵天然资源。Ming等[10]研究表明骆驼乳通过调节ALD 小鼠肠道菌群,来减轻肝脏炎症应答,发挥它的保肝作用。因此骆驼乳与牛乳一样,通过改变肠道菌群的生存环境,调节肠道菌群的丰度,来调整菌群结构,促进有益菌在肠道内黏附,减少有害菌的增殖,并且防止乙醇对小鼠结肠内微生态平衡的破坏。
2.5 物种结构分析
2.5.1 基于门水平上的物种结构分析 物种结构分析采用统计学的分析方法,可得到各样本在各水平(域、界、门、纲、目、科、属、种、OTU 等)上含有哪些优势物种,以及各优势物种的相对丰度[23]。本实验重点分析了门水平和属水平。
探究骆驼乳和牛乳对慢性ALD 小鼠肠道菌群结构的影响。由图7可知,在门水平上,4 组细菌种类的相对丰度,检测到厚壁菌门(Firmicutes)、拟杆菌门(Bacteroidetes)、疣微菌门(Verrucomicrobia)、变形菌门(Proteobacteria)、放线菌门(Actinobacteria)、脱铁杆菌门(Deferribacteres)等为小鼠结肠肠道中相对丰度较高的菌门,其中厚壁菌门和拟杆菌门的丰度最高,两者的相对丰度之和超过了80%。与Con 组相比,Et 组厚壁菌门丰度提高了4.97%,而拟杆菌门的丰度降低了4.30%。与Et 组相比,EtCM 组厚壁菌门丰度降低了30.96%,拟杆菌门丰度提高了27.9%。EtNM 组厚壁菌门丰度降低了20.71%,拟杆菌门丰度提高了18.70%。其中,EtCM 组有效提高了小鼠肠道菌群的丰度。
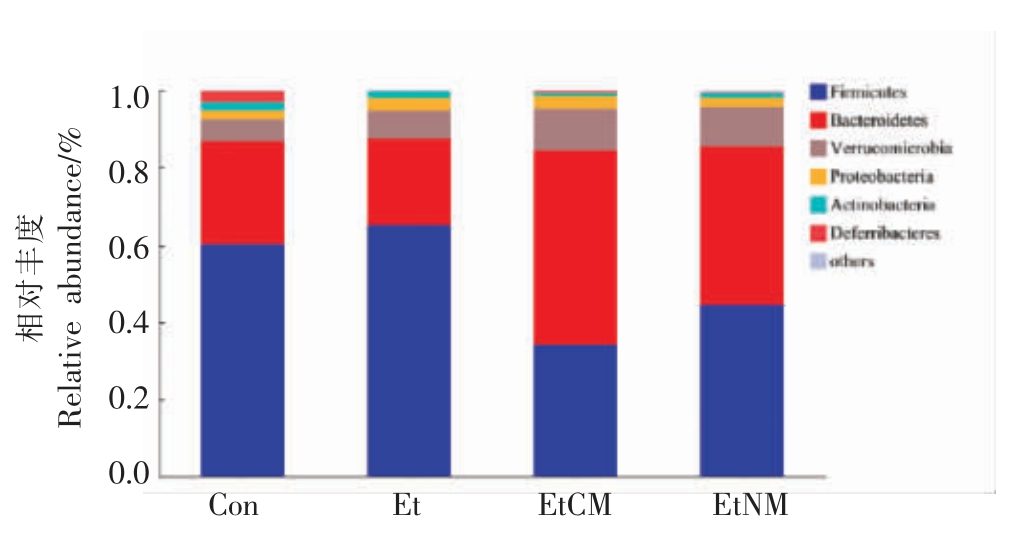
图7 小鼠肠道菌群门水平物种结构分析柱状图
Fig.7 Histogram of phylum level species composition analysis of intestinal flora in mice
2.5.2 基于属水平上的物种结构分析 由图8可知,在属水平上,瘤胃菌科(Ruminococcaceae)下的未知属Ruminococcaceae_UCG-013、副拟杆菌属(Parabacteroides)、拟杆菌属(Bacteroides)、阿克曼菌属 (Akkermansia)、Muribaculaceae 科下的未知属norank_f_Muribaculaceae 属、柔嫩梭菌属(Faecalibaculum)、Romboutsia 属、毛螺旋菌科(Lachnospiraceae)下的未知属norank_f_Lachnospiraceae等在小鼠结肠肠道中相对丰度较高的菌属。其中瘤胃菌科下的未知属Ruminococcaceae_UCG-013、副拟杆菌属、拟杆菌属、阿克曼菌属的丰度最高。此外,由图9可知,瘤胃菌科下的未知属Ruminococcaceae_UCG-013 是Et 组丰度最高的优势菌属,相对丰度占41%。副拟杆菌属、拟杆菌属和阿克曼菌属均为EtCM 组和EtNM 组的优势菌属。3 个菌属的丰度各占10%以上,其中EtCM 组的3个菌属丰度之和最高约为45%。而EtNM 组的3个菌属丰度之和最高约为36%,这与门水平结果相一致。Blacher 等[23]研究发现,瘤胃菌会加重肌萎缩性侧索硬化症。Ponziani 等[24]还研究发现肝癌患者肠道菌群中瘤胃菌属丰度增加,双歧杆菌丰度减少。本实验中Et 组小鼠结肠菌群的瘤胃菌科下的未知属Ruminococcaceae_UCG-013 丰度显著提高,表明瘤胃菌科下的未知属Ruminococcaceae_UCG-013 可能是加重慢性ALD 的一类菌属。Wang 等[25]研究发现白藜芦醇处理的小鼠微生物菌群结构发生了显着变化,拟杆菌、毛螺菌科下的未知属Lachnospiraceae_NK4A136_group、Lachnoclostridium、布劳特氏菌(Blautia)、副拟杆菌属和Ruminiclostridium_9 丰度显著提高,并且这些菌群能调节脂肪代谢,还通过减少炎症来改善肠屏障功能。Henning 等[26]也得到了同等结果。最新研究发现,罗斯伯里氏菌属(Roseburia)、阿克曼菌属、丙酸杆菌属(Propionibacterium)等可以参与到未来益生菌的开发中,称为潜在益生菌[27]。总的来说,骆驼乳和牛乳均拥有改善肠道屏障功能的优势菌群。而骆驼乳的优势菌群丰度比牛乳较高。骆驼乳的作用机制很可能是通过提高副拟杆菌属、拟杆菌属、阿克曼菌属的丰度,来降低瘤胃菌科下的未知属Ruminococcaceae_UCG-013 的丰度,达到调节小鼠结肠肠道菌群的目的。
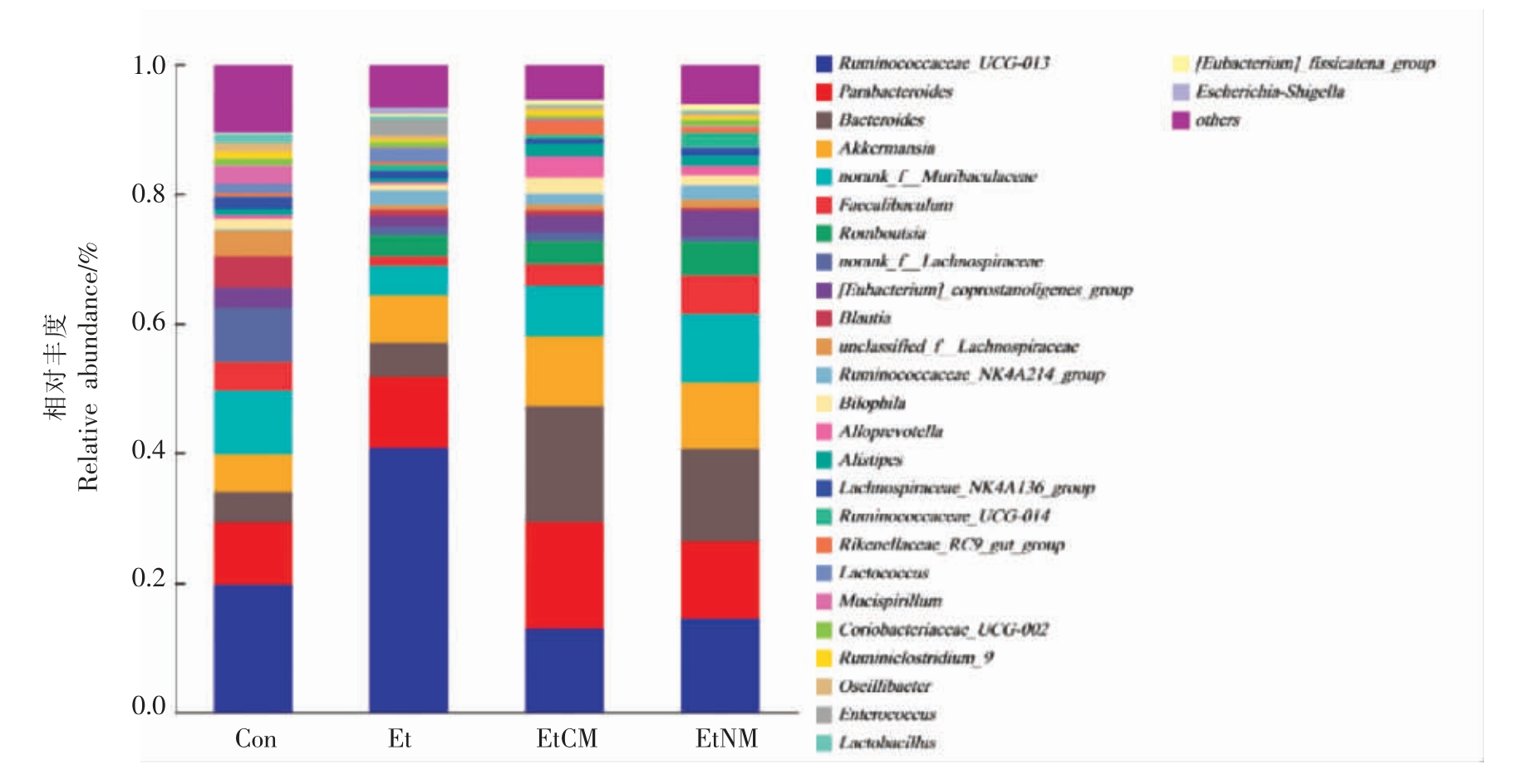
图8 小鼠肠道菌群属水平物种结构分析柱状图
Fig.8 Histogram of the analysis of the genus level species composition of the mouse intestinal flora
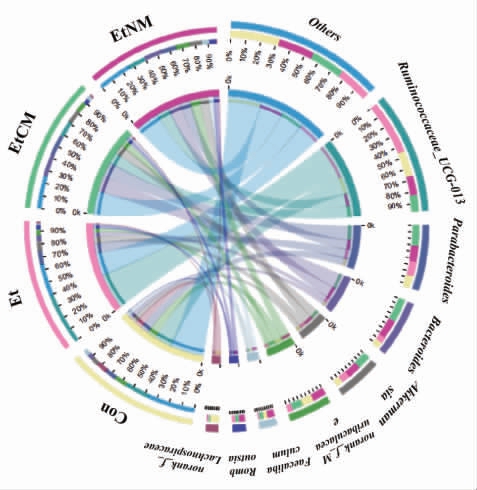
图9 样本与物种关系图
Fig.9 Relationship between samples and species
3 结论
肠道菌群结构主要受酒精的影响。已有文献报道,长期大量饮酒会增加血液酒精浓度[28]。并且降低Caco-2 肠上皮细胞中的紧密连接蛋白occludin 和zona-occludens-1(ZO-1)的表达,使肠道通透性增加,让肠道菌群发生定量和定性改变,从而导致细菌LPS 易位到血流中并产生炎症,促成ALD[29]。本实验研究发现,在慢性ALD 模型小鼠血清中细菌LPS 含量显著升高,这与Mutlu 等[30]的临床结果相一致。此外,研究还发现经骆驼乳和牛乳干预处理后小鼠血清中LPS 含量显著降低,其中骆驼乳效果最佳,表明骆驼乳可以预防酒精引起的结肠功能障碍,抑制血清中LPS 含量的升高。Chu 等[31]研究发现用益生元UG1601 治疗便秘后,肠道菌群结构发生改变,包括厚壁菌门的减少和丁酸盐细菌的增加,并推测这可能是减轻内毒素血症的主要原因。已有文献报道,丙酸和丁酸盐均被认为是有益菌的代谢产物[27];而拟杆菌门是产生丁酸盐的主要菌群[32]。以上研究进一步证明,Et 组的优势菌群厚壁菌门通过产生大量的LPS,促成ALD。而经骆驼乳和牛乳干预处理后,拟杆菌门成为优势菌群,降低了厚壁菌门的丰度,从而改善了肠道微生态平衡。
在本实验中,通过高通量测序,观察到骆驼乳和牛乳对ALD 小鼠肠道菌群有调节作用。其改善ALD 的主要机制是通过改变肠道菌群环境,来调节肠道菌群。其主要变化包括促进肠道优势益生菌的繁殖,来抑制肠道有害菌的生长。从有益菌的角度出发,在门水平上,骆驼乳和牛乳能提高拟杆菌门的丰度。在属水平上,提高副拟杆菌属、拟杆菌属、阿克曼菌属的丰度。从有害菌的角度出发,在门水平上,降低了厚壁菌门的丰度。在属水平上,降低了瘤胃菌科下的未知属Ruminococcaceae_UCG-013 的丰度,其中骆驼乳的有益菌丰度较牛乳高出9%,并且Et 组小鼠肠道菌群与EtCM 和EtNM 组的小鼠肠道菌群结构上有显著差异。除此之外,研究还发现EtCM 和EtNM 与Con 组小鼠肠道菌群有高度的相似性。由此可知,饮用骆驼乳来预防慢性ALD 有利于长期维持肠道健康。特别是对患有乳糖不耐受症的ALD 患者,饮用骆驼乳调节肠道菌群是最佳选择。本实验结果可为骆驼乳缓解慢性ALD 提供理论基础,并对骆驼乳益生菌产品的开发具有重要意义。
[1]DUBEY U S,LAI M,MITTEL A,et al.Therapeutic potential of camel milk[J].Emirates Journal of Food and Agriculture,2016,28(3):164-176.
[2]DONG F,HENNESSY D A,JENSEN H H.Factors determining milk quality and implications for production structure under somatic cell count standard modification[J].Journal of Dairy Science,2020,95(11):6421-6435.
[3]IZADI A,KHEDMAT L,MOJTAHEDI S Y.Nutritional and therapeutic perspectives of camel milk and its protein hydrolysates:A review on versatile biofunctional properties [J].Journal of Functional Foods,2019,60:1-13.
[4]XU R H,XIU L,ZHANG Y L,et al.Probiotic and hepatoprotective activity of lactobacillus isolated from Mongolian camel milk products[J].Beneficial Microbes,2019,10(6):699-710.
[5]SINGAL A K,BATALLER R,AHN J,et al.ACG clinical guideline:Alcoholic liver disease[J].American Journal of Gastroenterology,2018,113(2):175-194.
[6]SEHRAWAT T S,LIU M,SHAH V H.The knowns and unknowns of treatment for alcoholic hepatitis[J].Lancet Gastroenterology and Hepatology,2020,5(5):494-506.
[7]CHAN P,STARKEL P,TURNER J R,et al.Dysbiosis-induced intestinal inflammation activates TNFRI and mediates alcoholic liver disease in mice[J].Hepatology,2015,61(3):883-894.
[8]YANG A M,INAMINE T,HOCHRATH K,et al.Intestinal fungi contribute to development of alcoholic liver disease[J].Journal of Clinical Investigation,2017,127(7):2829-2841.
[9]BAJAJ J S.Alcohol,liver disease and the gut microbiota[J].Nature Reviews Gastroenterology and Hepatology,2019,16(4):235-246.
[10]MING L,QIAO X Y,YI LI,et al.Camel milk modulates ethanol-induced changes in the gut microbiome and transcriptome in a mouse model of acute alcoholic liver disease[J].Journal of Dairy Science,2020,103(5):3937-3949.
[11]BERTOLA A,MATHEWS S,KI S H,et al.Mouse model of chronic and binge ethanol feeding(the NIAAA model)[J].Nature Protocols,2013,8(3):627-637.
[12]LIN H,CHEN D,DU Q,et al.Dietary copper plays an important role in maintaining intestinal barrier integrity during alcohol-induced liver disease through regulation of the intestinal HIF-1 alpha signaling pathway and oxidative stress[J].Frontiers in Physiology,2020,11:1-10.
[13]VASSALLO G,MIRIJELLO A,FERRULLI A,et al.Review article:Alcohol and gut microbiota the possible role of gut microbiota modulation in the treatment of alcoholic liver disease[J].Alimentary Pharmacology & Therapeutics,2015,41(10):917-927.
[14]ZHANG W,ZHONG W,SUN Q,et al.Hepatic overproduction of 13-HODE due to ALOX15 upregulation contributes to alcohol-induced liver injury in mice[J].Scientific Reports,2017,7(1):1-10.
[15]YANG Y,MISRA B B,LIANG L,et al.Integrated microbiome and metabolome analysis reveals a novel interplay between commensal bacteria and metabolites in colorectal cancer[J].Theranostics,2019,9(14):4101-4114.
[16]BULL O L,FENG W K I,WANG Y,et al.Metagenomic analyses of alcohol induced pathogenic alterations in the intestinal microbiome and the effect of Lactobacillus rhamnosus GG treatment[J].Plos One,2013,8(1):1-10.
[17]QIAN X B,CHEN T,XU Y P,et al.A guide to human microbiome research:Study design,sample collection,and bioinformatics analysis [J].Chinese Medical Journal,2020,133(15):1844-1855.
[18]WANG Y,XU L,GU Y Q,et al.MetaCoMET:A web platform for discovery and visualization of the core microbiome[J].Bioinformatics,2016,32(22):3469-3470.
[19]FENG J,ZHAO F,SUN J,et al.Alterations in the gut microbiota and metabolite profiles of thyroid carcinoma patients[J].International Journal of Cancer,2019,144(11):2728-2745.
[20]MORRIN S T,LANE J A,MAROTTA M,et al.Bovine colostrum-driven modulation of intestinal epithelial cells for increased commensal colonization[J].Applied Microbiology and Biotechnology,2019,103(6):2745-2758.
[21]MORRIN S T,MCCARTHY G,KENNEDY D,et al.Immunoglobulin G from bovine milk primes intestinal epithelial cells for increased colonization of bifidobacteria[J].AMB Express,2020,10(1):1-10.
[22]ZHAO J,FAN H,KWOK L,et al.Analyses of physicochemical properties,bacterial microbiota,and lactic acid bacteria of fresh camel milk collected in Inner Mongolia[J].Journal of Dairy Science,2020,103(1):106-116.
[23]BLACHER E,BASHIARDES S,SHAPIRO H,et al.Potential roles of gut microbiome and metabolites in modulating ALS in mice[J].Nature,2019,572(7770):474-480.
[24]PONZIANI F R,BHOORI S,CASTELLI C,et al.Hepatocellular carcinoma is associated with gut microbiota profile and inflammation in nonalcoholic fatty liver disease[J].Hepatology,2019,69(1):107-120.
[25]WANG P,LI D,KE W,et al.Resveratrol-induced gut microbiota reduces obesity in high-fat diet-fed mice[J].International Journal of Obesity,2020,44(1):213-225.
[26]HENNING S M,YANG J,HSU M,et al.Decaffeinated green and black tea polyphenols decrease weight gain and alter microbiome populations and function in diet-induced obese mice[J].European Journal of Nutrition,2018,57(8):2759-2769.
[27]SANDERS M E,MERENSTEIN D J,REID G,et al.Probiotics and prebiotics in intestinal health and disease:From biology to the clinic[J].Nature Reviews Gastroenterology and Hepatology,2019,16(10):605-616.
[28]YI D,CRISTINA L,SONJA L,et al.Bacteriophage targeting of gut bacterium attenuates alcoholic liver disease[J].Nature,2019,575:505-509.
[29]SZABO G.Gut-liver axis in alcoholic liver disease[J].Gastroenterology,2015,148(1):30-36.
[30]MUTLU E,KESHAVARZIAN A,ENGEN P,et al.Intestinal dysbiosis:A possible mechanism of alcohol-induced endotoxemia and alcoholic steatohepatitis in rats[J].Alcoholism Clinical and Experimental Research,2009,33(10):1836-1846.
[31]CHU J R,KANG S Y,KIM S E,et al.Prebiotic UG1601 mitigates constipation-related events in association with gut microbiota:A randomized placebo-controlled intervention study[J].World Journal of Gastroenterology,2019,25(40):6129-6144.
[32]YANG C,DENG Q,XU J,et al.Sinapic acid and resveratrol alleviate oxidative stress with modulation of gut microbiota in high-fat diet-fed rats[J].Food Research International,2019,116:1202-1211.