益生元是膳食补充剂之一,通常不能直接被机体消化吸收,然而,可以被肠道有益菌发酵利用刺激其生长繁殖或是激活代谢功能活性,从而改善宿主健康[1-3]。益生元主要包括功能性低聚糖类、多糖类、天然植物提取物以及多元醇等,目前最常见的低聚糖类益生元主要包括低聚果糖、低聚半乳糖、低聚异麦芽糖和低聚木糖[4-5]。大多数益生元为碳水化合物且结构多样、组成复杂,在人和动物的饮食中普遍存在[6]。益生元从动、植物中提取得到,由于存在多个单糖成分,因此更容易被益生菌吸收利用[7]。有大量研究发现益生元可以改善体内菌群丰度,增强益生菌的肠道定植能力,减少有害微生物的代谢,增强机体免疫,调节便秘和腹泻等[8-11]。合格的益生元应具备在人体内不被上消化道水解吸收,并且在胃酸的环境下保持稳定,最终到达结肠,达到促进肠道有益菌群的生长与繁殖,改善宿主健康的效果。王艺苑等[12]将不同剂量的低聚半乳糖添至初断乳SD 大鼠饲料中,发现低聚半乳糖可有效促进大鼠体内乳酸杆菌和双歧杆菌的繁殖,在大鼠粪便中的相对丰度也明显增加。由于菌株之间具有差异性,各菌株的糖代谢途径也有所不同,从而导致不同益生菌对益生元的吸收利用也有所不同[13],亟需探究益生元对菌株的富集作用。
粪便是通过胃肠道代谢出来的生理产物,可以间接反映肠道环境以及肠道微生物的情况[14]。本研究以小鼠粪便为对象,探究小鼠粪便微生物生长所需限制性营养因子,并通过分析粪便微生物组成结构和多样性的变化,得出不同益生元对粪便微生物生长的影响。
1 材料与方法
1.1 实验动物与试剂
健康SPF 级雄性C57 BL/6J 小鼠,体质量(20± 2)g,购自上海杰思捷实验动物有限公司,许可证号码:SCXK(沪)2018-0006。饲养温度控制在(22 ± 2)℃,相对湿度控制在(50 ± 10)%,12 h 光照和12 h 黑暗交替循环,在整个饲养期间小鼠自由饮食饮水。所有涉及动物的程序均严格按照中国有关实验动物的相关饲养规范和法律规定。
低聚果糖,国药集团化学试剂有限公司;胰蛋白胨,上海润捷化学试剂有限公司;rTaq Mix,上海生物工程股份有限公司;DNA 提取试剂盒(E.Z.N.A.Stool DNA Kit),美国Omega Bio-Tek 公司;琼脂糖(RA1011-Agarose LE-100G),上海捷瑞生物工程有限公司;DL2000 DNA Marker,日本宝生物工程(大连)有限公司。
1.2 仪器与设备
PL2002 型电子天平,梅特勒-托利多仪器有限公司;酶标仪,奥地利美谷分子公司;Ruskinn 低氧厌氧培养工作站,英国Ruskinn 公司;BPHG082 型精密恒温培养箱,上海一恒科学仪器有限公司;Bioscreen C 全自动生长曲线分析仪、Univeral Hood Ⅱ型凝胶成像仪,美国Bio-Rad 公司;PCR 仪,上海申安医疗器械厂;DYY-6B 型稳压稳流电泳仪,北京市六一仪器厂。
1.3 试验方法
1.3.1 小鼠粪便样品制备 10 只小鼠适应性饲养一周后进行后续实验,用灭菌后的镊子收取小鼠粪便,并用无菌水将小鼠粪便溶解成6 mg/mL粪便匀浆后分为5 组,每组5 个重复,每个重复3组平行。5 组分别为:空白组、添加1% MRS 培养液组、添加1%胰蛋白胨组、添加1%低聚果糖组以及同时添加1%胰蛋白胨组和1%低聚果糖组,添加的益生元均经过0.22 μm 水相过滤器过滤。
1.3.2 粪便微生物生长曲线的测定 将上述5 组粪便培养液分别取200 μL 放置Bio-screen 全自动生长曲线分析仪的培养板中,37 ℃培养24 h,每组3 个平行,测定时间间隔为3 h。
1.3.3 粪便微生物的体外培养 将粪便培养液分别在添加1%胰蛋白胨低聚果糖和添加1% MRS培养液中进行培养,分别在培养的第8 小时和第24 小时取出100 μL 并用0.86%生理盐水稀释1 000 倍,取100 μL 稀释液在固体MRS 培养基上涂布,并放置在Ruskinn 低氧厌氧培养工作站中,37 ℃培养36 h 后进行单菌落计数。
在固体MRS 培养基上进行划线纯化并挑出纯化分离后的单菌落,将其接种于1 mL MRS 液体培养基中,37 ℃恒温培养12 h 后离心(5 000×g,15 min)收集菌体。用16S rRNA 通用引物27F(5’-RGAGAGTTTGATCCTGGCTCAG-3’)和1492R(5’-CGGCTACCTTGTTACGACTT-3’)进行PCR扩增,PCR 反应条件为:95 ℃预变性3 min;95 ℃变性30 s,58 ℃退火40 s,72 ℃延伸40 s,执行30个循环;最后72 ℃维持10 min。将扩增后的PCR产物用1%琼脂糖凝胶电泳检测。将PCR 产物寄送至生工生物工程股份有限公司进行测序,测序的结果与NCBI 数据库中进行局部序列比对。
1.3.4 粪便微生物的多样性检测 参考文献[15]报道的方法,采用E.Z.N.A. soil 试剂盒使用说明书对小鼠粪便的总DNA 进行提取,利用NanoDrop2000 对DNA 浓度和纯度进行检测,利用1%琼脂糖凝胶电泳检测DNA 提取的质量。以16S rDNA 的中V3-V4 可变区域为目标片段进行PCR 扩增[16-17],引物为338F(5'-ACTCCTACGGGAGGCAGCAG-3')和806R(5'-GGACTACHVGGG-TWTCTAAT-3'),使用2%琼脂糖凝胶回收PCR产物,利用AxyPrep DNA 凝胶提取试剂盒切胶回收纯化,Tris-HCl 洗脱并采用2%琼脂糖电泳检测。利用QuantiFluorTM-ST 进行检测定量。根据Illumina MiSeq 平台标准操作规程将纯化后的扩增片段构建PE 2*300 的文库。
soil 试剂盒使用说明书对小鼠粪便的总DNA 进行提取,利用NanoDrop2000 对DNA 浓度和纯度进行检测,利用1%琼脂糖凝胶电泳检测DNA 提取的质量。以16S rDNA 的中V3-V4 可变区域为目标片段进行PCR 扩增[16-17],引物为338F(5'-ACTCCTACGGGAGGCAGCAG-3')和806R(5'-GGACTACHVGGG-TWTCTAAT-3'),使用2%琼脂糖凝胶回收PCR产物,利用AxyPrep DNA 凝胶提取试剂盒切胶回收纯化,Tris-HCl 洗脱并采用2%琼脂糖电泳检测。利用QuantiFluorTM-ST 进行检测定量。根据Illumina MiSeq 平台标准操作规程将纯化后的扩增片段构建PE 2*300 的文库。
1.3.5 数据统计分析 对本实验中得到的结果进行统计分析,利用Origin 9.0 软件处理数据并作图。
2 结果与分析
2.1 不同营养物质对粪便微生物生长的影响
按照1.3.1 节所述的方法添加相应的益生元,利用Bio-screen 全自动生长曲线分析仪测定OD600nm,反应粪便微生物生长情况。如图1 所示,粪便微生物在无额外添加(空白组)以及添加了1%低聚果糖组中无生长,而添加1% MRS 组迅速进入对数生长期并在6 h 后进入稳定期,表明其对粪便微生物的生长具有明显的促进作用。添加1%胰蛋白胨组的粪便微生物在前9 h 内无明显生长,随后生长迅速,而与MRS 组相比生长缓慢;添加1%胰蛋白胨+1%低聚果糖组在第3 小时开始进入对数生长期并在15 h 后进入稳定期,OD 值介于添加1% MRS 组和添加1%胰蛋白胨组之间。这些结果表明小鼠粪便中自身的物质无法提供粪便微生物生长所需的营养,而单独添加氮源时,粪便微生物显著生长,单独添加碳源时,粪便微生物不生长,这说明氮源是粪便微生物生长的限制性因子,也证实了氮源是微生物生长中不可或缺的营养物质之一[18]。同时添加碳源和氮源组与添加1% MRS 组生长情况相似,具有经典的延滞期、指数生长期以及稳定期,这些结果表明MRS中的其它营养物质不是影响粪菌生长的重要限制性因子,也说明粪便本身也能够提供这些营养物质。
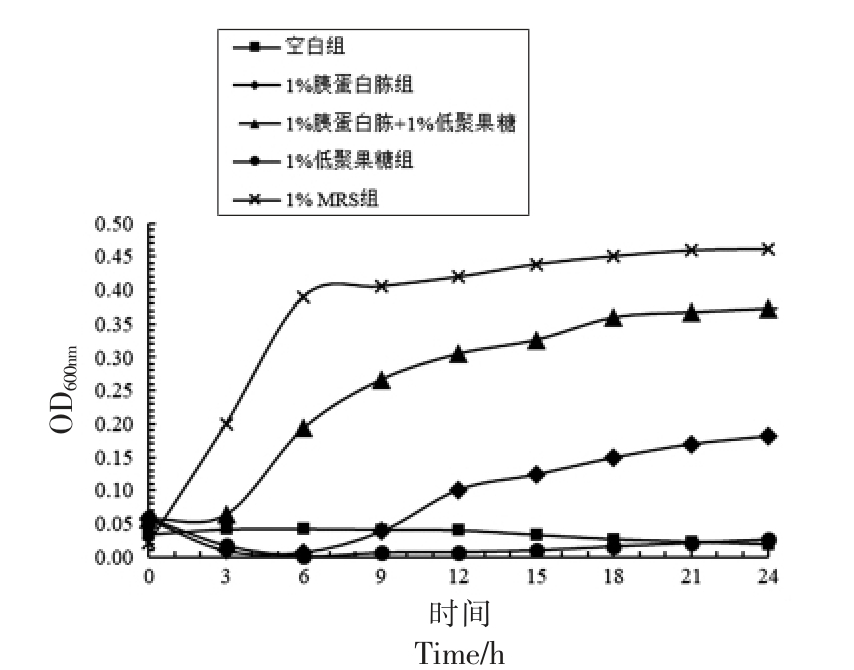
图1 不同C 源、N 源添加组粪便微生物生长曲线
Fig.1 Growth curves of fecal microorganisms with different carbon source and nitrogen source
2.2 不同方式富集粪便微生物后体外培养及鉴定研究
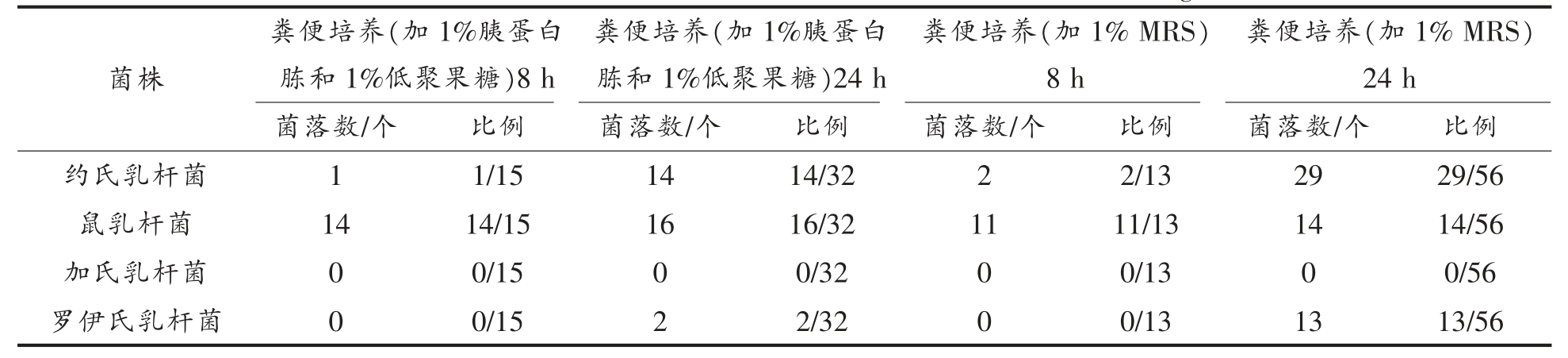
在只添加氮源体外培养时,筛选结果主要集中在致病菌镓肠球菌等,而乳杆菌数量很少,表明单独添加氮源不适合用于肠道乳杆菌的筛选(图1),因此没有对其进一步的研究。对比1%胰蛋白胨+1%低聚果糖组和1% MRS 组,可以发现培养至8 h 时,这两组的乳杆菌数量和种类相似,而在培养24 h 后,约氏乳杆菌和罗伊氏乳杆菌的数量显著增加,表明富集培养后乳杆菌的数量增加,尤其是添加MRS 组,约氏乳杆菌占主导地位。
表1 粪便微生物体外培养鉴定结果
Table 1 Results of in vitro culture and identification of fecal microorganisms
2.3 不同方式富集后粪便微生物的多样性研究
2.3.1 16S rRNA 测序质量评估 稀释曲线可评估样本的测序量是否足够且合理,同时也可以间接反映不同样本的物种丰富度[19]。依据送测的数据量以及分类水平下的物种量进行作图,得到稀释性曲线如图2 所示,所选的指数为sobs 指数和Shannon 指数。
由图2 可知,sobs 指数显示随着抽取样品的测序量增多,观测到的物种数量也在不断增加;Shannon 指数表现出曲线随样品序列数目的增加趋于平坦,表明测序数据量趋于饱和,表明本次测序数量饱和,质控较好,测序结果能够反映粪便微生物中菌群的多样性信息以便满足后续分析的要求。
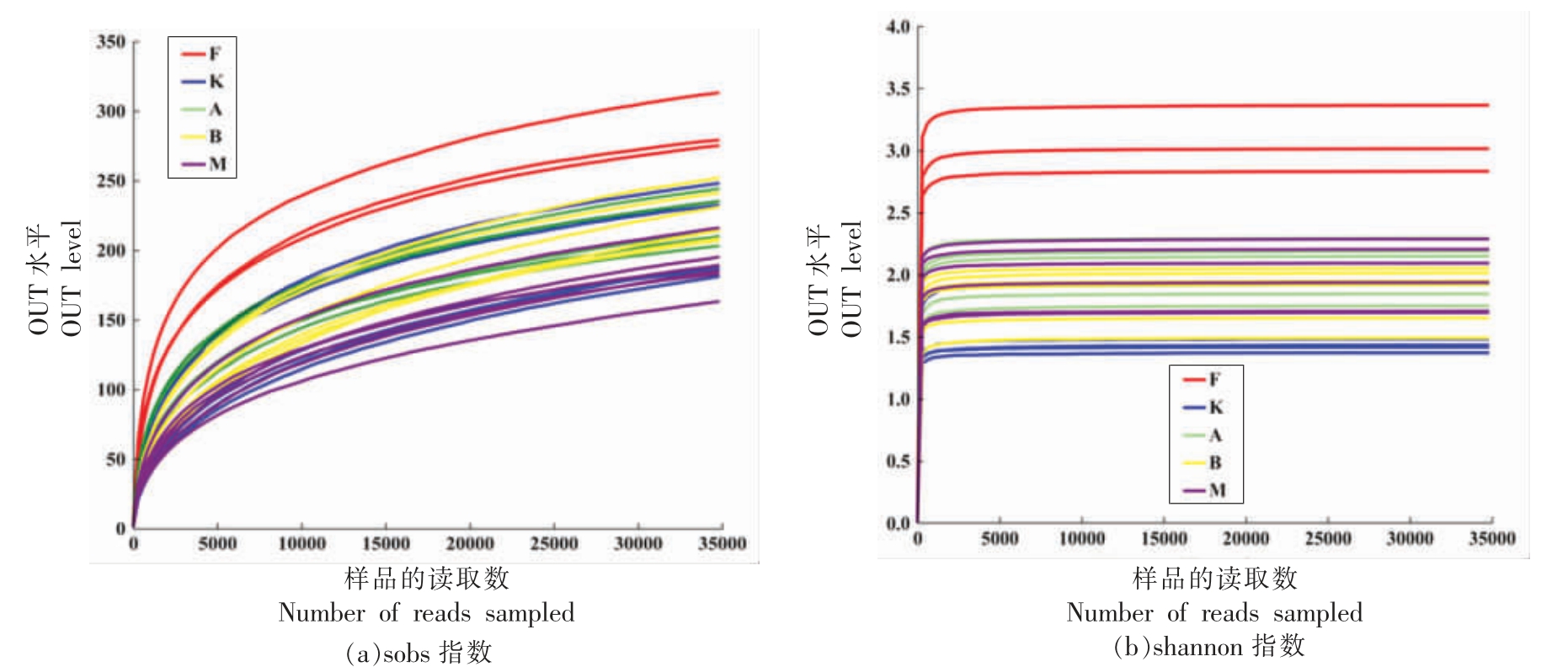
图2 各组粪便样品测序的稀释性曲线
Fig.2 Rarefaction curves of fecal sample in each group
注:F.正常小鼠粪便;K.空白组;A.添加1%胰蛋白胨组;B.添加1%胰蛋白胨和1%低聚果糖组;M.添加1% MRS 组,下同。
2.3.2 基于门、属分类水平的分析 根据微生物物种组成分析可知样本在各分类水平上的比对情况[20]。根据统计学的分析方法,能够观察到样本在门、纲、目、科、属这5 个水平的群落结构[21]。为了进一步分析粪便微生物菌的群落组成结构,对不同分类水平上优势菌的相对丰度进行统计分析,本文重点分析了粪便微生物在门水平和属水平的群落结构,结果如图3 所示。
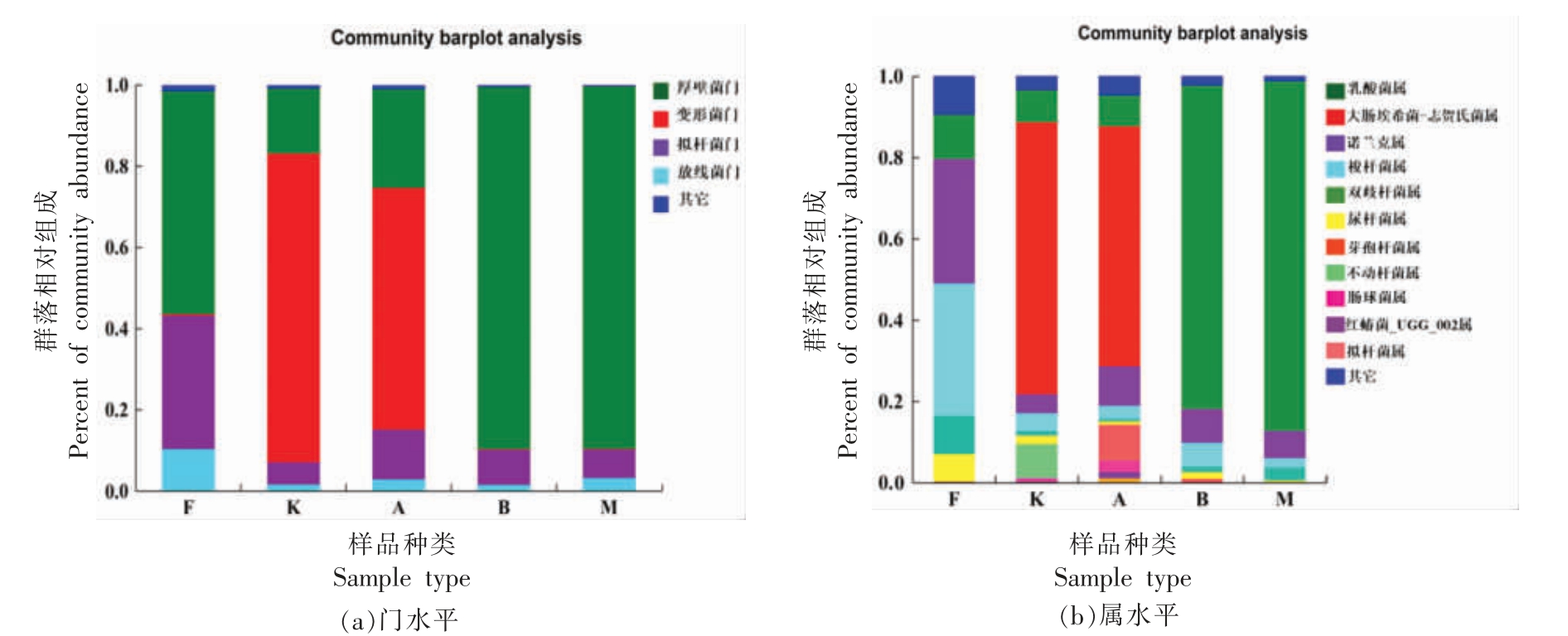
图3 粪便微生物在门水平与属水平的群落组成
Fig.3 The community structure of fecal microorganisms at the phylum level and genus level
图3a 显示粪便微生物在门水平上的群落组成,与正常小鼠的粪便组成相比,其它4 组的拟杆菌(Bacterodietes)的百分比均表现出明显下降。添加了1%胰蛋白胨组和添加1% MRS 组在门水平上的群组成较为相似,而与正常小鼠粪便群落结构相比,厚壁菌门(Firmicutes)的丰度明显升高,从54.90%上升至89.24%和89.38%,其它菌的比例均有不同程度的下降。添加了1%胰蛋白胨组和空白组的变形菌门(Proteobacteria)明显上升并成为主导菌门,物种百分比从0.34%上升至59.55%和76.05%,而厚壁菌门的丰度下降,从54.90%下降到24.25%和15.97%。
图3b 显示粪便微生物在属水平上的群落组成,与正常小鼠粪便组成相比,在属水平上,空白组与添加1%胰蛋白胨组的大肠埃氏菌属-志贺氏菌属(Esherichia-Shigella)明显上升成为主导菌属,占比分别为66.98%和59.09%,而另外两组下降到0.01%;梭杆菌属(Feacalibacterium)由32.53%分别下降至4.38%,3.22%,5.88%和2.37%,说明4种处理方式均不利于梭杆菌属的生长繁殖。双歧杆菌属(Bifidobacterium)由9.46%分别下降至1.13%,0.62%,1.34%和3.14%。乳杆菌属(Lactobacillus)由10.92%变为7.87%,7.59%,79.63%和86.07%,这些结果有力地证实了乳杆菌属的生长同时需要碳源和氮源。综上可得,小鼠肠道内的乳酸菌的生长需要同时添加碳源和氮源。
2.3.3 粪便样品主成分分析 对小鼠分均样品进行主成分分析,通过图中的样品点的距离可以判断个体或群体之间的差异,点距离越近说明样品间微生物群落组成越相似,距离越远说明样品间微生物群落组成差异越大,其结果如图4 所示。
如图4 所示,其它4 组在PC1 和PC2 坐标上均与正常小鼠粪便F 组有一定的距离,说明不同的处理方式,对粪菌组成影响较大,空白组K 与添加1%胰蛋白胨组A 相似度极高,紧密处于主坐标的左下角,证明两组样品的菌落组成相似。添加1%胰蛋白胨+1%低聚果糖组与添加1% MRS 组,在坐标轴的同一位置说明这两组组成相似,然而,与空白组、添加1%胰蛋白胨组相比,群落组成发生了明显的变化。
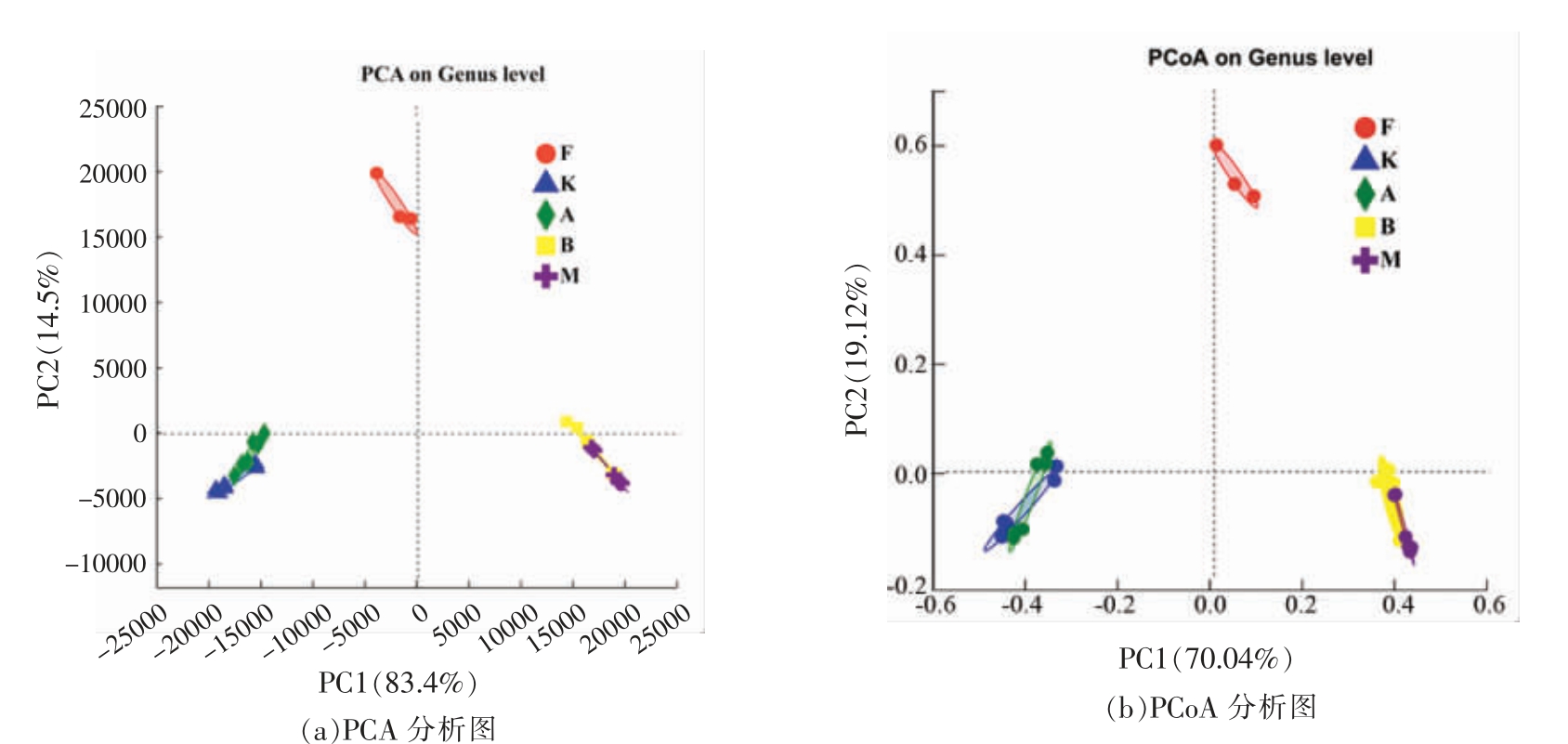
图4 小鼠粪便微生物样品的主坐标分析
Fig.4 Principal component analysis of microorganism in the mice feces
2.3.4 不同培养时间后粪便微生物变化分析 组间重叠部分的Venn 图的能够直观的观察到每组小鼠粪便样品中所共有和独有的OTU 数目,从而直观地反映不同样品微生物群落的相似程度[22]。
如图5 所示,4 组样品共有的OTU 数目为193,独有的数目B1 组为18,B2 组为16,M1 组为12,M2 组为5,这表明随着培养时间的加长,不同水平的粪菌逐渐得到富集,因此,两种处理方式独有的微生物会减少。比较8 h 和24 h 两种处理方案的OTU 数目,B 组OTU 从315 下降到303,M组OTU 从272 下降到255,均呈现下降趋势,也证实了同种处理方式,随时间的加长,粪菌相似性增加特有的微生物减少,因此培养8 h 后进行菌株分析较为合适,否则优势菌株将占绝对优势,减少多样性。M 组整体上OTU 的数目比B 组要低,说明MRS 的富集效果比只添加低聚果糖和胰蛋白胨的富集效果更为显著,而且由于富集显著减少了菌群多样性,因此用胰蛋白胨和低聚果糖处理更加合适。
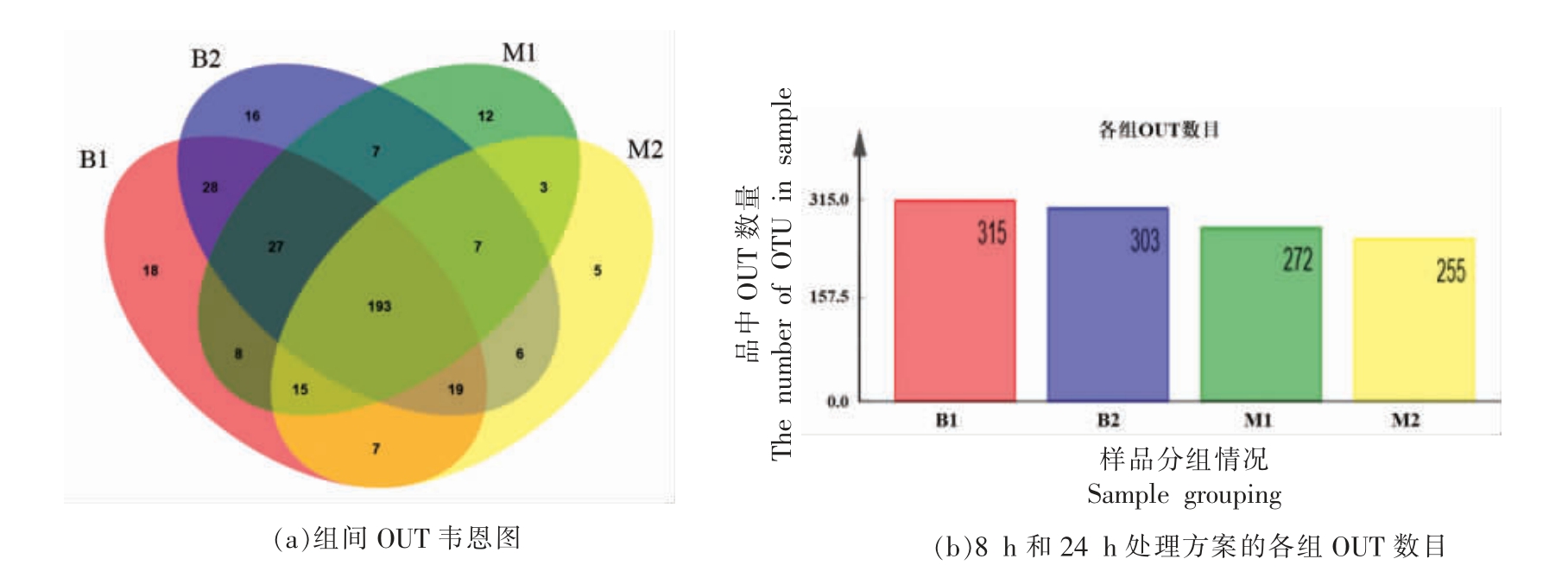
图5 样品中OTU 的数目
Fig.5 The number of OTU in sample
注:B1.添加1%胰蛋白胨+1%低聚果糖培养8 h 的样品;B2.添加1%胰蛋白胨+1%低聚果糖培养24 h 的样品;M1.添加1% MRS 培养8 h 的样品;M2.添加1% MRS 培养液培养24 h 的样品。
3 结论
本实验对小鼠粪便处理,通过添加胰蛋白胨,低聚果糖等来探究小鼠粪便微生物生长所需限制性营养因子,在粪菌的生长过程中,可以看出依赖自身的营养物质是无法生长的,而添加了氮元素的粪菌得到明显生长,表明氮元素是粪菌生长的限制性营养因子,MRS 培养基中除了碳源和氮源之外的其它物质虽会对乳酸菌的生长有一定的有益作用,但不起决定性作用。粪便微生物的体外筛菌的结果表明,单独氮源并不适合粪便微生物的微生物筛选,MRS 虽能有效富集肠道菌群,但是优势菌株占明显优势,尤其是培养24 h 后,菌群多样性明显降低。同时添加低聚果糖和胰蛋白胨培养8 h 后,物种丰度和多样性均较好,此时最适合粪便微生物的体外筛选。
[1] 齐景伟,安晓萍.反刍动物生物饲料开发与应用[M].呼和浩特:内蒙古大学出版社,2014:46.
QI J W,AN X P.Development and application of ruminant biological fee[M].Hohhot:Inner Mongolia University Press,2014:46.
[2] FARIAS D P,ARAUJO F F,NERI-NUMA I A,et al.Prebiotics:Trends in food,health and technological applications[J].Trends in Food Science &Technology,2019,93(9):23-35.
[3] GIBSON G R,HUTKINS R,SANDERS M E,et al.Expert consensus document:The International Scientific Association for Probiotics and Prebiotics(ISAPP)consensus statement on the definition and scope of prebiotics[J].Nature Reviews Gastroenterology &Hepatology,2017,14(8):491-502.
[4] 尚晋伊,刘丽萍.益生元营养及应用研究现状[J].现代食品,2018,64(4):52-55.
SHANG J Y,LIU L P.Research status of prebiotic nutrition and its application[J].Modern Food,2018,64(4):52-55.
[5] NERI-NUMA I A,PASTORE G M.Novel insights into prebiotic properties on human health:A review[J].Food Research International,2020,131(2):108-937.
[6] PAULINA M,KATARZNA  .Effects of probiotics,prebiotics,and synbiotics on human health[J].Nutrients,2017,9(9):1021.
.Effects of probiotics,prebiotics,and synbiotics on human health[J].Nutrients,2017,9(9):1021.
[7] WANG Y B.Prebiotics:Present and future in food science and technology[J].Food Research International,2008,42(1):8-12.
[8] ANDREA M A,ROBERT A R,GLENN R,et al.Adhesion mechanisms mediated by probiotics and prebiotics and their potential impact on human health[J].Applied Microbiology and Biotechnology,2019,103(16):6463-6472.
[9] 沈定树,郑静.益生元与肠道微生态[J].中国微生态学杂志,2013,25(6):128-130.
SHEN D S,ZHENG J.Probiotics and intestinal microecology[J].Chinese Journal of Microecology,2013,25(6):128-130.
[10] RICHARD Y W,MICHAEL P J,KATHENE C,et al.Impact of prebiotics,probiotics,and gut derived metabolites on host immunity[J].Microbes in Food and Health,2016,4(1):1-24.
[11] YAHFOUFI N,MALLET J F,GRAHAM E,et al.Role of probiotics and prebiotics in immunomodulation[J].Current Opinion in Food Science,2018,20(4):82-91.
[12] 王艺苑,林姗姗,热依拉·阿巴拜克热,等.不同浓度低聚半乳糖对初断乳大鼠肠道菌群的影响[J].食品研究与开发,2019,40(5):17-22.
WANG Y Y,LING S S,RAYILA A,et al.Effects of different concentrations of galactooligosaccharides on intestinal flora of weaned rats[J].Food Research and Development,2019,40(5):17-22.
[13] SADEQ H A,AMIN I,MOHD Y M,et al.Prebiotics as functional foods:A review[J].Journal of Functional Foods,2013,5(4):1542-1553.
[14] 李俊,王军,万艳,等.基于GC/TOF-MS 技术对流感感染小鼠粪便代谢组学的研究[J].畜牧与兽医,2019,51(4):115-120.
LI J,WANG J,WANG Y,et al.GC/TOF-MS based study on fecal metabonomics of mice infected with influenza[J].Animal Husbandry and Veterinary Medicine,2019,51(4):115-120.
[15] SHEN B,WU Z F,LI H T,et al.Effects of inoculated starter of lactic acid bacteria on quality and microbial diversity of pickled wax gourd in eastern Zhejiang[J].Journal of Food Processing and Preservation,2016,41(2):1-9.
[16] GREGORY C J,CHRISTIAN L L,WILLIAM A W,et al.Global patterns of 16S rRNA diversity at a depth of millions of sequences per sample[J].Proceedings of the National Academy of Sciences of the United States of America,2011,108(S1):4516-4522.
[17] LIANG T T,XIE X Q,ZHANG J M,et al.Bacterial community and composition of different traditional fermented dairy products in China,South Africa,and Sri Lanka by high-throughput sequencing of 16S rRNA genes[J].LWT,2021,144(7):111209.
[18] 杜丽红,郝亚男,陈宁,等.有机氮源及其在微生物发酵中的应用[J].发酵科技通讯,2019,48(1):1-4.
DU L H,HE Y N,CHEN N,et al.Organic nitrogen source and its application in microbial fermentation[J].Bulletin of Fermentation Science and Technology,2019,48(1):1-4.
[19] AMATO K R,YEOMAN C J,KENT A,et al.Habitat degradation impacts black howler monkey(Alouatta pigra)gastrointestinal microbiomes[J].ISME Journal,2013,7(7):1344-1353.
[20] LI A Y,WANG Y P,PEI L L,et al.Influence of dietary supplementation with Bacillus velezensis on intestinal microbial diversity of mice[J].Microbial Pathogenesis 2019,136(16):103671.
[21] 刘冰.不同聚合度菊粉对健康肠道微生物群落结构影响的研究[D].兰州:兰州大学,2018.
LIU B.Effect of inulin with different polymerization degree on microbial community structure in healthy intestines[D].Lanzhou:Lanzhou University,2018.
[22] LI Y,CHEN Q Q,CHEN Z,et al.Altered microbial community structure in PI3Kγ knockout mice with colitis impeding relief of inflammation:Establishment of new indices for intestinal microbial disorder[J].International Immunopharmacology,2020,2020(79):105901.