酱油是起源于中国的传统发酵调味品,其独特的鲜味备受人们的青睐,成为中国、日本、韩国等亚洲国家常用的调味品,并逐渐被西方国家所接受[1]。酱油的生产涉及霉菌、细菌、酵母等多种微生物的协同作用,混菌发酵体系中的微生物对酱油发酵进程和风味形成至关重要[1-2]。微生物作为酱油生产的核心,其群落多样性一直是研究的热点。自20 世纪初,日本学者从酱醪中分离培养乳酸菌以来[3],研究者采用传统微生物培养法[4]、磷脂脂肪酸分析法[5]、聚合酶链式反应-变性梯度凝胶电泳技术(polymerase chain reaction-denatured gradient gel electrophoresis,PCR-DGGE)[6]、高通量测序技术[7]等多种方法对酱油微生物多样性进行研究[8]。目前,酱油生产过程中的优势微生物已比较明晰,优势细菌属包括:魏斯氏菌属(Weissella)[9-11]、葡萄球菌属(Staphylococcus)[6,12]、库特氏菌属(Kurthia)[13]、芽孢杆菌属(Bacillus)[4]、四联球菌属(Tetragenococcus)[7]等;优势真菌属包括:曲霉属(Aspergillus)[10]、假丝酵母属(Candida)[9]、接合酵母属(Zygosaccharomyces)[10]等。然而,酱油品种繁多,风味各异,这与其特有的酱油酿造微生态体系息息相关,发酵环境和条件的变化影响微生物群落结构,且微生物群落丰度在酱油生产的不同阶段也各不相同,进而影响酱油的风味[1]。科学认识酱油生产中微生物群落的多样性、演替规律,对调控菌群,提升酱油风味和品质具有重要意义。
酱油生产工艺主要分为3 个阶段:制曲、酱醪发酵和淋油[1,14]。制曲是在大豆和小麦原料中添加米曲霉或酱油曲霉进行固态发酵[3]。酱曲微生物多样性研究显示,细菌属较丰富,主要包括魏斯氏菌属、葡萄球菌属、库特氏菌属等,真菌属主要是添加的曲霉,同时含有低丰度的酵母[6,10,12-13]。酱醪发酵是在成熟的酱曲中加入盐水进行长时间发酵,时间一般为几个月,最长可达4 年[1,4]。酱醪微生物多样性研究显示:大部分微生物来自于酱曲,而群落多样性高于酱曲[13]。酱醪发酵时间长,根据微生物变化分为乳酸和乙醇发酵两个阶段,乳酸发酵阶段的细菌以乳酸菌为主,真菌主要是曲霉;乙醇发酵阶段细菌量减少,酵母成为优势真菌[10]。淋油是将发酵成熟的酱醅通过压榨或浸提的方式获得酱油原油[14],该阶段的微生物研究鲜见报道。
酿造酱油半开放式的发酵体系中,来源复杂且多样的微生物以发酵原料为基质进行代谢,在添加盐水的干预作用下进行选择性增殖,形成微生物群落演替并推动发酵的进程[1,13]。酱曲和酱醪的微生物多样性研究,丰富了人们对制曲和酱醪发酵阶段微生物群落结构、时间轴上演替规律的认识。然而,在食品发酵过程中,不同空间位置的差异也会影响微生物的定殖、繁衍与代谢,且微生物群落空间异质性对风味形成起着重要作用[15-16]。目前酱油酿造中的微生物空间分布规律尚未明确。本研究跟踪酱油主产区的广东某酱油厂中同一批次样品,利用高通量测序技术结合生物信息学方法,分析制曲、酱醪发酵和淋油阶段的微生物群落变化,并解析酱醪发酵阶段的上、中、下层的微生物群落组成,以期揭示酱油发酵主要阶段的微生物演替规律和空间异质性,为优化酱油生产工艺,提升酱油品质提供科学依据。
1 材料与方法
1.1 材料与试剂
酱油发酵样品,采自广东某酱油厂。定期跟踪同一批次高盐稀态酱油生产不同阶段的样品,样品采集和处理参照Wang 等[13]方法。样品分别为:制曲阶段(S1),酱醪发酵时的乳酸发酵阶段上、中、下层酱醪(S2、S3、S4),酱醪发酵时的乙醇发酵阶段上、中、下层酱醪(S5、S6、S7),淋油阶段(S8)。
DNA 提取试剂盒(FastDNA 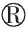 Spin Kit for Soil),美国MP Biomedicals 公司;琼脂糖(Biowest Agarose),法国Biowest 公司;FastPfu Polymerase,北京全式金生物技术有限公司;AxyPrep DNA 凝胶回收试剂盒,美国Axygen 公司。
Spin Kit for Soil),美国MP Biomedicals 公司;琼脂糖(Biowest Agarose),法国Biowest 公司;FastPfu Polymerase,北京全式金生物技术有限公司;AxyPrep DNA 凝胶回收试剂盒,美国Axygen 公司。
1.2 仪器与设备
NanoDrop2000 超微量分光光度计,美国Thermo Fisher Scientific公司;QuantusTM 荧光计,美国Promega 公司;BioTek ELx800 酶标仪,美国Biotek 公司;DYY-6C 电泳仪,北京市六一仪器厂;GeneAmp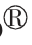 9700 型PCR 扩增仪,美国ABI 公司;Illumina Miseq 测序仪,美国Illumina 公司;5424R 高速冷冻离心机,德国Eppendorf 公司。
9700 型PCR 扩增仪,美国ABI 公司;Illumina Miseq 测序仪,美国Illumina 公司;5424R 高速冷冻离心机,德国Eppendorf 公司。
1.3 方法
1.3.1 基因组DNA 提取 酱油发酵样品总DNA用FastDNA Spin Kit for Soil 试剂盒提取,按照试剂盒说明书操作流程进行。用分光光度计和1%琼脂糖凝胶电泳检测DNA 的提取质量。
Spin Kit for Soil 试剂盒提取,按照试剂盒说明书操作流程进行。用分光光度计和1%琼脂糖凝胶电泳检测DNA 的提取质量。
1.3.2 PCR 扩增及测序 以酱油发酵样品总DNA 为模板,用引物 338F(5' -ACTCCTACGGGAGGCAGCAG-3')和806R(5'-GGACTACHVGGGTWTCTAAT-3')对细菌16S rRNA 基因V3-V4 区进行PCR 扩增;用引物ITS1F(5'-CTTGGTCATTTAGAGGAAGTAA-3')和ITS2R(5'-GCTGCGTTCTTCATCGATGC-3')对真菌ITS1 区进行扩增。PCR 扩增程序:95 ℃预变性3 min,27个循环(95 ℃变性30 s,55 ℃退火30 s,72℃延伸45 s),72 ℃稳定延伸10 min。PCR 扩增体系(20 μL):5×FastPfu Buffer 4 μL,2.5 mmol/L dNTPs 2 μL,5 μmol/L 上游引物0.8 μL,5 μmol/L 下游引物0.8 μL,FastPfu 聚合酶0.4 μL,BSA 0.2 μL,模板DNA 10 ng,补足ddH2O 至20 μL。PCR 产物的建库与测序在上海美吉生物医药科技有限公司完成,测序平台为Illumina Miseq PE300。
1.3.3 数据分析 使用Fastp 等软件对原始测序序列进行质控和拼接。优化的数据使用Uparse 软件,在97%的相似度水平下进行操作分类单元(operational taxonomic units,OTU)聚类分析。用RDP classifier 对OUT 代表序列进行物种分类注释,细菌比对Silva 数据库,真菌比对Unite 数据库,置信度阈值设置为0.7。基于分类学信息,在各分类水平上进行群落结构的统计分析。使用Qiime、Mothur、Mega 等软件绘制样品序列的菌群可视化分析图。以上分析在上海美吉生物医药科技有限公司的生信云分析平台完成(https://cloud.majorbio.com/)。
2 结果与分析
2.1 多样性分析结果
2.1.1 微生物的Alpha 多样性 采用MiSeq 高通量测序方法分析酱油生产中制曲、酱醪发酵(乳酸发酵上、中、下层,乙醇发酵上、中、下层)、淋油阶段的微生物多样性。通过细菌16S rRNA V3-V4和真菌ITS1 测序,分别优化获得280 352 和430 288 条有效序列,序列经OTU 聚类与注释,分别获得271,27 个OTU。所有酱油发酵样品的覆盖率均大于0.99(表1),表明样本文库覆盖率高,本次测序结果可真实反应样品中的细菌和真菌群落。
多样性指数是反映物种丰富度和均匀度的综合指标,通过Alpha 多样性指数分析,比较酱油生产样品中物种的多样性。OTU 数和Chao 指数反映群落中的物种数,数值越大微生物群落的丰富度越高;Shannon 指数反映群落中物种的丰富度和均匀度,数值越大微生物群落的多样性越高[17]。由表1 可知,细菌的多样性指数高于真菌,表明酱油生产中细菌的群落丰富度和均匀度高于真菌,与文献[10]报道的成曲和酱醪中细菌比真菌更丰富的结果一致。表1 中细菌多样性指数显示,酱醪发酵和淋油阶段的OTU 数、Chao 指数和Shannon 指数高于制曲阶段,且酱醪发酵上层的指数高于下层。真菌多样性指数显示,乙醇发酵和淋油阶段的OTU 数、Chao 指数和Shannon 指数高于制曲,制曲阶段指数高于乳酸发酵阶段,且酱醪发酵下层的指数高于上层。细菌和真菌的Alpha 多样性分析表明:时间轴上,制曲阶段的细菌和真菌多样性较低,酱醪发酵和淋油阶段较高;空间上,酱醪发酵阶段上、中、下层的细菌和真菌多样性有差异,细菌多样性上层较高,真菌多样性下层较高。
表1 酱油发酵样品中细菌和真菌多样性指数表
Table 1 Diversity indexes of bacteria and fungi in soy sauce

2.1.2 微生物的Beta 多样性分析 采用Weighted UniFrac 距离算法衡量不同样品间物种组成的相似性[18],并通过非加权组平均法(Unweighted Pair-group Method with Arithmetic Mean,UPGMA)对样品进行聚类分析,研究酱油生产各阶段样品间群落组成的相似性或差异性。细菌层级聚类图1a 显示,制曲阶段样品S1 自成一类,酱醪发酵和淋油阶段由两小类聚为一大支,其中酱醪发酵上层样品(S2 和S5)聚为一类,酱醪发酵中、下层和淋油阶段样品(S3、S4、S6~S8)聚为一类。结果表明:时间轴上,制曲阶段的细菌与其它阶段有差异,而酱醪发酵与淋油阶段的细菌相似;空间上,酱醪上层的细菌与中、下层有差异,而中层与下层相似性高。真菌层级聚类图1b 显示,所有样品聚为两大支,制曲、酱醪发酵上层和中层样品(S1~S5)聚为一类,酱醪发酵下层与淋油阶段样品(S6~S8)聚为一类。结果表明:时间轴上,制曲与酱醪乳酸发酵阶段的真菌相似,且酱醪酒精发酵与淋油阶段的真菌相似;空间上,酱醪上层与中层的真菌相似性高,而上、中层与下层相似性低。细菌和真菌的Beta 多样性分析表明,酱油生产微生物的物种进化关系及丰度随时间和空间而变化,其中Beta 多样性随时间变化与LIU 等[11]研究结果相似。
图1 酱油发酵样品中细菌(a)和真菌(b)层级聚类树
Fig.1 Hierarchical clustering tree of bacteria(a)and fungi(b)in soy sauce
2.2 群落分布与组成分析
2.2.1 微生物的群落分布 在属分类水平下,从所有酱油发酵样品中共检测到147 个细菌属和19 个真菌属。采用韦恩图统计酱油生产时间轴上制曲(K)、乳酸发酵(MA)、乙醇发酵(MB)、淋油(S)阶段和空间上酱醪发酵上层(MU)、中层(MM)、下层(ML)样品的独有和共有微生物属,比较酱油生产过程中微生物属数目组成的相似性和重叠情况,结果如图2 和图3 所示。
细菌韦恩图2a 显示,酱油生产各阶段共有细菌属21 个,占细菌属总数的14.29%;乳酸发酵、乙醇发酵与淋油阶段共有细菌属55 个;乳酸与乙醇发酵阶段共有细菌属17 个;4 个阶段细菌特有菌属分别为:制曲1 个、乳酸发酵19 个,乙醇发酵12 个,淋油6 个。时间轴上,酱油生产制曲阶段,除1 个细菌属外,其余细菌进入后续发酵和淋油阶段,与Wang 等[13]报道的酱醪中大部分细菌来源于酱曲的结果一致。同时,乳酸发酵、乙醇发酵和淋油阶段中出现多种其它细菌属参与发酵,且这3个阶段的细菌群落具有一定的相似性。细菌韦恩图2b 显示,酱醪发酵上、中、下层共有细菌属38个,占细菌属总数的27.14%;上层与中层共有细菌属22 个;3 个空间位置特有细菌属分别为:上层60 个,中层6 个,下层3 个。空间上,酱醪发酵阶段的上、中、下层细菌属数目依次减小,上层与中层的细菌群落具有一定的相似性,上层特有细菌属数目较多,表明酱醪原核微生物组成具有一定的空间异质性。

图2 酱油生产不同发酵阶段(a)和不同位置(b)样品中细菌属水平韦恩图
Fig.2 Venn diagram of bacteria in soy sauce at genus level from different fermentation stages(a)and locations(b)
真菌韦恩图3a 显示,酱油生产中各阶段共有真菌属4 个,占真菌属总数的21.05%;仅乙醇发酵和淋油阶段存在特有真菌属,分别为6 个和2个。表明时间轴上,酱油生产制曲阶段中的全部真菌属进入后续阶段,且乙醇发酵阶段中出现多种其它真菌属参与发酵。真菌韦恩图3b 显示,酱醪发酵上、中、下层共有真菌属4 个,占细菌属总数的23.53%;3 个空间位置特有真菌属分别为:上层1 个,中层3 个,下层7 个。表明空间上,酱醪发酵阶段的上、中、下层真菌属数目依次增大,下层特有真菌属数目较多。
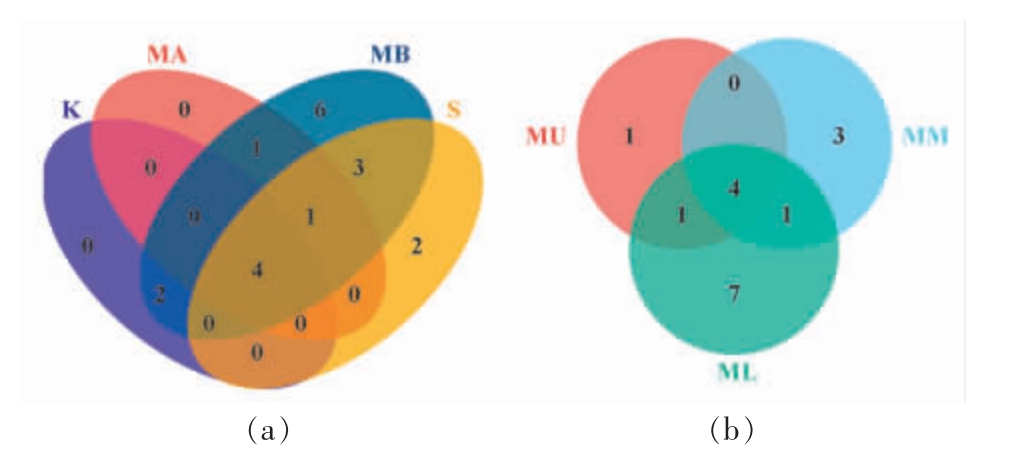
图3 酱油生产不同发酵阶段(a)和不同位置(b)样品中真菌属水平韦恩图
Fig.3 Venn diagram of fungi in soy sauce at genus level from different fermentation stages(a)and locations(b)
2.2.2 微生物的群落组成 在属分类水平下,统计各样品的细菌和真菌组成及各物种相对丰度,将丰度低于1%的合并为一类(Others),结果如图4 所示。将样品中相对丰度≥5%的属定义为优势属。根据相对丰度划分出各样品的优势细菌和真菌,结果见表2。
图4a 细菌属水平相对丰度显示,在相对丰度≥1%阈值下,共得到10 个细菌属,分别为:葡萄球菌属(Staphylococcus)、魏斯氏菌属(Weissella)、四联球菌属(Tetragenococcus)、假单胞菌属(Pseudomonas)、库特氏菌属(Kurthia)、考克氏菌属(Kocuria)、肠杆菌属(Enterobacter)、棒状杆菌属(Corynebacterium)、鱼果菌属(Piscicoccus)、明串珠菌属(Leuconostoc)。除鱼果菌属外,其余菌属在酱油微生物研究文献[9-10,13]中均有报道。Piscicoccus中文属名为鱼果菌属[19],该属仅有1 个种肠鱼果菌(Piscicoccus intestinalis),分离自鱼肠道[20]。本试验中鱼果菌属为发酵食品中首次报道,该菌属仅在S2(酱醪发酵时乳酸发酵阶段上层酱醪)样品中丰度较高(1.2%),其余样品的丰度小于1%,推测环境中的鱼果菌属在乳酸发酵时期从上层酱醪进入酱油发酵体系。表2 优势细菌显示,时间轴上细菌的相对丰度变化较大,魏斯氏菌属为制曲阶段绝对优势菌属(相对丰度71.27%),进入后续发酵阶段而急剧下降(相对丰度15.96%);葡萄球菌属随时间而逐渐升高,并成为酱醪发酵和淋油阶段的绝对优势菌属(相对丰度63.40%~83.36%);四联球菌属仅在酱醪发酵和淋油阶段大量存在(相对丰度8.20%~21.76%)。本试验中,制曲阶段的绝对优势是魏斯氏菌,与胡传旺[10]对酱曲高通量测序结果一致;酱醪发酵阶段的绝对优势是葡萄球菌,与WANG 等[13]的酱醪优势细菌结果一致,而与胡传旺[10]和LIU 等[11]报道的酱醪优势细菌为魏斯氏菌结果有差异,推测酱醪发酵阶段的时间和环境差异会引起菌群丰度的变化[1]。空间上,乳酸发酵阶段中,魏斯氏菌属丰度上层高,葡萄球菌属丰度中层高,四联球菌属丰度下层高;乙醇发酵阶段中,魏斯氏菌属丰度上层高,葡萄球菌属丰度下层高,四联球菌属丰度中层高。表明优势细菌丰度在不同层间存在差异,推测这种差异性与不同层间营养和条件的差异有关[16]。
图4b 真菌属水平相对丰度和表2 优势真菌显示,曲霉属(Aspergillus)和接合酵母属(Zygosaccharomyces)为酱油生产的主要真菌属。时间轴上,乙醇发酵和淋油阶段的真菌属的相对丰度变化较大。曲霉属为制曲和乳酸发酵阶段的绝对优势菌属(相对丰度>99%),并在酱油生产后期明显下降(相对丰度最低为25.03%);而接合酵母属后期明显上升(相对丰度36.83%~74.95%),并成为优势真菌之一。所得结果与文献中酱油发酵真菌高通量测序结果一致[10]。空间上,乳酸发酵阶段的真菌组成变化不大;乙醇发酵阶段的曲霉属相对丰度上层>下层>中层,接合酵母属变化相反。

图4 酱油发酵样品中细菌(a)和真菌(b)属水平群落组成
Fig.4 Distribution of bacterial(a)and fungal(b)community in soy sauce at genus level during fermentation
表2 发酵过程中酱油样品的优势细菌和真菌属
Table 2 Dominant bacterial and fungal genus in soy sauce samples during fermentation

2.3 功能预测结果
通过PICRUSt(Phylogenetic Investigation of Communities by Reconstruction of Unobserved States)软件分别对16S rRNA 和ITS 测序结果进行微生物群落功能预测,得到细菌COG 功能相对丰度(图5)和真菌MetaCyc pathway 相对丰度(表3)。
细菌PICRUSt 功能预测图5 显示,忽略未知功能(F18)后,得到22 个功能基因家族,主要包括:能量产生与转化(F3,5.65%~6.46%),氨基酸运输和代谢(F5,9.97%~11.03%),碳水化合物运输和代谢(F7,6.69%~7.85%),翻译、核糖体结构和生物合成(F10,6.78%~8.54%),转录(F11,6.51%~6.90%),复制、重组和修复(F12,4.81%~5.95%),细胞壁/膜/被膜生物合成(F13,5.00%~5.35%),无机离子运输和代谢(F16,6.68%~7.45%)。这8 类主要功能基因家族占比之和在50%以上,其中,氨基酸运输和代谢相对丰度最高。酱油发酵各样品氨基酸运输和代谢的相对丰度排序为S5(乙醇发酵上层)>S2(乳酸发酵上层)>S7(乙醇发酵下层)>S8(淋油)>S6(乙醇发酵中层)>S3(乳酸发酵中层)>S4(乳酸发酵下层)>S1(制曲)。细菌COG 功能预测结果表明:酱油发酵体系中的细菌功能主要集中在氨基酸代谢,时间轴上,酱醪发酵和淋油阶段的细菌氨基酸代谢相对丰度高于制曲;空间上,酱醪上层的细菌氨基酸代谢相对丰度高于中、下层。氨基酸是酱油鲜味的主要来源,而酱油中的氨基酸由微生物代谢产生[3,21]。结合图4a 中酱醪发酵和淋油阶段的葡萄球菌丰度较高,表2 中上层魏斯氏菌丰度较高,表明葡萄球菌和魏斯氏菌对酱油氨基酸的形成具有重要作用。
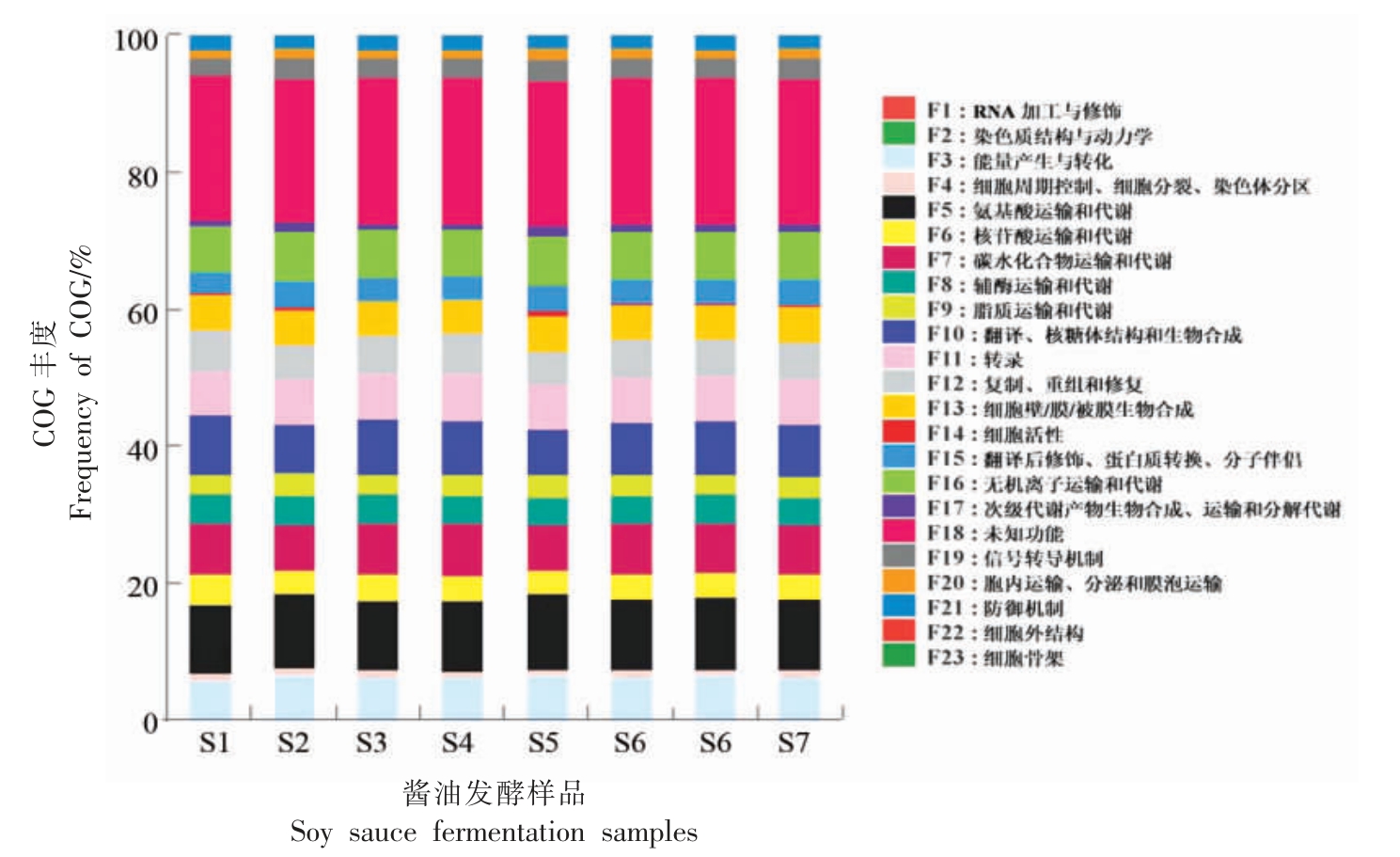
图5 酱油发酵样品细菌功能预测的COG 相对丰度
Fig.5 Relative abundance of bacterial genus predicted COG functions in soy sauce
酱油酿造样品真菌PICRUSt 功能预测的通路如表3 所示。其中,脂质代谢相关的途径有脂肪酸β 氧化、甲基酮生物合成、乙醛酸循环和三羧酸循环;碳水化合物代谢相关的途径有磷酸戊糖途径、GDP 甘露糖生物合成和三羧酸循环。真菌Meta-Cyc pathway 功能预测结果表明:酱油发酵体系中的真菌功能主要集中在脂肪和碳水化合物代谢,这些相关的代谢通路,时间轴上,制曲和乳酸发酵阶段丰度一致,乙醇发酵和淋油阶段丰度主要呈下降趋势;空间上,乳酸发酵阶段上、中、下层的丰度一致,乙醇发酵阶段中层的大部分通路丰度较低。酱油发酵体系中的真菌主要来源于制曲时添加的曲霉,曲霉具有淀粉酶、脂肪酶、蛋白酶等多种复合酶系,可将酱油原料分解为糖类、有机酸、氨基酸等成分[10,14]。结合图4b 中制曲和乳酸发酵阶段的曲霉丰度较高,表2 中乙醇发酵阶段上层的曲霉丰度较高,表明曲霉对酱油原料中脂肪和碳水化合物的分解作用主要集中于制曲和乳酸发酵阶段,曲霉在乙醇发酵阶段的分解作用主要集中在上层。
表3 酱油发酵样品真菌功能预测的MetaCyc pathway 丰度(前10)
Table 3 Relative abundance of fungal genus predicted MetaCyc pathway in soy sauce(top 10)

3 讨论与结论
采用高通量测序,分析酱油酿造过程主要阶段(制曲、酱醪发酵和淋油)的微生物群落演替和酱醪菌群空间分布规律。研究显示,酱油酿造中微生物的多样性、群落分布与组成、预测功能呈现明显的阶段特征和空间异质性。
酱油酿造依次经历制曲、酱醪发酵和淋油阶段,其中微生物群落结构随发酵进程而不断变化[1]。制曲阶段的微生物多样性较低,群落组成与其它阶段有差异,这主要是由于原料经制曲后加入高盐并转入发酵池而引起条件和环境的改变,进而改变了微生物群落结构[1,13]。制曲阶段的核心细菌为魏斯氏菌和葡萄球菌,核心真菌为曲霉,与胡传旺[10]酱曲高通量测序结果、TANAKA 等[22]酱曲PCR-DGGE 结果、YAN 等[12]酱曲微生物培养法结果一致。酱醪发酵阶段的微生物多样性高于制曲,推测酱醪发酵周期长,半开放式的发酵池更有利于酱醪微生物与环境微生物之间的交互作用[1,4],形成更丰富的微生物群落。酱醪发酵阶段的核心细菌为魏斯氏菌、葡萄球菌和四联球菌。酱醪核心细菌种类与文献[5],[7],[10] 类似,然而,多篇文献显示酱醪魏斯氏菌丰度最高[9-11]。本试验葡萄球菌丰度最高,可能与不同工厂的发酵环境差异有关[1]。葡萄球菌在酱油发酵过程中可降低生物胺[23],减少褐变[24],作为发酵肉制品中的常见微生物,能分解脂肪和蛋白质,提供风味前体物质[25]。本试验酱醪中高丰度的葡萄球菌是否影响酱油生物胺、颜色和风味的形成,后续将分离葡萄球菌并进行功能验证。酱醪发酵阶段的核心真菌为曲霉和接合酵母,与之前多篇文献研究结果一致[6,10,22]。淋油阶段的微生物多样性、群落组成、核心微生物与酱醪发酵阶段相似,可能与本试验酱油酿造采用浸提淋油方式有关。综上,魏斯氏菌和葡萄球菌是酱油酿造过程中的优势细菌,曲霉和接合酵母是酱油酿造过程中的优势真菌。
中国传统发酵食品的发酵体系在营养、环境、生态位等方面存在空间差异性,且微生物空间异质性在醋和酒的发酵中已被证实[15-16]。酱油酿造过程的酱醪发酵阶段主要在大型发酵池或发酵罐中完成,容器中混合盐水的酱曲在重力作用、溶氧量、温度等因素的影响下,发酵体系不均一[1,16],体系中微生物的空间分布规律仍未明确。本试验从酱醪发酵阶段发酵池的上、中、下位置取样,分析不同空间位置的酱醪微生物变化。上层发酵样品与空气接触,有利于与外界环境中的物质和微生物进行交换[16]。本试验酱醪上层的细菌多样性和特有菌属数目较高,表明酱油发酵体系中的细菌更容易与环境微生物进行交互作用。中层发酵环境营养物质丰富、发酵条件适宜,适合微生物的生长[26]。本试验酱醪中层的葡萄球菌属和接合酵母属的丰度较高,表明酱油发酵体系中层环境更适合优势菌生长。下层为固、液两相发酵,高盐、高酸环境适合具有耐盐、耐酸的微生物生长[26]。本试验酱醪下层的真菌多样性较高,且这些特有真菌多为酵母,表明酱油发酵体系中的酵母具有较强的耐盐、耐酸特性。综上,酱醪上层细菌多样性较高,酱醪下层真菌多样性较高。
本研究运用高通量测序技术对酱油酿造中的微生物群落进行解析,阐明发酵时间和空间差异对微生物群落的影响,为酱油酿造过程中微生物质量控制、菌群调控、发酵工艺优化等提供前期基础数据。
[1] DEVANTHI P V P,GKATZIONIS K.Soy sauce fermentation:Microorganisms,aroma formation,and process modification[J].Food Research International,2019,120:364-374.
[2] 方冠宇,姜佳丽,蒋予箭.多菌混合发酵对酱油的风味物质形成及感官指标的影响[J].中国食品学报,2019,19(9):154-163.
FANG G Y,JIANG J L,JIANG Y J.The influence of multi-strain fermentation on the flavor substances and sensory evaluation in soy sauce[J].Journal of Chinese Institute of Food Science and Technology,2019,19(9):154-163.
[3] O'TOOLE D K.The role of microorganisms in soy sauce production[J].Advances in Applied Microbiology,2019,108:45-113.
[4] YANG Y,DENG Y,JIN Y L,et al.Dynamics of microbial community during the extremely long-term fermentation process of a traditional soy sauce[J].Journal of the Science of Food and Agriculture,2017,97(10):3220-3227.
[5] ZHANG L Q,ZHOU R Q,CUI R Y,et al.Characterizing soy sauce moromi manufactured by highsalt dilute-state and low-salt solid-state fermentation using multiphase analyzing methods[J].Journal of Food Science,2016,81(11):C2639-C2646.
[6] WEI Q Z,WANG H B,CHEN Z X,et al.Pro?ling of dynamic changes in the microbial community during the soy sauce fermentation process[J].Applied Microbiology and Biotechnology,2013,97(20):9111-9119.
[7] QI Q,HUANG J,ZHOU R Q,et al.Characterizing microbial community and metabolites of Cantonese soy sauce[J].Food Bioscience,2021,40:100872.
[8] 张书泰,周斌,童星,等.酱油酿造过程中微生物多样性分析方法研究进展[J].中国调味品,2019,44(2):193-200.
ZHANG ST,ZHOU B,TONG X,et al.Research progress on analysis methods of microbial diversity in soy sauce brewing process[J].China Condiment,2019,44(2):193-200.
[9] SULAIMAN J,GAN H M,YIN W F,et al.Microbial succession and the functional potential during the fermentation of Chinese soy sauce brine [J].Frontiers in Microbiology,2014,5(556):1-9.
[10] 胡传旺.酱油发酵过程微生物群落解析及功能研究[D].无锡:江南大学,2017.
HU C W.Dynamics of microbial community during soy sauce fermentation and functional analysis strains isolated from moromi[D].Wuxi:Jiangnan University,2017.
[11] LIU X Y,QIAN M,SHEN Y X,et al.An highthroughput sequencing approach to the preliminary analysis of bacterial communities associated with changes in amino acid nitrogen,organic acid and reducing sugar contents during soy sauce fermentation[J].Food Chemistry,2021,349:129131.
[12] YAN Y Z,QIAN Y L,JI F D,et al.Microbial composition during Chinese soy sauce koji-making based on culture dependent and independent methods[J].Food Microbiology,2013,34(1):189-195.
[13] WANG H B,WEI Q Z,GUI S Q,et al.Metagenomic profiling of the bacterial community changes from koji to mash stage in the brewing of soy sauce[J].Polish Journal of Microbiology,2017,66(4):537-541.
[14] 赵述淼,葛向阳.酿造学[M].北京:高等教育出版社,2018:211-252.
ZHAO S M,GE X Y.Zymurgy[M].Beijing:Higher Education Press,2018:211-252.
[15] ZHENG J,WU C D,HUANG J,et al.Spatial distribution of bacterial communities and related biochemical properties in Luzhou-flavor liquor-fermented grains[J].Journal of Food Science,2014,79(12):M2491-M2498.
[16] 张晓娟,陆震鸣,柴丽娟,等.中国传统酿造食品微生物生态学及其研究策略[J].中国科学生命科学,2019,49(5):575-584.
ZHANG X J,LU Z M,CHAI L J,et al.Research strategies for microbial ecology of traditional Chinese fermented foods[J].Scientia Sinica Vitae,2019,49(5):575-584.
[17] KIM B R,SHIN J,GUEVARRA R,et al.Deciphering diversity indices for a better understanding of microbial communities[J].Journal of Microbiology and Biotechnology,2017,27(12):2089-2093.
[18] LOZUPONE C A,HAMADY M,KELLEY S T,et al.Quantitative and qualitative beta diversity measures lead to different insights into factors that structure microbial communities[J].Applied and Environmental Microbiology,2007,73(5):1576-1585.
[19] 万云洋,赵国屏.球类原核微生物[J].微生物学杂志,2017,37(4):82-92.
WAN Y Y,ZHAO G P.Species and genera of microorganisms in spherical/spheroid shapes in prokaryotes[J].Journal of Microbiology,2017,37(4):82-92.
[20] HAMADA M,IINO T,IWAMI T,et al.Mobilicoccus pelagius gen.nov.sp.nov.and Piscicoccus intestinalis gen.nov.sp.nov.two new members of the family Dermatophilaceae,and reclassification of Dermatophilus chelonae(Masters et al.1995)as Austwickia chelonae gen.nov.comb.nov[J].The Journal of General and Applied Microbiology,2010,56(6):427-436.
[21] ZHAO C J,SCHIEBER A,G?NZLE M G.Formation of taste-active amino acids,amino acid derivatives and peptides in food fermentations -A review[J].Food Research International,2016,89(1):39-47.
[22] TANAKA Y,WATANABE J,MOGI Y.Monitoring of the microbial communities involved in the soy sauce manufacturing process by PCR-denaturing gra dient gel electrophoresis[J].Food Microbiology,2012,31(1):100-106.
[23] GUO J,LUO W,FAN J,et al.Co-inoculation of Staphylococcus piscifermentans and salt -tolerant yeasts inhibited biogenic amines formation during soy sauce fermentation[J].Food Research International,2020,137:109436.
[24] DET-UDOM R,PRAKITCHAIWATTANA C,MAHAWANICH T.Autochthonous microbes and their key properties in browning reduction during soy sauce fermentation[J].LWT-Food Science and Technology,2019,111:378-386.
[25] 王松,郭柯宇,唐林,等.发酵肉制品中凝固酶阴性葡萄球菌的应用研究进展[J].食品与发酵工业,2020,46(23):293-300.
WANG S,GUO K Y,TANG L,et al.Research progress on application of coagulase-negative staphylococci in fermented meat products[J].Food and Fermentation Industries,2020,46(23):293-300.
[26] 刘森,李林光,李可,等.中国浓香型白酒窖池窖泥中原核微生物群落的空间异质性[J].食品科学,2013,34(21):221-226.
LIU S,LI L G,LI K,et al.Spatial heterogeneity of prokaryotic microbial communities in luzhou-flavor liquor pit mud[J].Food Science,2013,34(21):221-226.