新疆地区草原面积广阔,畜牧业发达,生活着一些游牧民族,如哈萨克族等,古老的游牧民族孕育了悠久的乳制品制作和食用历史, 一些传统发酵乳制品的制作方式传承至今, 仍是人们生活中不可或缺的一部分[1]。酸马奶,也叫马奶酒,就是其中的一种,是以鲜马奶为原料,经乳酸菌等微生物发酵后产生的一种低酒精酸味乳饮料。 鲜马奶的营养成分接近人乳,容易被消化吸收,发酵过程中乳糖被代谢产生有机酸和乙醇, 其它成分基本保持不变[2]。 酸马奶凭借营养价值高,能有效抑制有害菌等优点改善了人们的身体状况, 对心血管疾病、消化道感染、糖尿病以及精神性疾病等均能产生一定的辅助治疗作用[3]。
乳酸菌是一类能将可利用的碳水化合物代谢生成乳酸的无芽孢、革兰氏阳性细菌,广泛存在于发酵制品中,且很多乳酸菌具有一定的益生作用[4-6]。目前关于发酵制品的乳酸菌菌群结构的研究十分广泛[7-8]。 本研究团队前期基于7 个属的443 个乳酸菌菌种的16S rRNA 设计乳酸菌 (Lactic acid bacteria, LAB)特异性引物,经论证其对于挖掘低丰度的乳酸菌菌种具有优势[9]。 Pacific Biosciences公司研发的单分子实时测序系统 (Pacbio SMRT测序技术)是一种具有长读长、高通量、快速以及免PCR 等优点的第3 代测序技术,现被广泛应用于菌种检测[10-11]。 结合纠错软件,其准确率可达99.9%[12]。
本文运用Pacbio SMRT 测序技术,基于LAB特异性引物对鲜马奶和酸马奶中的乳酸菌菌群结构进行探究, 以期阐明自然发酵状态下乳酸菌在鲜马奶和酸马奶中的菌群特征, 丰富酸马奶乳酸菌菌群研究基础数据, 为乳酸菌的质量控制和品质改良提供理论依据。
1 材料与方法
1.1 试验试剂及仪器
RNA/DNA 保护液, 大连宝生物工程有限公司;OMEGA DNA isolation kit, 美国OMEGA 公司;5×TBE 电泳缓冲液(Tris 碱54 g、硼酸27.5 g、Na2EDTA·2H2O 3.72 g,定容1 L,pH 8.0)、1.0%琼脂糖凝胶 (1.0 g 琼脂糖溶于100 mL 0.5×TBE 缓冲液), 天津基准化学试剂公司;DL2000 DNA Marker, 宝日医生物技术有限公司;KAPA HiFi HotStartReadyMix PCR Kit,KAPA 公 司;PacBio SMRTbellTM Template Prep Kit 1.0、DNA/Polymerase Binding Kit P6 V2、DNA Sequencing Kit 4.0 V2,Life Technologies 公司。
Eppendorf 5810R 台式高速冷冻离心机, 德国Eppendorf 公司;DYY-12 电泳仪, 北京六一仪器厂;UPV 凝胶成像分析系统,北京赛智创业科技有限公司; 微量紫外分光光度计,NanoDrop 公司;ProFlexTMPCR 仪, 美国AppliedBiosystems 公司;Pacifico SMRT RS II 测序平台, 美国Pacific Bioseciences 公司。
1.2 试验方法
1.2.1 样品采集 样品采自新疆伊犁哈萨克地区的昭苏县(东经80°08′~81°30′, 北纬43°09′~43°15′)和阿克陶县(东经73°26′~76°43′,北纬37°41′~39°29′),共19 份样品,其中酸马奶11 份,鲜马奶8 份。同一采样点既采集鲜马奶也采集酸马奶,具体信息见表1。 吸取搅拌均匀的30 mL 鲜马奶或酸马奶至50 mL 无酶无菌采样管中, 加入10 mL DNA 保护液, 混匀后用液氮极速冷冻1 min,置于低温(2~8 ℃)采样箱中运回实验室,-80 ℃保藏备用。
表1 鲜马奶和酸马奶样品信息及乳酸菌的α 多样性
Table 1 The information and alpha diversity of lactic acid bacteria of fresh mare milk and Koumiss
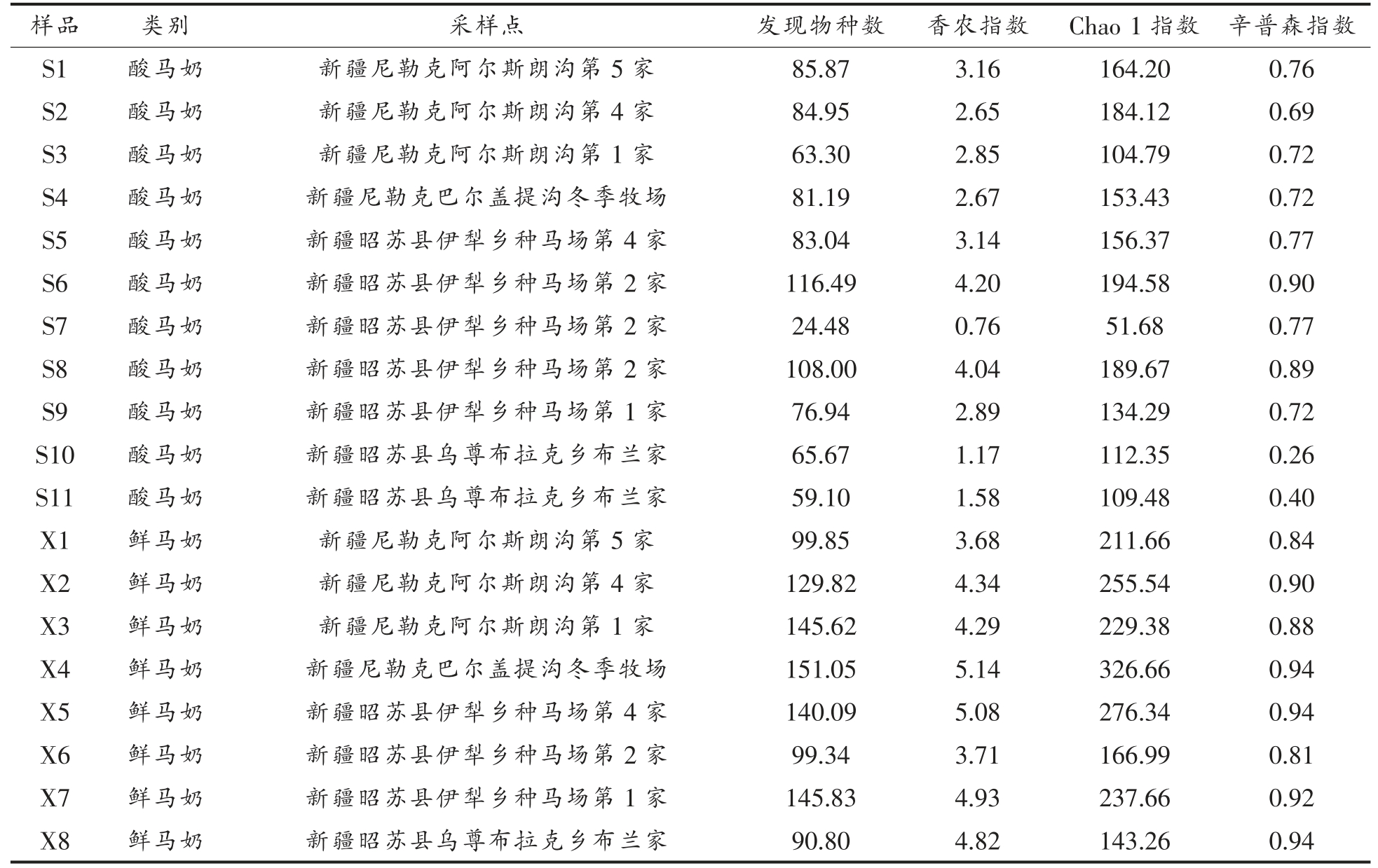
样品 类别 采样点 发现物种数 香农指数 Chao 1 指数 辛普森指数S1 酸马奶 新疆尼勒克阿尔斯朗沟第5 家 85.87 3.16 164.20 0.76 S2 酸马奶 新疆尼勒克阿尔斯朗沟第4 家 84.95 2.65 184.12 0.69 S3 酸马奶 新疆尼勒克阿尔斯朗沟第1 家 63.30 2.85 104.79 0.72 S4 酸马奶 新疆尼勒克巴尔盖提沟冬季牧场 81.19 2.67 153.43 0.72 S5 酸马奶 新疆昭苏县伊犁乡种马场第4 家 83.04 3.14 156.37 0.77 S6 酸马奶 新疆昭苏县伊犁乡种马场第2 家 116.49 4.20 194.58 0.90 S7 酸马奶 新疆昭苏县伊犁乡种马场第2 家 24.48 0.76 51.68 0.77 S8 酸马奶 新疆昭苏县伊犁乡种马场第2 家 108.00 4.04 189.67 0.89 S9 酸马奶 新疆昭苏县伊犁乡种马场第1 家 76.94 2.89 134.29 0.72 S10 酸马奶 新疆昭苏县乌尊布拉克乡布兰家 65.67 1.17 112.35 0.26 S11 酸马奶 新疆昭苏县乌尊布拉克乡布兰家 59.10 1.58 109.48 0.40 X1 鲜马奶 新疆尼勒克阿尔斯朗沟第5 家 99.85 3.68 211.66 0.84 X2 鲜马奶 新疆尼勒克阿尔斯朗沟第4 家 129.82 4.34 255.54 0.90 X3 鲜马奶 新疆尼勒克阿尔斯朗沟第1 家 145.62 4.29 229.38 0.88 X4 鲜马奶 新疆尼勒克巴尔盖提沟冬季牧场 151.05 5.14 326.66 0.94 X5 鲜马奶 新疆昭苏县伊犁乡种马场第4 家 140.09 5.08 276.34 0.94 X6 鲜马奶 新疆昭苏县伊犁乡种马场第2 家 99.34 3.71 166.99 0.81 X7 鲜马奶 新疆昭苏县伊犁乡种马场第1 家 145.83 4.93 237.66 0.92 X8 鲜马奶 新疆昭苏县乌尊布拉克乡布兰家 90.80 4.82 143.26 0.94
1.2.2 宏基因组提取 使用OMEGA DNA isolation kit 试剂盒提取鲜马奶和酸马奶样品中微生物的宏基因组,具体操作参照试剂盒中的说明书。用微量紫外分光光度计检验DNA 的浓度和纯度,保留OD260nm/OD280nm 值在1.8~2.0、质量浓度大于20 ng/μL 的DNA,于-20℃低温保存。
1.2.3 乳酸菌基因组扩增 使用KAPA HiFi HotStartReadyMix PCR Kit,以本实验室设计并评估过的LAB 特异性引物 ( 正向引物15F GCTCAGGAYGAACGCYGG, 反向引物 687R CACCGCTACACATGRADTTC)为扩增引物,对乳酸菌DNA 进行PCR 扩增。 反应体系: 正向引物(10 μmol/L)1.5 μL,反向引物(10 μmol/L)1.5 μL,模板DNA (<100 ng)1.5 μL,KAPA Mix 25 μL,ddH2O 补足至50 μL。 反应程序:95 ℃预变性3 min,98 ℃变性20 s,60 ℃退火15 s,72 ℃延伸30 s,26 个循环;72 ℃末端延伸2 min。用1.0%琼脂糖凝胶电泳结合2 000 bp 的DNA Mark 预估PCR产物的扩增情况及其完整度, 用磁珠法对电泳目标条带清晰的PCR 产物进行纯化, 纯化产物于-20 ℃保藏备用。
1.2.4 Pacbio SMRT 测序 使用Pacific Biosciences SMRTbellTM Template Prep Kit 1.0,对纯化后满足测序量(2 μg)的PCR 产物构建文库,使 用DNA/Polymerase Binding Kit P6 V2、DNA Sequencing Kit 4.0 V2 等试剂上机测序。 建库上机的具体操作参照厂家提供的操作流程。
1.2.5 数据下机及分析 通过Pacific Bioseciences 公司提供的服务平台(smrtanalysis_2.3.0.140936.p5.167094),运用RS_ReadsOfInsert.1 对完成测序的序列进行质控,质控条件:最小循环次数为5 次,最小预测精确度为90,最小插入序列长度为550 bp,最大序列长度为950 bp[9]。将19 份样品的测序结果(原始数据)上传至MG-RAST(项目编号:mgp96186)。
借助QIIME(v1.9.1)平台对质控后的序列进行生物信息学分析。 运用PyNAST 对序列进行比对校准后, 以97%的相似性阈值划分操作分类单元(Operational taxonomic units, OTU),选取一条序列作为该OTU 的代表性序列进行同源性比对,确定其分类学地位。 用于序列同源性比对的数据库 有RDP (Ribosomal database project)、Greengenes(V13.8)和Silva V128。整合3 个数据库的比对结果,挑选属于目标菌属的OTU。 整合目标OTU 的序列后重新进行比对校准、OTU 划分, 分类学地位确定,得到最终注释结果。
结合PyNAST, 使用FastTree 构建OTU 代表性序列的系统发育树,计算各样品的Chao1 指数、辛普森指数(Simpson index)、香农指数(Shannon index)、发现物种数(Observed_species),结合物种含量评估样品的生物多样性。
1.2.6 数据处理 试验数据的统计分析和可视化基于R 语言(v4.0.2)和Origin(v9.7.5.184)。
2 结果与讨论
2.1 鲜马奶和酸马奶中乳酸菌的α 多样性
对19 份样品的乳酸菌基因组的测序结果进行质控后, 运用QIIME 平台进行生物信息学分析。其中,QIIME 平台计算的辛普森指数越大,说明样品的多样性越高。本文测序结果的辛普森指数随序列数目的增加逐渐升高, 最终趋于稳定,到达平台期(见图1),说明测序量满足后续分析要求。
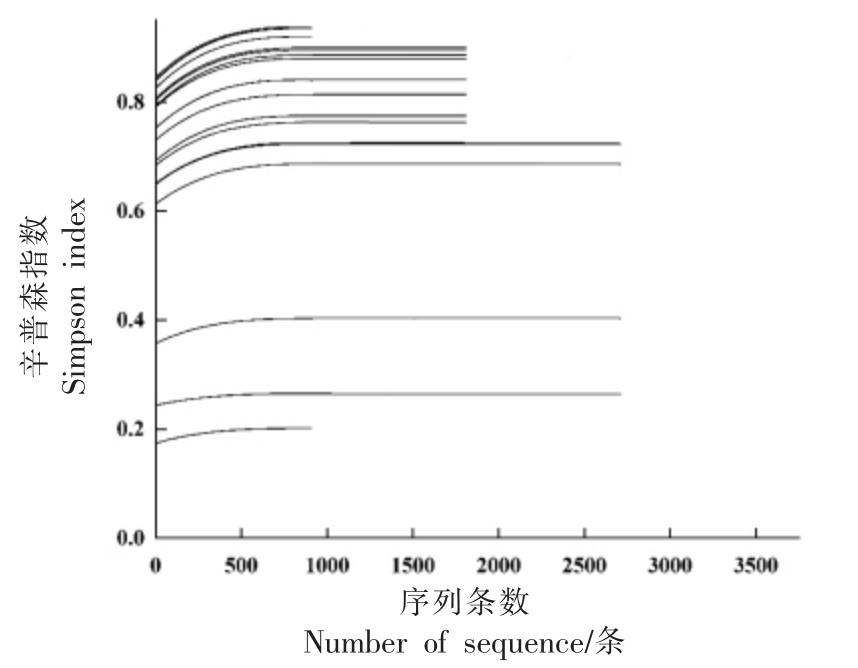
图1 乳酸菌基因测序结果的辛普森指数
Fig.1 Simpson index of lactic acid bacteria gene sequencing results
以样品类别为分组依据,对α 多样性指数进行mann-whitney 显著性检验,结果显示:鲜马奶样品的物种丰度和丰富度显著(P<0.005)高于酸马奶样品,与张猛等[13]对鲜马奶和酸马奶细菌多样性的分析结果一致。
2.2 鲜马奶和酸马奶中乳酸菌菌群结构
基于OTU 注释结果对样品的乳酸菌菌群组成进行统计分析。在属水平上,19 个样品注释到7个含量大于0.1%的乳酸菌菌属,在样品中的分布见图2。 另外, 酸马奶以乳杆菌属(Lactobacillus,84.87%)、乳球菌属(Lactococcus,9.81%)及链球菌属(Streptococcus,5.10%)为优势属;鲜马奶以乳杆菌属(Lactobacillus,48.38%)、 链球菌属(Streptococcus,21.07%)、 葡萄球菌属(Staphylococcus,12.77%)、乳球菌属(Lactococcus,10.04%)、肠球菌属(Enterococcus,5.35%)及魏斯氏菌属(Weissella,1.27%)为优势属。
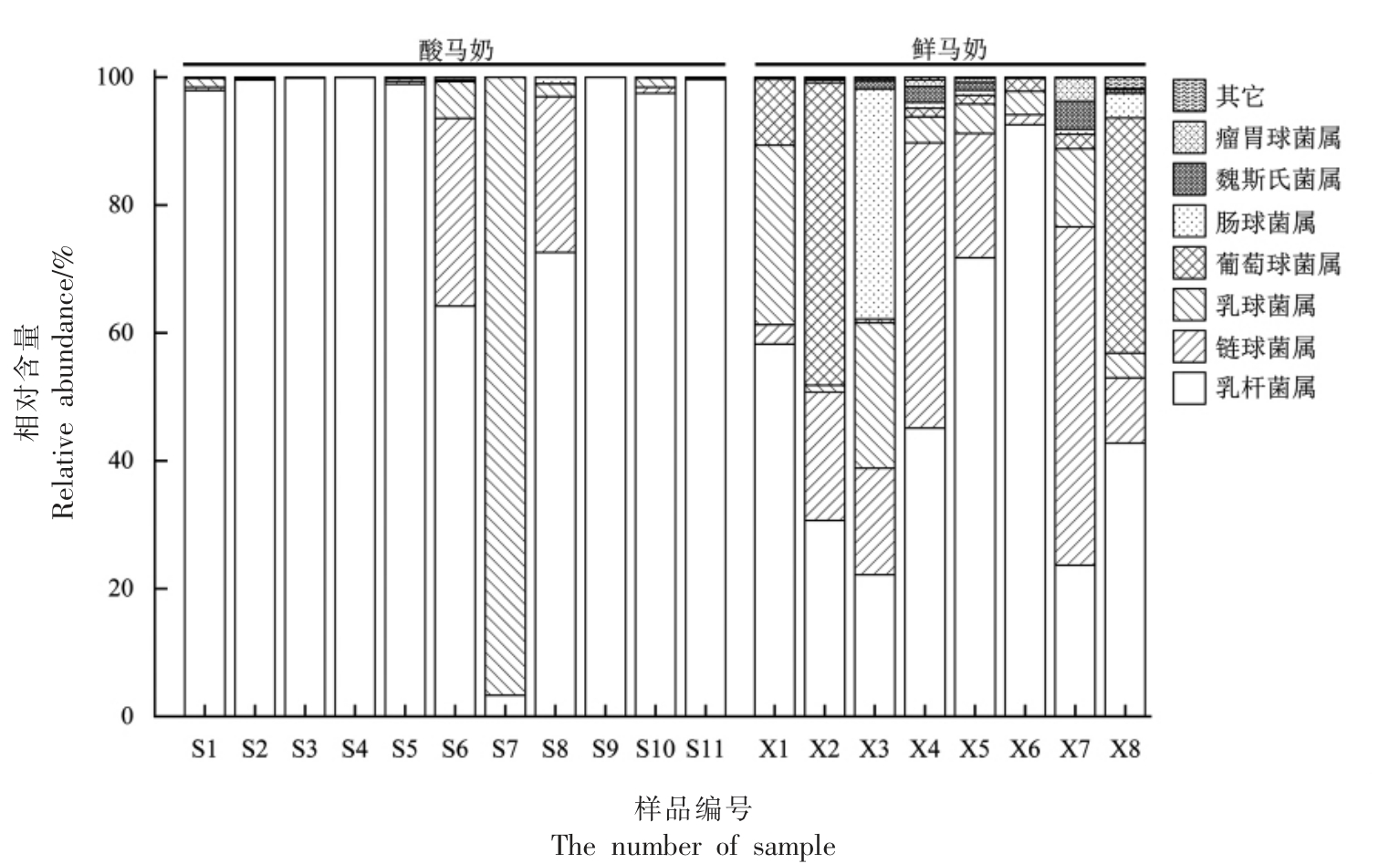
图2 酸马奶和鲜马奶中乳酸菌属水平上的菌群组成
Fig.2 The composition of lactic acid bacteria in Koumiss and fresh mare milk in the genus level
在种水平上,19 个样品共注释到109 个乳酸菌菌种,其中,相对含量大于1%的乳酸菌菌种在各样品中的分布如图3 所示。 在酸马奶中共注释到52 个乳酸菌菌种, 其中, 瑞士乳杆菌(Lactobacillus helveticus,61.77%)、 马乳酒样乳杆菌(Lactobacillus kefiranofaciens,14.91%)、乳酸乳球菌(Lactococcus lactis,9.76%)、德氏乳杆菌(Lactobacillus delbrueckii,4.13%)、嗜热链球菌(Streptococcus thermophilus,3.76%)、 高加索酸奶乳杆菌(Lactobacillus kefiri,2.15%) 及桥乳杆菌(Lactobacillus pontis,1.06%) 等7 个菌种为酸马奶的优势乳酸菌菌种。 在鲜马奶中共注释到103 个乳酸菌菌种,其中,瑞士乳杆菌(35.52%)、唾液链球菌(Streptococcus salivarius,10.11%)、 乳酸乳球菌(9.49%)、嗜热链球菌(5.96%)、德氏乳杆菌(5.37%)、粪肠球菌(Enterococcus faecalis,4.8%)、腐生葡萄球菌(Staphylococcus saprophyticus,4.45%)、金黄色葡萄球菌(Staphylococcus aureus,2.62%)、瘤胃乳杆菌(Lactobacillus ruminis,2.40%)、松鼠葡萄球菌(Staphylococcus sciuri,2.05%)、琥珀葡萄球菌(Staphylococcus succinus,1.8%)、 唾液乳杆菌(Lactobacillus salivarius,1.2%) 及融合魏斯氏菌(Weissella confusa,1.1%) 等13 个菌种为鲜马奶的优势乳酸菌菌种。
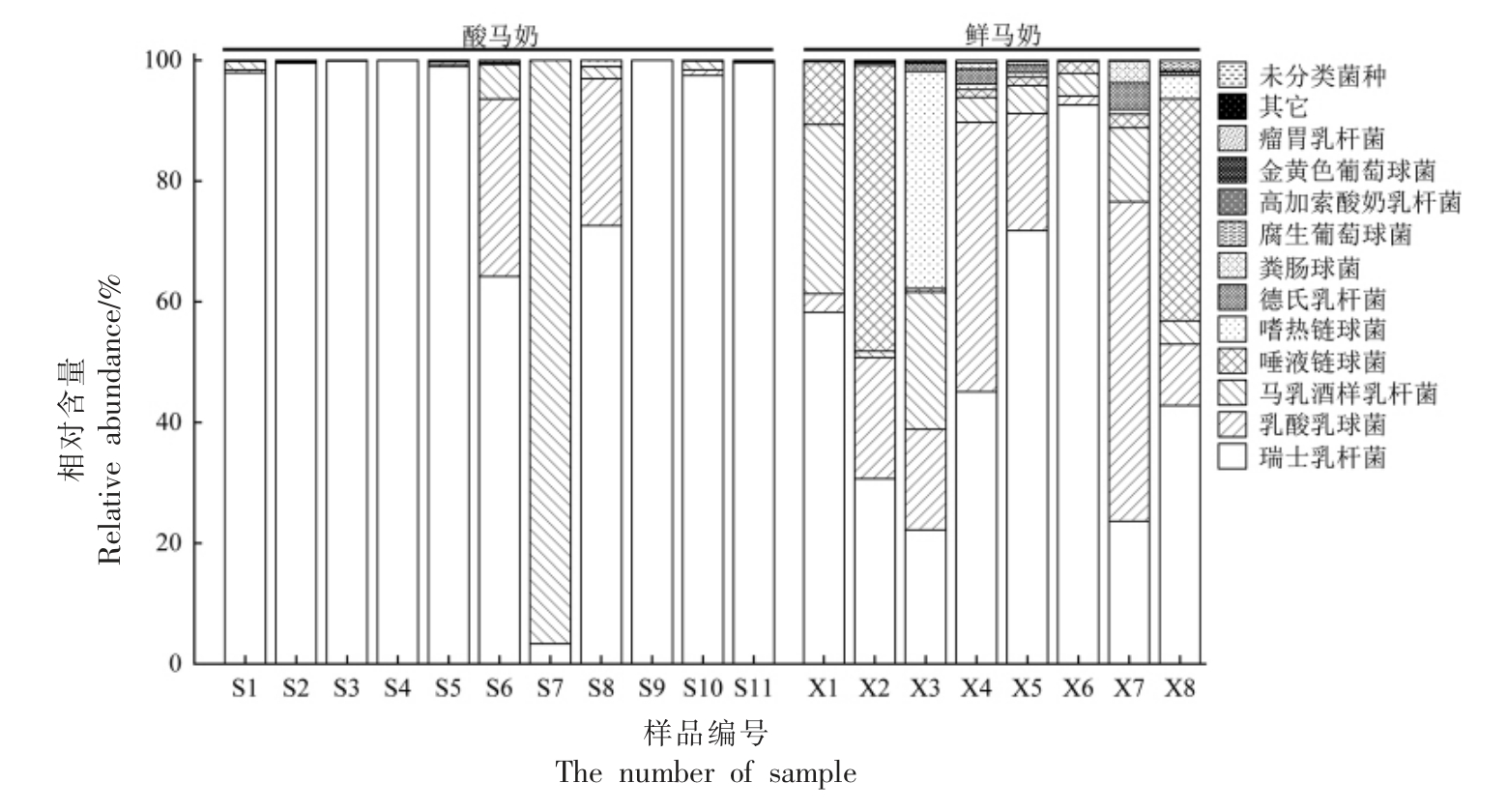
图3 酸马奶和鲜马奶中乳酸菌种水平上的菌群组成
Fig.3 The composition of lactic acid bacteria in Koumiss and fresh mare milk in species level
可以看出,鲜马奶乳酸菌菌群十分丰富,除了一些常见于发酵制品的乳酸菌菌种外, 还发现一些具有致病性的菌种,如腐生葡萄球菌、金黄色葡萄球菌等容易引起食物中毒的菌种, 以及轻链球菌 (Streptococcus mitis)[14]、 无乳链球菌(Streptococcus agalactiae)[15]、 解没食子酸链球菌(Streptococcus gallolyticus)[16]、 肺炎链球菌(Streptococcus pasteurianus)[17]和巴黎链球菌(Streptococcus_lutetiensis)[18]等容易引起各类炎症反应的菌种。 这可能是由于开放的挤奶环境以及未杀菌处理的传统发酵工艺造成的。 然而,随着鲜马奶的持续发酵,pH 3.8 的酸性环境使大部分致病菌无法生存[19],提高了酸马奶的饮用安全性。
相比于鲜马奶, 酸马奶具有更加简单的乳酸菌菌种组成,这与α 多样性的检验结果一致。其原因可能是酸马奶低温、 微氧且代谢产生乙醇的发酵环境[20]抑制了部分菌种的生长繁殖;同时为部分乳酸菌菌种提供了生长条件, 例如在富氮环境中,酿酒酵母通过分泌代谢产物(尤其是氨基酸)来调节其代谢, 从而使Lactococcus lactis 和植物乳杆菌(Lactobacillus plantarum)得以生存[21]。
用Pacbio SMRT 测序技术检测细菌菌种,莫兰馨[22]发现内蒙古锡林郭勒盟的酸马奶以副乳房链球菌(Streptococcus parauberis,62.82%)、瑞士乳杆菌(27.12%)为优势菌种,马乳酒样乳杆菌、高加索酸奶乳杆菌、 德氏乳杆菌等菌种只在个别样品中检出。 刘文俊等[23]发现蒙古国酸马奶以瑞士乳杆菌(94.64%)和乳酸乳球菌(1.70%)为优势菌种,而马乳酒样乳杆菌、 高加索酸奶乳杆菌等菌种丰度很低。 以上两组结果是基于16S rRNA 全长的细菌菌群结构的分析。 结合本文酸马奶乳酸菌菌群结构,可以发现酸马奶中的细菌以乳酸菌为主,不同地区的酸马奶中的乳酸菌菌群结构存在差异, 而瑞士乳杆菌在不同地区的酸马奶中均具优势,可能是因为其适应酵母发酵环境[24],且大量瑞士乳杆菌菌株被研究证实具有益生特性[25]。
基于乳酸菌菌种的相对含量做相关性(Spearman 相关系数)分析,部分结果见图4。 相对含量大于0.1%的乳酸菌菌种中,瑞士乳杆菌与桥乳杆菌显著正相关(r=0.582,P<0.05),与副猪链球菌 (Streptococcus parasuis,r=-0.503)、 轻链球菌(r =-0.542) 以及副血链球菌(Streptococcus parasanguinis,r=-0.685)等显著(P<0.05)负相关。瑞士乳杆菌与桥乳杆菌在酸马奶中的含量高,且共同出现在其它含有酵母的发酵制品中[26],两者在含有酵母的发酵环境中能很好地生长。 另外,Lactobacillus helveticus 可产生抑制生物膜形成的生物表面活性物质[27],这可能是其与副猪链球菌、轻链球菌以及副血链球菌等呈负相关的原因之一。

图4 鲜马奶和酸马奶样品中乳酸菌菌种相关性热图
Fig.4 Heatmap of correlation of lactic acid bacteria species in fresh mare milk and Koumiss
2.3 鲜马奶和酸马奶中乳酸菌的β 多样性
11 个酸马奶和8 个鲜马奶样品中OTU 分布统计结果(图5)显示,只有9 个OTU 是19 个样品共有的,有299 个OTU 是各样品特有的,然而,共有OTU 所包含的序列数目(20 284 条)是总序列数目的47.1%,其注释结果显示均为瑞士乳杆菌。
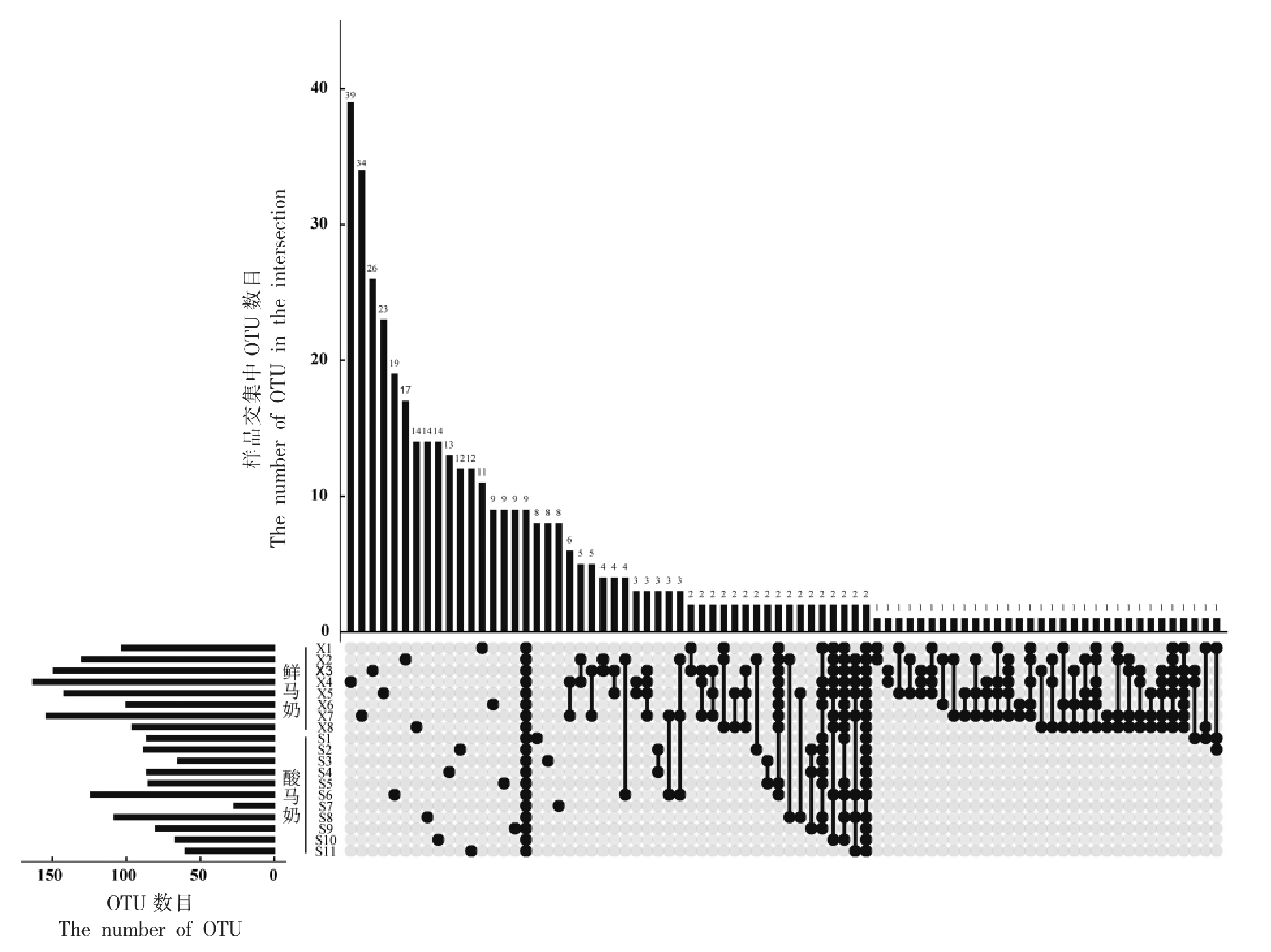
图5 鲜马奶和酸马奶中OTU 分布图
Fig.5 Distribution of OTU in fresh mare milk and Koumiss
注:柱状图中每个柱子代表一个OTU 集合,以集合中包含的OTU 数目排序展示。 柱子下的黑点表示该集合中的OTU 所属样品。左侧条形图的长度代表样品中OTU 数目。
根据OTU 在鲜马奶和酸马奶样品中的分布情况, 统计仅存于鲜马奶样品中的OTU 有244个,注释到85 个乳酸菌菌种,其中瑞士乳杆菌、唾液链球菌、 瘤胃乳杆菌和嗜热链球菌4 个乳酸菌菌种的出现频率较高, 其余大量存在的乳酸菌菌种出现频率较低。 仅存于酸马奶样品中的OTU 数目比鲜马奶的少,共126 个,且注释结果基本为发酵乳制品中常见的乳酸菌菌种——瑞士乳杆菌、马乳酒样乳杆菌、 高加索酸奶乳杆菌、 德氏乳杆菌、桥乳杆菌、乳酸乳球菌以及嗜热链球菌等。
基于以上分析结果,不难看出,鲜马奶中乳酸菌菌种的丰富度、 丰度以及物种组成存在明显的差异。基于系统进化关系,计算各样品间的unifrac距离,对样品的β 多样性进行分析,以加权unifrac距离进行主坐标分析(图6),以加权和非加权unifrac 距离统计鲜马奶组和酸马奶组内样品的离散情况及组间差异(图7)。
从图6 可以看出鲜马奶和酸马奶样品被很好地区分。 其中,酸马奶样品S7 与其它样品的距离较远,S6 和S8 与部分鲜马奶样品较近。 鲜马奶样品X6 与部分酸马奶样品较近。这与乳酸菌菌群组成的分析结果一致,酸马奶样品S7 中乳酸乳球菌含量极高(96.61%),是19 个样品平均值的10 倍;S6 和S8 的乳酸菌菌群结构更接近鲜马奶样品;鲜马奶样品X6 的乳酸菌菌群结构更接近酸马奶样品。
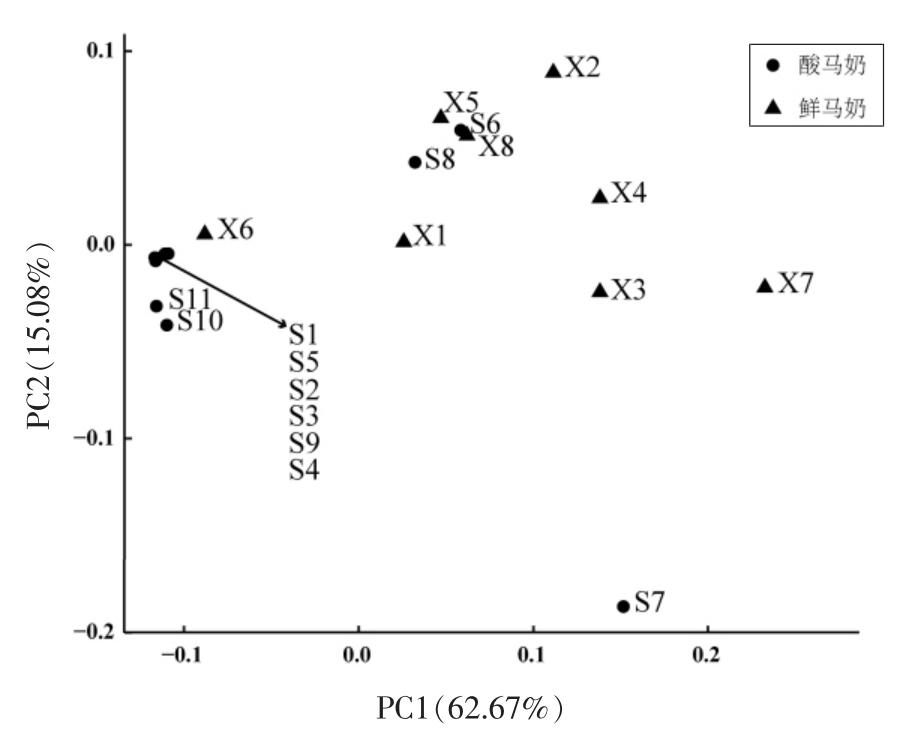
图6 鲜马奶和酸马奶样品的主坐标分析(PCoA)
Fig.6 Principal coordinate analysis (PCoA)of fresh mare milk and Koumiss
wilcoxon 秩和检验结果显示,酸马奶样品的组内非加权unifrac 距离显著(P<0.005,图7a)高于鲜马奶样品, 说明酸马奶中乳酸菌菌种组成差异大。考虑乳酸菌菌种含量后,两组样品间的组内加权unifrac 距离的P 值为0.051(图7b),鲜马奶具有更复杂的乳酸菌菌群结构, 这与上述α 多样性和菌群结构的分析结果一致。以上结果说明,自然发酵的酸马奶品质参差不齐。
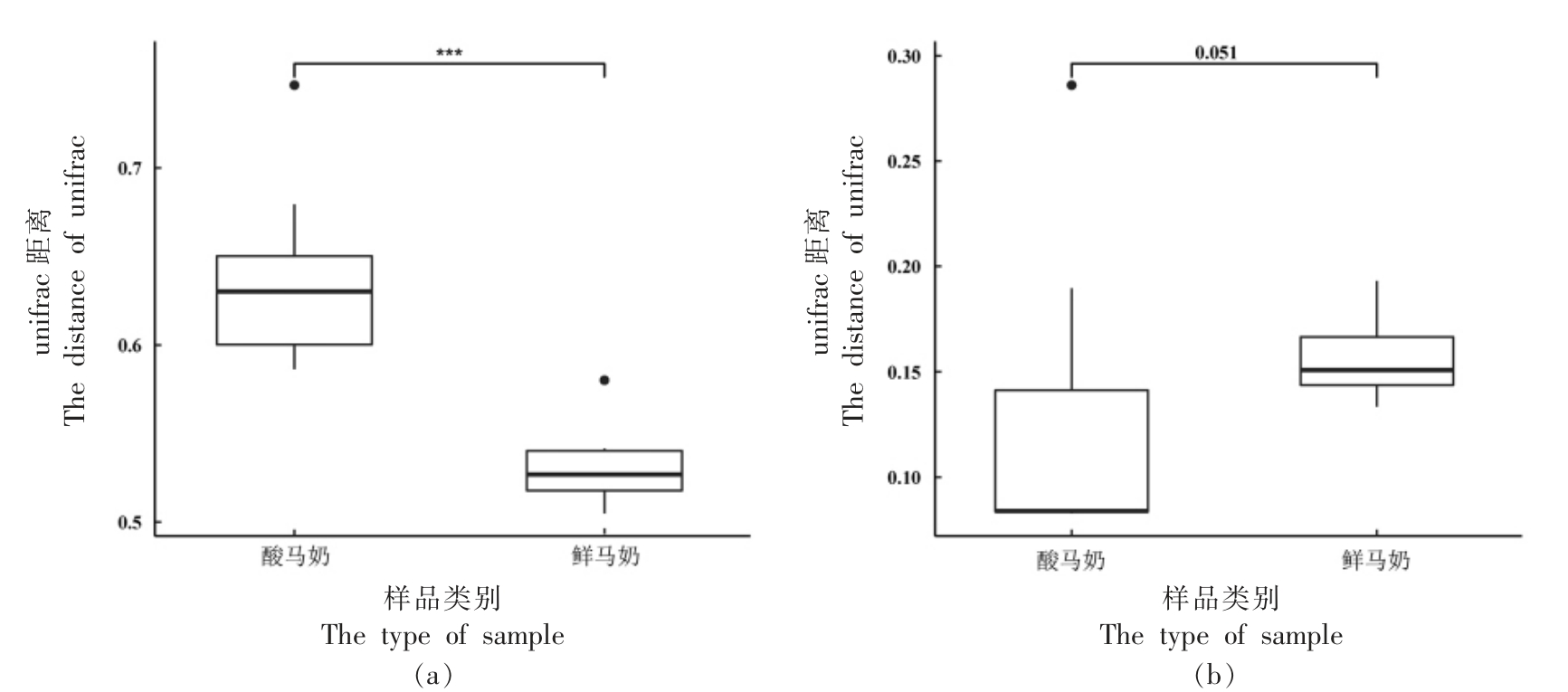
图7 鲜马奶和酸马奶样品间unifrac 距离
Fig.7 The unifrac distance of fresh mare milk and Koumiss
3 结论
应用Pacbio SMRT 测序技术,基于LAB 特异性引物对新疆伊犁哈萨克地区昭苏县和阿克陶县的鲜马奶和酸马奶样品中的乳酸菌多样性进行研究,结果显示,鲜马奶样品中乳酸菌的α 多样性显著高于酸马奶, 这是因为开放的挤奶环境和不杀菌的传统发酵工艺使鲜马奶中的乳酸菌十分丰富,而酸马奶发酵过程中,低温、微氧且代谢产生乙醇的发酵条件抑制了部分乳酸菌菌种的生长繁殖,使其乳酸菌菌群的丰富度降低。鲜马奶中还检测到一些具有致病性的菌种,不适合直接饮用,而酸马奶不存在这种情况。 在酸马奶中检测到瑞士乳杆菌等一些具有益生潜力的乳酸菌菌种, 这可能也是酸马奶具有强大益生功能的原因之一。 酸马奶样品的组内非加权unifrac 距离显著高于鲜马奶,说明酸马奶品质参差不齐。鲜马奶与酸马奶中乳酸菌多样性的分析研究结果, 对酸马奶的品质改良和质量控制以及工业化生产提供参考。
[1] 李星科, 李开雄, 邹圣东. 酸马奶酒[J]. 中国乳业,2006(7): 58-60.
LI X K, LI K X, ZOU S D. Koumiss[J]. China Dairy, 2006(7): 58-60.
[2] 孙天松, 孟和毕力格, 王俊国, 等. 新疆维吾尔自治区酸马奶化学组成分与微生物学分析[J]. 中国乳品工业, 2005(10): 10-14.
SUN T S, MENGHE B L G, WANG J G, et al.Analysis of chemical composition and microorganism flora of traditionally home-made Koumiss in Xinjiang[J]. China Dairy Industry, 2005(10): 10-14.
[3] 哈斯苏荣, 阿木古楞, 芒来. 酸马奶及其医学价值[J]. 中国中药杂志, 2003(1): 15-18.
HA S S R, A M G L, MANG L. The Koumiss and its medical value[J]. China Journal of Chinese Materia Medica, 2003(1): 15-18.
[4] O'CALLAGHAN J, W. O'TOOLE P. Lactobacillus:Host-microbe relationships[J]. Curr Top Microbiol Immunol, 2013, 358(1): 119-154.
[5] GHOSH T, BENIWAL A, SEMWAL A, et al.Mechanistic insights into probiotic properties of lactic acid bacteria associated with ethnic fermented dairy products[J]. Frontiers in Microbiology, 2019, 10(1):502-502.
[6] HUANG Y, WANG X J, WANG J F, et al. Lactobacillus plantarum strains as potential probiotic cultures with cholesterol-lowering activity[J]. Journal of Dairy Science, 2013, 96(5): 2746-2753.
[7] 张哲, 刘小鸣, 陈卫. 内蒙古传统发酵驼乳中乳酸菌和酵母菌的分离鉴定及其生物多样性分析[J]. 中国食品学报, 2018, 18(7): 230-238.
ZGANG Z, LIU X M, CHEN W. Isolation and identification of lactic acid bacteria and yeast and biodiversity analysis of traditional fermented camel milk in Inner Mongolia[J]. Journal of Chinese Institute of Food Science and Technology, 2018, 18(7): 230-238.
[8] 尚雪娇, 代程洋, 王玉荣, 等. 腌菜中细菌多样性研究及乳酸菌分离鉴定[J]. 中国酿造, 2018, 37(11): 40-44.
SHANG X J, DAI C Y, WANG Y R, et al. Bacterial diversity of bacteria and isolation and identification of lactic acid bacteria from pickles[J]. China Brewing, 2018, 37(11): 40-44.
[9] HOU Q C, BAI X Y, LI W C, et al. Design of primers for evaluation of lactic acid bacteria populations in complex biological samples[J]. Front Microbiol, 2018, 9(1): 2045-2045.
[10] 边燕飞, 席晓霞, 郑艺, 等. 基于PacBio SMRT 技术的婴幼儿配方奶粉微生物安全性评价[J]. 中国食品学报, 2019, 19(10): 212-220.
BIAN Y F, XI X X, ZHENG Y, et al. Evaluation of microbial safety in infant formula based on PacBio SMRT sequencing[J]. Journal of Chinese Institute of Food Science and Technology, 2019, 19(10): 212-220.
[11] 舒明月, 付爱思, 张洁, 等. 基于PacBio SMRT 测序技术评估妊娠期糖尿病人的肠道菌群[J]. 微生物学报, 2019, 59(9): 1823-1839.
SHU M Y, FU A S, ZHANG J, et al. Pacbio SMRT sequencing of intestinal flora in patients with gestational diabetes mellitus[J]. Acta Microbiologica Sinica, 2019, 59(9): 1823-1839.
[12] CHUL S S, HWAN A D, JIN K S, et al. Advantages of single-molecule real-time sequencing in high-GC content genomes[J]. Plos One, 2013, 8(7):e68824.
[13] 张猛, 党娜, 孟和毕力格, 等. 基于Pac-Bio 单分子实时测序技术比较酸马奶和生马奶中细菌多样性[C]//第十五届益生菌与健康国际研讨会摘要集. 南京: 中国食品科学技术学会, 2020.
ZHANG M, DANG N, MENGHE B L G, et al.Comparison of bacterial diversity in yogurt and raw horse milk based on PacBio single molecule realtime sequencing[C]// Proceedings of the Fifteen International Symposium on Probiotics and Health.Nanjing: China Society of Food Science and Technology, 2020.
[14] MITCHELL J. Streptococcus mitis: Walking the line between commensalism and pathogenesis[J]. Molecular Oral Microbiology, 2011, 26(2): 89-98.
[15] ROSINI R, MARGARIT I. Biofilm formation by Streptococcus agalactiae: Influence of environmental conditions and implicated virulence factors[J]. Frontiers in Cellular and Infection Microbiology, 2015,5(6): 6-6.
[16] ISENRING J, KOHLER J, NAKATA M, et al.Streptococcus gallolyticus subsp. gallolyticus endocarditis isolate interferes with coagulation and activates the contact system[J]. Virulence, 2018, 9(1):248-261.
[17] KLATTE J M, CLARRIDGE III J E, BRATCHER D, et al. A longitudinal case series description of meningitis due to Streptococcus gallolyticus subsp.pasteurianus in infants[J]. J Clin Microbiol, 2012,50(1): 57-60.
[18] CHONGPRASERTPON N, CUSACK R, COUGHLAN J J, et al. Streptococcus Bovis endocarditis post colonic polypectomy [J]. European Journal of Case Reports in Internal Medicine, 2019, 6(5):001110.
[19] QIMU G, ZHENG Y, XI X X, et al. Investigating bacterial population structure and dynamics in traditional Koumiss from Inner Mongolia using single molecule real-time sequencing[J]. Journal of Dairy Science, 2016, 99(10): 7852-7863.
[20] 孙天松. 中国新疆地区传统发酵酸马奶的化学组成及乳酸菌生物多样性研究[D]. 呼和浩特: 内蒙古农业大学, 2006.
SUN T S. Chemical composition and biodiversity of lactic acid bacteria of Koumiss - A traditional fermented mare milk product in Xinjiang of China[D].Hohhot: Inner Mongolia Agricultural University,2006.
[21] PONOMAROVA O, GABRIELLI N, SEVIN D C,et al. Yeast creates a niche for symbiotic lactic acid bacteria through nitrogen overflow[J]. Cell Systems,2017, 5(4): 345-357.
[22] 莫蓝馨. 内蒙古锡林郭勒盟和蒙古国巴彦洪格尔省传统发酵乳制品中细菌多样性研究[D]. 呼和浩特:内蒙古农业大学, 2019.
MO L X. Investigation of bacterial diversity in traditional fermented dairy products from Xilingol of Inner Mongolia and Bayankhonger of Mongolia[D]. Hohhot: Inner Mongolia Agricultural University, 2019.
[23] 刘文俊, 多拉娜, 刘亚华, 等. 基于纯培养方法和PacBio 三代测序技术研究蒙古国传统酸马奶中乳酸菌多样性[J]. 中国食品学报, 2019, 19(4): 27-37.
LIU W J, DUOLANA, LIU Y H, et al. Lactic acid bacteria diversity in Mongolia traditional Koumiss based on culture-dependent and PacBio sequencing technology[J]. Journal of Chinese Institute of Food Science and Technology, 2019, 19(4): 27-37.
[24] VIIARD E, MIHHALEVSKI A, RUHKA T, et al.Evaluation of the microbial community in industrial rye sourdough upon continuous back-slopping propagation revealed Lactobacillus helveticus as the dominant species [J]. Journal of Applied Microbiology,2013, 114(2): 404-412.
[25] TAVERNITI V, GUGLIELMETTI S. Health-promoting properties of Lactobacillus helveticus[J]. Frontiers in Microbiology, 2012, 3(1): 392-392.
[26] VOGELMANN S A, SEITTER M, SINGER U, et al. Adaptability of lactic acid bacteria and yeasts to sourdoughs prepared from cereals, pseudocereals and cassava and use of competitive strains as starters[J].International Journal of Food Microbiology, 2009,130(3): 205-212.
[27] JIANG X, YAN X, GU S, et al. Biosurfactants of Lactobacillus helveticus for biodiversity inhibit the biofilm formation of Staphylococcus aureus and cell invasion[J]. Future Microbiology, 2019, 14 (13):1133-1146.