瑞士乳杆菌(Lactobacillus helveticus,L.helveticus)是一种专性同型发酵乳酸菌(Lactic acid bacteria,LAB),因高蛋白水解活性而被广泛用于奶酪、发酵乳饮料等的生产,是开发功能性发酵乳制品的优良菌种[1]。瑞士乳杆菌本身具有一定益生特性,其发酵生产的乳制品含有丰富的生物活性肽,在提高产品营养价值的同时赋予产品缓解高血压、调节免疫反应等益生功能[2]。
随着第2 代高通量测序技术的发展,测序所需的物质和时间成本大大降低,使得大规模微生物基因组测序成为可能[3]。比较基因组学(Comparative genomics) 以不同物种或同一物种的不同个体的全基因组序列相似性为基础,预测基因数量、功能及其表达机制[4]。通过比较基因组学可识别物种的保守基因和差异基因,进而讨论其遗传背景和表型演化[3,5],而全基因组完成图可提供更全面的基因组信息,有利于准确比较基因组分析[6]。瑞士乳杆菌作为重要的发酵剂菌种,利用比较基因组学方法解析其基因组信息,对开发其工业生产潜力至关重要。
瑞士乳杆菌H9 和H10 是本团队分别从中国西藏发酵牦牛乳和传统发酵乳中分离的瑞士乳杆菌菌株。瑞士乳杆菌H9 蛋白水解活性高,其发酵乳制品中含有VPP、IPP 等生物活性肽,而瑞士乳杆菌H10 同样具有高蛋白水解活性,然而其发酵乳不产生上述寡肽[7]。本团队已完成对瑞士乳杆菌H9 和H10 的全基因组测序[8-9]。本研究以瑞士乳杆菌H9 和H10 基因组及NCBI 数据库中其它16株瑞士乳杆菌基因组完成图为研究对象,利用比较基因组学方法从基因组层面探究不同瑞士乳杆菌菌株间的遗传差异,为其进一步开发应用奠定遗传学基础。
1 材料与方法
1.1 瑞士乳杆菌基因组序列
本研究所用18 株瑞士乳杆菌全基因组完成图均来自NCBI 数据库(https://www.ncbi.nlm.nih.gov/genome/?term=lactobacillus%20helveticus),详情见表1。
1.2 平均核苷酸一致性(Average nucleotide identity,ANI)的计算
本研究参考Goris 等[10]报道的方法,计算18株瑞士乳杆菌基因组序列的ANI 值。
1.3 基因预测与注释
利用Prokka(v1.14)[11]软件进行基因预测,获得基因功能注释信息。
1.4 泛-核心基因集的构建(Pan-core genome)
采用Roary(v3.13.0)[12]软件以氨基酸序列相似性大于90%的标准识别核心基因集(Core genes)和特有基因,其中核心基因为所有菌株共有基因,特有基因指只存在于1 个菌株的基因。
1.5 系统发育树的构建
基于核心基因核苷酸序列,采用FastTree(2.1.3)[13]软件通过最大似然法(Maximum Likelihood,ML)构建系统发育树,使用iTol 软件(http://https://itol.embl.de/)对系统发育树进行展示。
1.6 共线性分析
使用 Mauve (v20150226)软件[14]对瑞士乳杆菌H9、瑞士乳杆菌H10 和瑞士乳杆菌DPC4571 的基因组序列进行共线性分析。
1.7 数据作图
使用R(V3.5.0)软件中的“pheatmap”程序包绘制热图。
2 结果和分析
2.1 瑞士乳杆菌H9 和H10 基因组基本特征
通过Prokka 软件注释获得瑞士乳杆菌H9 和H10 及其它瑞士乳杆菌菌株基因组特征 (表1)。瑞士乳杆菌H9 基因组大小1.87 Mb,不含质粒,GC(%)含量为37.02%,预测到2 015 个编码序列(Coding sequence,CDS),12 个rRNA 和61 个tRNA(表1);瑞士乳杆菌H10 基因组由1 条染色体和1 个质粒组成,全基因组大小为2.17 Mb,其中环状染色体大小为2.15 Mb,GC(%)含量为36.79%,预测到2 237 个CDS,63 个tRNA。
乳酸菌在适应其富营养生存环境过程中,缺失多种合成代谢途径,其基因组趋于缩小[15],瑞士乳杆菌基因组也正经历衰减[16]。所以推测瑞士乳杆菌H9 基因组较小可能与其对乳环境的适应性有关。
2.2 系统发育分析
构建系统发育分析是研究种群结构和物种进化的重要方法,系统发育树可直观地表现同一群体不同个体间的遗传距离与进化关系[17]。为了解瑞士乳杆菌H9 和H10 与其它瑞士乳杆菌菌株之间的遗传进化关系,本研究以近源物种母鸡乳杆菌(Lactobacillus gallinarum)HFD4 作为外群,基于核心基因的核苷酸序列,通过最大似然法(Maximum Likelihood,ML)构建系统发育树。
结果显示(图1),系统发育树可分为4 个分支,瑞士乳杆菌H9 在距离外群菌株最远的分支IV,该分支内的全部7 个菌株均为发酵乳制品分离株,其中包括工业发酵剂菌株瑞士乳杆菌CNRZ32;瑞士乳杆菌H10 和CUAH18 聚类于分支II,该分支离外群菌株最近;4 个分支中人类肠道/粪便分离株和发酵乳制品分离株呈混合聚类,如除瑞士乳杆菌H10、CUAH18 和R0052 外,其它乳制品分离株均聚类于分支III 和分支IV,而3 株俄罗斯人类肠道分离株聚类于分支III 中的一个亚分支,发酵乳制品分离株ROO52 与2 株韩国人类粪便分离株聚类于分支I。王晓伟[18]基于245 株瑞士乳杆菌11 个持家基因序列型利用邻接法构建系统发育树发现,其聚类结果与菌株分离地最小生成树一致。关于分离地与分离源对瑞士乳杆菌进化的影响有待进一步研究。
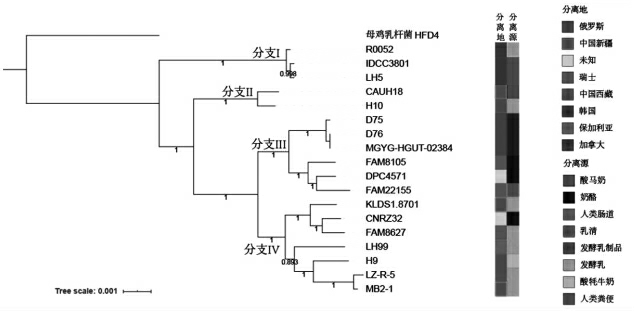
图1 基于核心基因序列的系统发育树
Fig.1 Phylogenetic tree based on core gene sequences
2.3 平均核苷酸一致性(Average nucleotide identity,ANI)分析
平均核苷酸一致性(Average nucleotide identity,ANI)是用以评估遗传多样性的常用方法。该方法通过对基因组进行同源序列比对来判断菌株是否为同一个种或亚种[19]。在比较基因组学分析中,ANI 可评估基因组间的多态性和相似性,一般认为ANI 大于95%为同一个种[20]。Zhong 等[21]通过计算两两序列之间的ANI 值发现161 株粪肠球菌不同菌株之间存在遗传差异。本研究计算18 株瑞士乳杆菌基因组完成图(表1)两两之间ANI 值,并绘制热图。
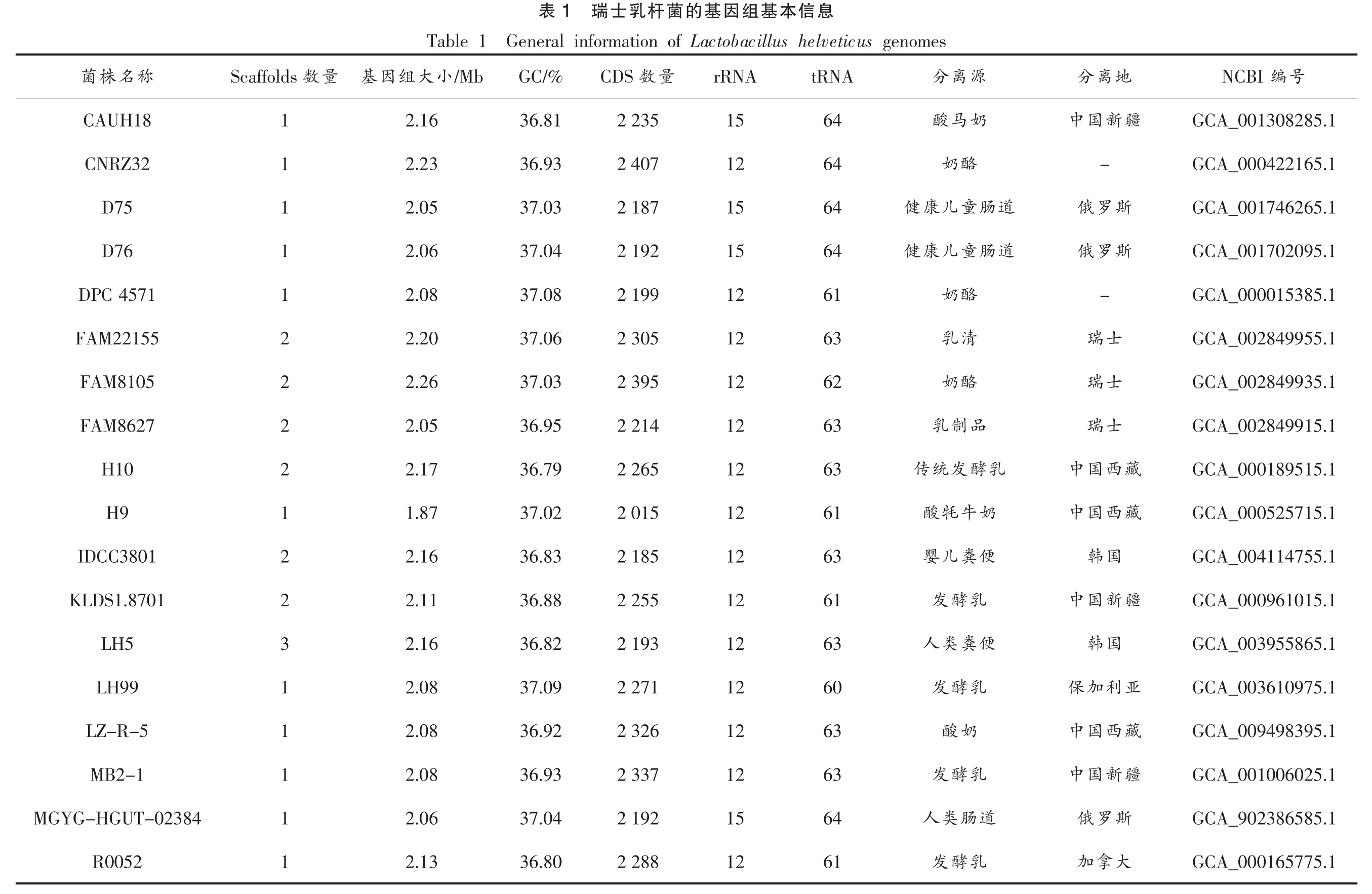
号编NCBI地离分GCA_001308285.1疆新国中GCA_000422165.1-GCA_001746265.1斯罗俄GCA_001702095.1斯罗俄GCA_000015385.1-GCA_002849955.1士瑞GCA_002849935.1士瑞GCA_002849915.1士瑞GCA_000189515.1藏西国中GCA_000525715.1藏西国中GCA_004114755.1国韩GCA_000961015.1疆新国中GCA_003955865.1国韩GCA_003610975.1亚利加保GCA_009498395.1藏西国中GCA_001006025.1疆新国中GCA_902386585.1斯罗俄GCA_000165775.1大拿加息信本基组因基的菌杆乳士瑞1表General information of Lactobacillus helveticus genomes源离分tRNA rRNA量数CDS GC/%奶马酸64 15 2 235 36.81酪奶64 12 2 407 36.93道肠童儿康健64 15 2 187 37.03道肠童儿康健64 15 2 192 37.04酪奶61 12 2 199 37.08清乳63 12 2 305 37.06酪奶62 12 2 395 37.03品制乳63 12 2 214 36.95乳酵发统传63 12 2 265 36.79奶牛牦酸61 12 2 015 37.02便粪儿婴63 12 2 185 36.83乳酵发61 12 2 255 36.88便粪类人63 12 2 193 36.82乳酵发60 12 2 271 37.09奶酸63 12 2 326 36.92乳酵发63 12 2 337 36.93道肠类人64 15 2 192 37.04乳酵发61 12 2 288 36.80 Table 1 /Mb小大组2.16 2.23 2.05 2.06 2.08 2.20 2.26 2.05 2.17 1.87 2.16 2.11 2.16 2.08 2.08 2.08 2.06 2.13因基量数Scaffolds称名株菌1 CAUH18 1 CNRZ32 1 D75 1 D76 1 DPC 4571 2 FAM22155 2 FAM8105 2 FAM8627 2 H10 1 H9 2 IDCC3801 2 KLDS1.8701 3 LH5 1 LH99 1 LZ-R-5 1 MB2-1 1 MGYG-HGUT-02384 1 R0052
结果如图2所示,18 株瑞士乳杆菌基因组之间ANI 均大于97.25%,平均ANI 值为98.52%。根据ANI 值分布,18 株瑞士乳杆菌可分为4 个类群,即类群I、类群II、类群III 和类群IV,瑞士乳杆菌H9 和H10 分别属于类群III 和类群I,聚类结果与系统发育树基本一致;瑞士乳杆菌H9 与H10 间的ANI 值为98.02%,瑞士乳杆菌H9 与包括工业发酵剂菌株瑞士乳杆菌CNRZ32 在内的9株发酵乳制品分离株之间平均ANI 值为99.01%,瑞士乳杆菌H10 与CAUH18 之间的ANI 值最高(99.48%),与其它菌株之间的ANI 值均较低(平均97.99%);人类肠道和粪便分离株类群(类群II和类群IV)内部菌株间ANI 值极高(平均99.99%),而乳制品分离株类群(类群I 和类群III)内部菌株间ANI 值较低(平均98.50%),表明瑞士乳杆菌发酵乳制品分离株的基因组序列一致性比人类肠道/粪便分离株低,推测瑞士乳杆菌基因组序列多样性可能与其分离源有关。
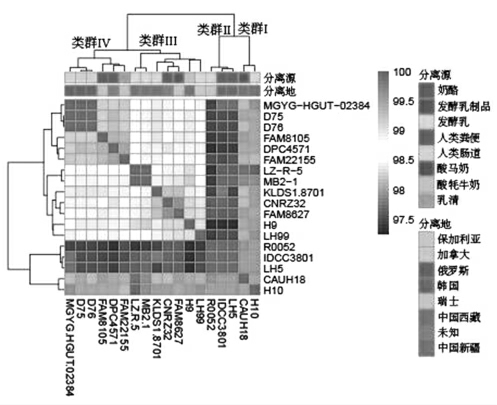
图2 18 株瑞士乳杆菌的ANI 值热图
Fig.2 Heatmap of ANI based on the genome sequences of 18 L.helveticus
2.4 共线性分析
共线性分析可通过检测不同物种间或同一物种不同个体基因组间同源序列及其排列顺序的一致性来研究基因组间的相关性,基因组相关性越高则其中同源序列一致性越高[22]。瑞士乳杆菌DPC4571 分离自干酪,并于2008年完成全基因组测序,是第1 株完成全基因组测序的瑞士乳杆菌菌株[23]。本研究以瑞士乳杆菌DPC4571 菌株基因组序列作为参考,利用Mauve 软件对瑞士乳杆菌H9 和H10 基因组进行共线性分析。
结果如图3所示,与瑞士乳杆菌DPC4571 比较,瑞士乳杆菌H10 有976 kb 的大片段倒置,而瑞士乳杆菌H9 基因组只存在局部倒置,其中主要含有cpoA (α-半乳糖基葡萄糖基二酰基甘油合酶)、ItaS1(脂磷壁酸合酶1)等与细胞膜、细胞壁合成及tRNA 和rnr(核糖核酸酶R)等tRNA 修饰相关基因。瑞士乳杆菌H9 基因组中有2 处约17 kb 左右片段缺失,其中主要含有bbmA(糖基水解酶)、cysE(丝氨酸乙酰转移酶)、ymdB(O-乙酰基-ADP-核糖脱乙酰酶)等涉及多糖、氨基酸及能量代谢等过程相关基因;瑞士乳杆菌H9 基因组有一处约10 kb 的特有片段,其中主要含有msrB(肽蛋氨酸亚砜还原酶)和deoD(嘌呤核苷磷酸化酶)等与蛋白质和核酸的修饰相关基因;瑞士乳杆菌H10 有一处35.36 kb 特有片段,包含sasA(自适应感应激酶)、pepDA(二肽酶A)等基因,主要与环境感应和蛋白水解等过程相关。
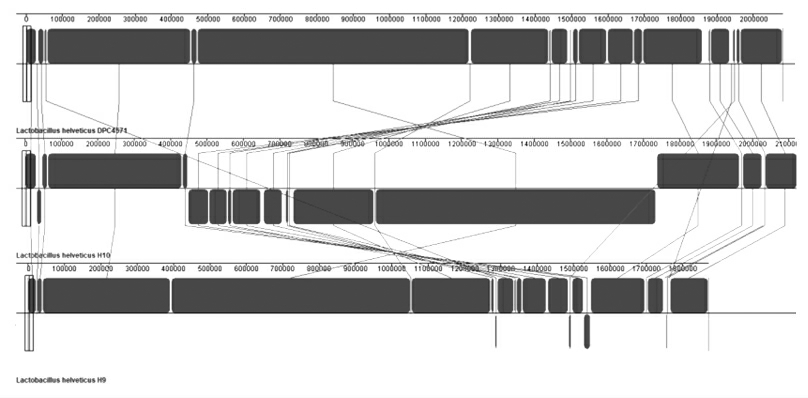
图3 瑞士乳杆菌基因组序列共线性分析
Fig.3 Synteny analysis of L.helveticus genomic sequences
基因片段插入和缺失是LAB 进化过程中的常见现象。LAB 菌株在适应其富营养环境过程中,通过基因插入和缺失等事件丢失非必须基因和功能,同时以同源重组和水平基因转移等途径获得有利于其适应生存环境的功能基因[15,24]。两株瑞士乳杆菌均有一定的基因片段插入或缺失现象发生,提示菌株在进化过程中可能经历了基因重组和水平基因转移等事件[25]。
2.5 泛-核心基因集(Pan-core genome)的构建及菌株特有功能基因分析
为比较不同菌株所含基因差异,利用Roary软件构建泛-核心基因集。18 株瑞士乳杆菌的泛基因集包括5 792 个基因,其中核心基因1 107个,特有基因1 527 个。
由图4可知,随着基因组数量的增加,核心基因个数逐渐减少,且趋于稳定,泛基因个数持续增加,表明瑞士乳杆菌基因组具有开放性,即随着基因组数量的增加,仍有出现新基因的趋势[26]。
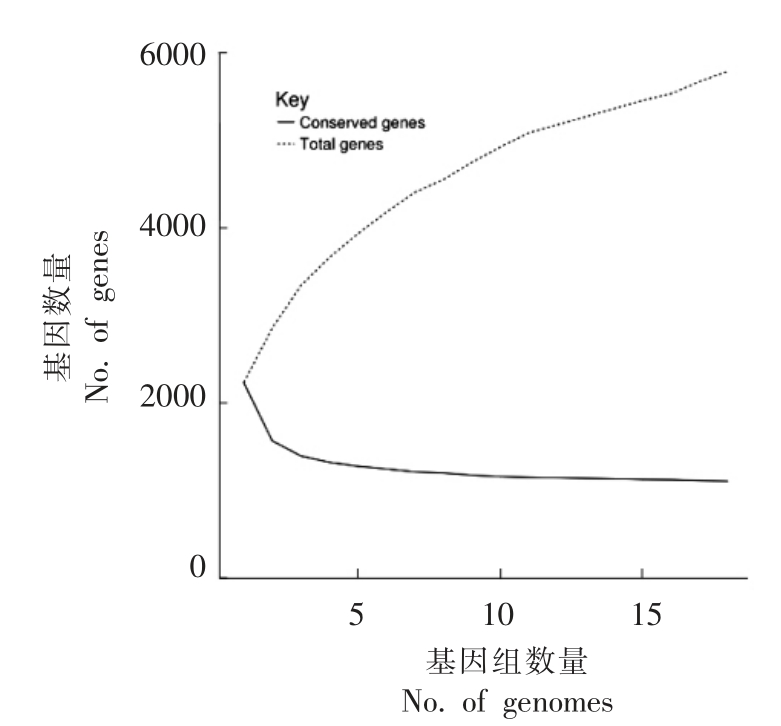
图4 泛-核心基因集变化趋势图
Fig.4 The trend chart of the size of core-pan genes
在18 株瑞士乳杆菌的1 527 个特有基因中,除去假定蛋白和插入序列等单元,瑞士乳杆菌H9有5 个、瑞士乳杆菌H10 有22 个特有功能基因(表2)。瑞士乳杆菌H9 的特有功能基因主要涉及氨基酸合成(dapH)和能量代谢(dkgA)等途径,其中dapH 有2 个拷贝;瑞士乳杆菌H10 的特有功能基因,主要参与氨基酸代谢(Csd、sufS、fbiB、pspA、sdhA)、能量代谢(Hpr、pox5、apbE、nfr1、nfr2)、跨膜转运(lutP、lysP、ytrE)等途径。将糖类转化为乳酸是乳酸菌获取能量的主要途径[27]。瑞士乳杆菌在其适应乳环境过程中丢失多种氨基酸的合成代谢途径相关基因,依靠其蛋白水解系统降解乳蛋白获取所需氨基酸[28]。瑞士乳杆菌H10 特有功能基因更多涉及能量代谢和氨基酸代谢,推测瑞士乳杆菌H9 的营养缺陷相对更严重,瑞士乳杆菌H10 可能具有更强的产酸能力,但需进一步研究验证。
表2 瑞士乳杆菌H9 和H10 特有功能基因
Table 2 The unique genes of L.helveticus H9 and L.helveticus H10
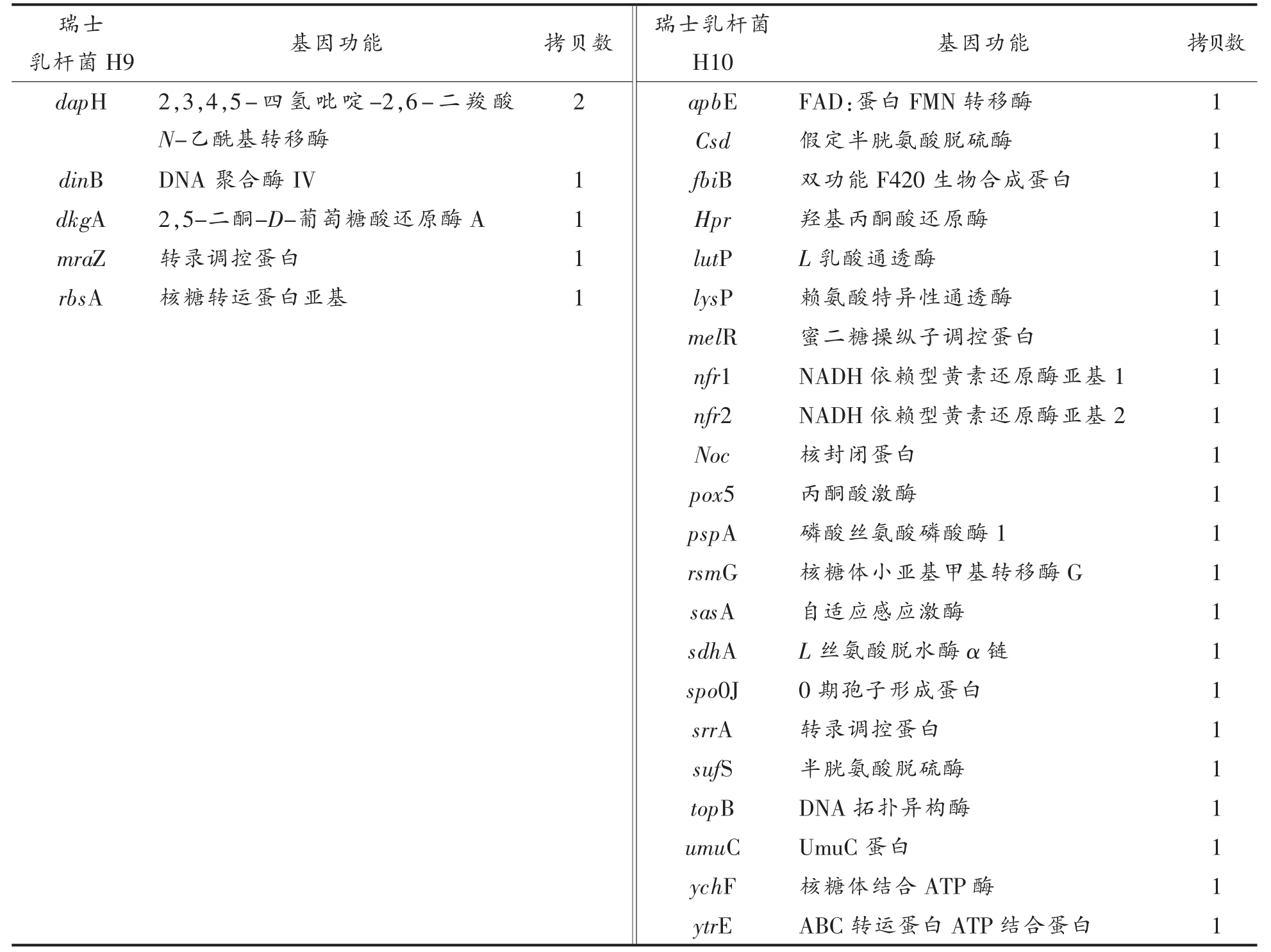
乳杆菌H9 基因功能 拷贝数 瑞士乳杆菌H10 基因功能 拷贝数瑞士dapH 2,3,4,5-四 氢 吡 啶-2,6-二 羧 酸N-乙酰基转移酶2 apbE FAD:蛋白FMN 转移酶 1 Csd 假定半胱氨酸脱硫酶 1 dinB DNA 聚合酶IV 1 fbiB 双功能F420 生物合成蛋白 1 dkgA 2,5-二酮-D-葡萄糖酸还原酶A 1 Hpr 羟基丙酮酸还原酶 1 mraZ 转录调控蛋白 1 lutP L 乳酸通透酶 1 rbsA 核糖转运蛋白亚基 1 lysP 赖氨酸特异性通透酶 1 melR 蜜二糖操纵子调控蛋白 1 nfr1 NADH 依赖型黄素还原酶亚基1 1 nfr2 NADH 依赖型黄素还原酶亚基2 1 Noc 核封闭蛋白 1 pox5 丙酮酸激酶 1 pspA 磷酸丝氨酸磷酸酶1 1 rsmG 核糖体小亚基甲基转移酶G 1 sasA 自适应感应激酶 1 sdhA L 丝氨酸脱水酶α 链 1 spo0J 0 期孢子形成蛋白 1 srrA 转录调控蛋白 1 sufS 半胱氨酸脱硫酶 1 topB DNA 拓扑异构酶 1 umuC UmuC 蛋白 1 ychF 核糖体结合ATP 酶 1 ytrE ABC 转运蛋白ATP 结合蛋白 1
3 结论
本研究以瑞士乳杆菌H9 和H10 为例,结合NCBI 数据库中其它16 株瑞士乳杆菌全基因组完成图,基于比较基因组学分析发现瑞士乳杆菌不同菌株基因组大小、质粒数量、CDSs、tRNA、rRNA数量均存在差异;瑞士乳杆菌H9 和H10 基因组大小、质粒、CDSs、tRNA 数量和染色体结构均有差异。系统发育树结果显示,瑞士乳杆菌H9 与工业发酵剂菌株瑞士乳杆菌CNRZ32 的系统发育关系更近,瑞士乳杆菌H10 更接近外群菌株;平均核苷酸一致性聚类结果与系统发育树大致相同,瑞士乳杆菌H9 和H10 聚类于不同类群,瑞士乳杆菌发酵乳制品分离株的基因组序列一致性低于人类肠道/粪便分离株;与参考菌株瑞士乳杆菌DPC4571 相比,瑞士乳杆菌H9 和H10 均有不同程度的片段插入、缺失和倒置;瑞士乳杆菌H9 和H10 特有功能基因存在差异,瑞士乳杆菌H10 的特有功能基因更多涉及氨基酸和能量代谢。本文通过比较基因组学分析发现瑞士乳杆菌不同菌株基因组之间存在遗传差异,为后续对瑞士乳杆菌的研究和应用奠定遗传学基础。
[1]SLATTERY L,O'CALLAGHANET J,FITZGERALD G F,et al.Invited review:Lactobacillus helveticus——a thermophilic dairy starter related to gut bacteria[J].J Dairy Sci,2010,93(10):4435-4454.
[2]SKRZYPCZAK K,GUSTAWW ANDWASKO A.Health-promoting properties exhibited by Lactobacillus helveticus strains[J].ActaBiochim Pol,2015,62(4):713-720.
[3]孙志宏.群体基因组学在乳酸菌研究中的应用[J].中国食品学报,2017,17(8):11-18.
SUN Z H.Application of population genomics in the research of lactic acid bacteria[J].Journal of Chinese Institute of Food Science and Technology,2017,17(8):11-18.
[4]MILLER W,MAKOVAKATERYNA D,NEKRUTENKO A,et al.Comparative genomics[J].Annu Rev Genomics Hum Genet,2004,5(1):15-56.
[5]KANT R,BLOMJOCHEN,PALVAAIRI,et al.Com parative genomics of Lactobacillus[J].Microb Biotechnol,2011,4(3):323-332.
[6]RICKER N,QIAN H,FULTHORPER.R.The limitations of draft assemblies for understanding prokaryotic adaptation and evolution[J].Genomics,2012,100(3):167-175.
[7]CHEN Y,LIU W,XUE J,et al.Angiotensin-converting enzyme inhibitory activity of Lactobacillus helveticus strains from traditional fermented dairy foods and antihypertensive effect of fermented milk of strain H9[J].J Dairy Sci,2014,97(11):6680-6692.
[8]CHEN Y,ZHANG W,SUN Z,et al.Complete genome sequence of Lactobacillus helveticus H9,a probiotic strain originated from kurut[J].J Biotechnol,2015,194(1):37-38.
[9]ZHAO W,CHEN Y,SUN Z,et al.Complete genome sequence of Lactobacillus helveticus H10[J].J Bacteriol,2011,193(10):2666-2667.
[10]GORIS J,KONSTANTINIDISET K T,KLAPPENBACH J A,et al.DNA-DNA hybridization values and their relationship to whole-genome sequence similarities[J].Int J SystEvolMicrobiol,2007,57(Pt 1):81-91.
[11]SEEMANN T.Prokka:rapid prokaryotic genome an notation[J].Bioinformatics,2014,30(14):2068-2069.
[12]PAGE A J,CUMMINSET C A,HUNT M,et al.Roary:rapid large-scale prokaryote pan genome analysis[J].Bioinformatics,2015,31(22):3691-3693.
[13]PRICE M N,Dehal P S,ARKIN A P.FastTree:computing large minimum evolution trees with profiles instead of a distance matrix[J].MolBiolEvol,2009,26(7):1641-1650.
[14]DARLINGA C E,MAU B,BLATTNERF R,et al.Mauve:Multiple alignment of conserved genomic sequence with rearrangements[J].Genome Research,2004,14(7):1394-1403.
[15]MAKAROVAK,SLESAREV A,WOLF Y,et al.Comparative genomics of the lactic acid bacteria[J].PNAS,2006,103(42):15611-15616.
[16]BROADBENT J R,HUGHES J E,WELKER D L,et al.Complete genome sequence for Lactobacillus helveticus CNRZ32,an industrial cheese starter and cheese flavor adjunct[J].Genome Announc,2013,1(4):e00590-13.
[17]宋宇勤.德氏乳杆菌保加利亚亚种的群体遗传学和功能基因组学研究[D].呼和浩特:内蒙古农业大学,2018.
SONG Y Q.Study on population genetics and functional genomics of Lactobacillus delbrueckii subsp.bulgaricus[D].Hohhot:Inner Mongolia Agricultural University,2018.
[18]王晓伟.不同地区自然发酵乳制品中瑞士乳杆菌的多位点序列分型研究[D].呼和浩特:内蒙古农业大学,2015.
WANG X W.Multilocus sequence typing of Lactobacillus helveticus from naturally fermented dairy products in different regions[D].Hohhot:Inner Mongolia Agricultural University,2015.
[19]SUN Z,HARRISET H M B,MCCANN A,et al.Expanding the biotechnology potential of lactobacilli through comparative genomics of 213 strains and associated genera[J].Nat Commun,2015,6(1):8322.
[20]RICHTER M,ROSSELLO-MORA R.Shifting the genomic gold standard for the prokaryotic species definition[J].PNAS,2009,106(45):19126-19131.
[21]ZHONG Z,KWOK L,HOU Q,et al.Comparative genomic analysis revealed great plasticity and environmental adaptation of the genomes of Enterococcus faecium[J].BMC Genomics,2019,20(1):602.
[22]TANG H,BOWERS J E,WANG X,et al.Synteny and collinearity in plant genomes[J].Science,2008,320(1):486-488.
[23]CALLANAN M,KALETA P,O'CALLAGHAN J,et al.Genome sequence of Lactobacillus helveticus,an organism distinguished by selective gene loss and insertion sequence element expansion[J].J Bacteriol,2008,190(2):727-735.
[24]CREMONESI P,CHESSA S,CASTIGLIONI B.Genome sequence and analysis of Lactobacillus helveticus[J].Front Microbiol,2012,3(1):435.
[25]LI W,YANG L,NAN W,et al.Whole-genome sequencing and genomic-based acid tolerance mechanisms of Lactobacillus delbrueckii subsp.bulgaricusLJJ[J].Appl Microbiol Biotechnol,2020,104(17):7631-7642.
[26]MEDINI D,DONTANI C,TETTELIN H,et al.The microbial pan-genome[J].Curr Opin Genet Dev,2005,15(6):589-594.
[27]GANZLEMICHAEL G.Lactic metabolism revisited:metabolism of lactic acidbacteria in food fermentations and food spoilage[J].Current Opinion in Food Science,2015,2(1):106-117.
[28]CHRISTIANSEN J K,HUGHES J E,WELKER D L,et al.Phenotypic and genotypic analysis of amino acid auxotrophy in Lactobacillus helveticus CNRZ32[J].Appl Environ Microbiol,2008,74(2):416-423.