克罗诺杆菌属(Cronobacter spp.)是肠杆菌科内一类兼性厌氧、周生鞭毛、能运动、不产芽孢、短杆状革兰氏阴性菌,2008年被定义为新属,并被分为16 个生物群(biogroups)[1]。1980年以前,因该菌产黄色素的特性,故一直被称为“产黄色素阴沟肠杆菌(yellow-pigmented Enterobacter cloacae)”。1980年Farmer 等[2]通过核酸杂交、生化试验、色素产生、药敏特性分析,将该菌从阴沟肠杆菌中分离开来,更名为阪崎肠杆菌(Enterobacter sakazakii)。2008年,Iversen 等[3-5]通过荧光标记-扩增片段长度多态性指纹图谱(f-AFLP)、DNA 杂交、16s rDNA 全序列分析方法重新对阪崎肠杆菌进行系统分类研究,结果发现这一菌种的许多菌株的鉴定结果差异较大,建议将阪崎肠杆菌定义为一个新属,即克罗诺杆菌属(Cronobacter spp.),该属包括7 个种,3 个亚种。
阪崎克罗诺杆菌(Cronobacter sakazakii)是克罗诺杆菌属中重要的条件致病菌,主要感染早产儿、出生体重偏低等身体状况较差的新生儿及婴幼儿,引起新生儿脑膜炎、小肠结肠炎、菌血症等疾病,并可能引发严重的神经系统后遗症[6-7]。有研究表明,该菌可感染老人及免疫力低下人群[8]。婴儿配方奶粉被认为是阪崎克罗诺杆菌的主要传播媒介。许多文献[9-12]报道在奶酪、肉制品、谷物制品、香肠、冰激凌、蔬菜等即食食品中均可分离到阪崎克罗诺杆菌。目前,食品中克罗诺杆菌的最初来源还未确定,推测植物性材料是重要来源。
关于阪崎克罗诺杆菌的致病机制目前了解较少,不同菌株的毒力差异明显,采用合适的分型方法对食品中污染的阪崎克罗诺杆菌进行溯源分析,阐明其在食品中流行规律,对控制食品中阪崎克罗诺杆菌污染意义重大。多位点序列分型(Multi Locus Sequence Typing,MLST)技术简单快速,分辨率高,数据结果易于实验室间进行比对,被广泛应用于细菌分子分型研究[13-14]。截至2018年5月,克罗诺杆菌MLST 数据库(https://pubmlst.org/cronobacter/)已收录来源为食品样品、临床样品、环境样品等分离菌株1 875 株,包括602 种ST 型,为克罗诺杆菌MLST 序列比对、进化分析提供充足的数据支撑。
本文对即食食品中阪崎克罗诺杆菌分离菌株进行鉴定及分子分型,研究不同样品中阪崎克罗诺杆菌的亲缘关系,为寻找食品中阪崎克罗诺杆菌可能的污染来源、流行规律及其防控提供参考依据。
1 材料与方法
1.1 材料与试剂
试验样品为市售预包装即食食品,所有样品按照《食品安全抽样检验管理办法》的要求进行抽样和保存。
α-氰基-4-羟基肉桂酸(HCCA)基质,德国Bruker 公司;Bacteria Test Standard(BTS)校准标准品,德国Bruker 公司;革兰氏阴性细菌鉴定卡(货号:21341),法国梅里埃公司;细菌基因组提取试剂盒(货号:9164),日本Takara 公司;阪崎克罗诺杆菌标准菌株,美国典型培养物保存中心(ATCC);阴沟肠杆菌标准菌株,中国医学细菌保藏管理中心(CMCC)。
1.2 仪器与设备
Autoflex speed 基质辅助激光解析/电离飞行时间质谱仪 (配有BioTyperTM 2.0 比对软件及flexAnalysis 3.4 质谱分析软件),德国Bruker 公司;VITEK 2 Compact System 全自动微生物鉴定系统,法国梅里埃公司;梯度PCR 仪,美国Eppendorf 公司;Universal Hood II 型凝胶成像仪,美国Bio-Rad 公司;5804R 型Eppendorf 冷冻离心机,美国Eppendorf 公司。
1.3 方法
1.3.1 试验菌株分离 所有样品均在无菌条件下进行处理,按照GB 4789.40-2016《食品微生物学检验 克罗诺杆菌属(阪崎肠杆菌)检验》[15]标准检验方法,对即食食品样品中克罗诺杆菌进行分离培养,分离的菌株用胰酪大豆胨琼脂(TSA)纯化2次后,保存菌种备用,试验菌株编号及样品来源见表1。
1.3.2 基质辅助激光解析/电离飞行时间质谱(MALDI-TOF-MS)数据采集及分析 将试验菌株划线接种于TSA 平板,36 ℃,培养16~18 h,挑取单菌落涂布在MALDI-TOF-MS 靶板上,滴加1 μL 70%(V/V)的甲酸,室温自然干燥后,加1 μL α-氰基-4-羟基肉桂酸(HCCA)基质溶液,自然晾干备用。同样方法在靶板上制备Bacteria Test Standard (BTS) 标准品校准靶位。采用FlexControlTM 3.0 软件中细菌鉴定标配方法MBT_FC 参数获取BTS 标准品质谱图,对机器进行校准。使用校准后的方法参数获取样品质谱图,每个样品叠加测定5 次。将获取的样品质谱导入BioTyperTM 2.0菌种鉴定软件进行质谱数据比对分析,获得鉴定结果。利用flexAnalysis 3.4 软件分析试验菌株质谱图,分析各试验菌株质谱离子峰差异。
1.3.3 试验菌株生化鉴定 采用全自动微生物鉴定系统(VITEK 2 Compact System)配套的革兰氏阴性细菌鉴定卡,按照说明书操作,对试验菌株进行生化鉴定。
1.3.4 16 s rDNA 序列分析 采用细菌基因组提取试剂盒,按照说明书操作提取试验菌株的基因组DNA。16s rDNA 序列扩增按照文献[16]报道方法进行。通过NCBI 网站(http://blast.ncbi.nlm.nih.gov/Blast.cgi)BLAST 程序获取试验菌株16s rDNA的同源序列。利用MEGA4.0 软件采用邻接法算法(Neighbor-Joining),以1 000 循环进行稳定性检测,构建进化树。同时,采用最大简约算法(maximum-parsimony)和最大似然算法(maximum-likelihood)构建进化树,验证邻接法进化树的准确性。
1.3.5 多位点序列分型分析 采用PubMLST 数据 库(https://pubmlst.org/cronobacter/info/protocol.shtml) 提供的克罗诺杆菌属多位点序列分型引物和扩增程序,对分离菌株的7 个等位基因(atpD、fusA、glnS、gltB、gyrB、infB、pps) 进行扩增并测序。将获得的等位基因序列输入PubMLST 数据库分析待测菌株序列型(Sequence type,ST)。将分离菌株的7 个等位基因序列串联后利用MEGA4.0软件构建分离菌株进化树,同时将分离菌株串联序列与PubMLST 数据库中322 株(截至2018年5月)中国分离的阪崎克罗诺杆菌构建进化树,分析本研究分离的10 株阪崎克罗诺杆菌与中国分离菌株之间的亲缘关系。
2 结果与分析
2.1 试验菌株分离鉴定结果
从即食食品样品中共分离164 株细菌,经MALDI-TOF-MS 快速筛选鉴定,其中10 株为阪崎克罗诺杆菌。阪崎克罗诺杆菌标准菌株(ATCC 29544)和阴沟肠杆菌标准菌株(CMCC(B)45301)分别作为标准阳性和标准阴性参考菌株,试验菌株鉴定结果及分离来源见表1。
表1 试验菌株鉴定结果及分离来源
Table 1 The resource and identification results of the isolates
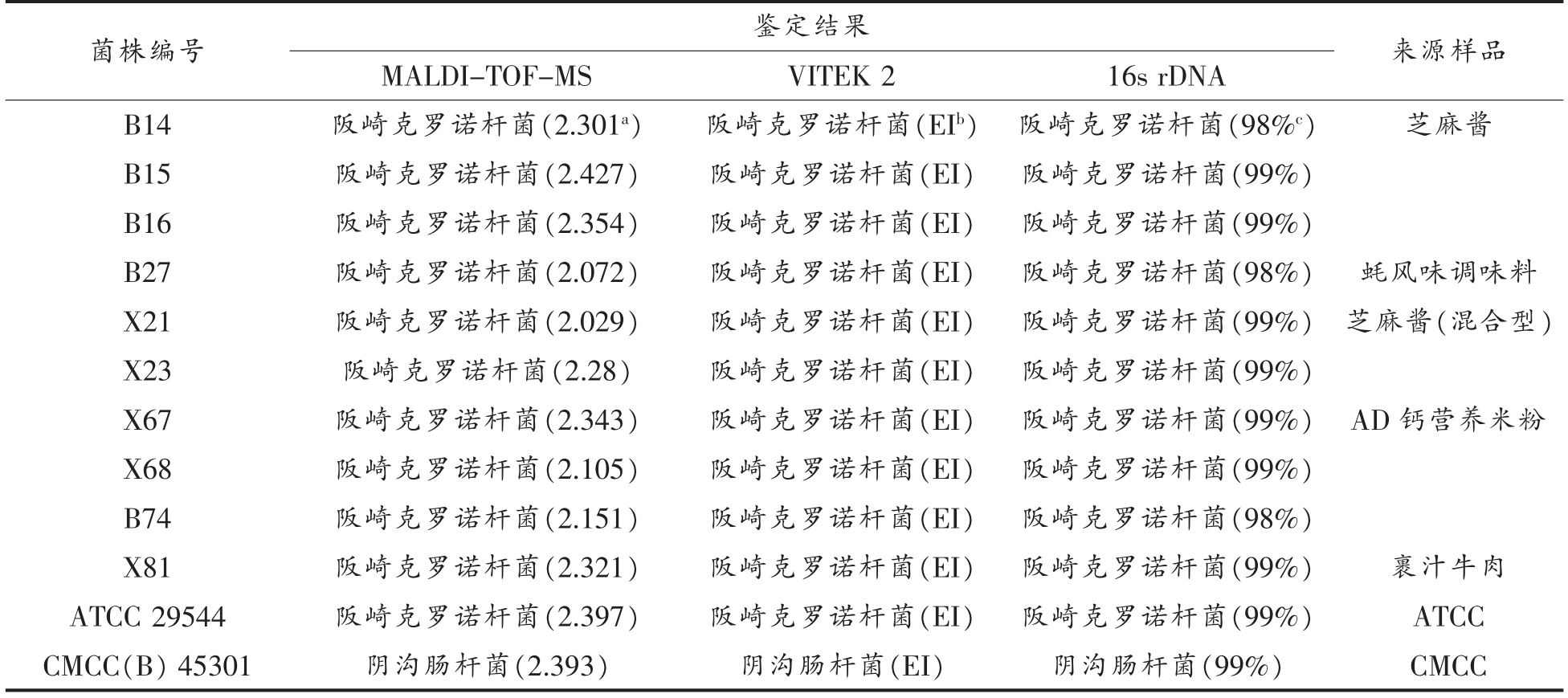
备注:a 表示BioTyperTM 2.0 菌种鉴定软件给出的鉴定分值;b 表示VITEK 2 鉴定结果为Excellent identification;c 表示16s rDNA 序列与鉴定结果的相似度。
菌株编号 鉴定结果 来源样品MALDI-TOF-MS VITEK 2 16s rDNA B14 阪崎克罗诺杆菌(2.301a) 阪崎克罗诺杆菌(EIb) 阪崎克罗诺杆菌(98%c) 芝麻酱B15 阪崎克罗诺杆菌(2.427) 阪崎克罗诺杆菌(EI) 阪崎克罗诺杆菌(99%)B16 阪崎克罗诺杆菌(2.354)阪崎克罗诺杆菌(EI) 阪崎克罗诺杆菌(99%)B27 阪崎克罗诺杆菌(2.072)阪崎克罗诺杆菌(EI) 阪崎克罗诺杆菌(98%) 蚝风味调味料X21 阪崎克罗诺杆菌(2.029) 阪崎克罗诺杆菌(EI) 阪崎克罗诺杆菌(99%) 芝麻酱(混合型)X23 阪崎克罗诺杆菌(2.28) 阪崎克罗诺杆菌(EI) 阪崎克罗诺杆菌(99%)X67 阪崎克罗诺杆菌(2.343)阪崎克罗诺杆菌(EI) 阪崎克罗诺杆菌(99%) AD 钙营养米粉X68 阪崎克罗诺杆菌(2.105)阪崎克罗诺杆菌(EI) 阪崎克罗诺杆菌(99%)B74 阪崎克罗诺杆菌(2.151)阪崎克罗诺杆菌(EI) 阪崎克罗诺杆菌(98%)X81 阪崎克罗诺杆菌(2.321)阪崎克罗诺杆菌(EI) 阪崎克罗诺杆菌(99%) 裹汁牛肉ATCC 29544 阪崎克罗诺杆菌(2.397) 阪崎克罗诺杆菌(EI) 阪崎克罗诺杆菌(99%) ATCC CMCC(B) 45301 阴沟肠杆菌(2.393)阴沟肠杆菌(EI) 阴沟肠杆菌(99%) CMCC
2.2 MALDI-TOF-MS 分析结果
所有试验菌株经MALDI-TOF-MS 鉴定分值均大于2.0(可靠鉴定到属的水平,有可能鉴定到种水平),且鉴定结果与VITEK 2 生化鉴定及16s rDNA 鉴定结果一致(表1)。利用flexAnalysis 3.4软件分析试验菌株质谱图(图1),所有阪崎克罗诺杆菌在5 740 m/z 附近均检测到离子峰,阴沟肠杆菌标准菌株(CMCC(B) 45301)在5 740 m/z 附近未检测到离子峰,表明5 740 m/z 附近的蛋白离子峰为阪崎克罗诺杆菌鉴定的特征蛋白。分析即食食品样品来源的阪崎克罗诺杆菌与标准菌株(ATCC 29544)的质谱图,发现所有野生菌株在7 159 m/z 附近均检测到离子峰,但ATCC 29544在7 159 m/z 附近未检测到离子峰,表明食品样品中的阪崎克罗诺杆菌与标准菌株ATCC 29544 菌型差异较大。比较B16、B27、B74、X23 野生菌株质谱图,在5 380,6 255 和7 159 m/z 附近的离子峰形差异较大,这些蛋白峰信息可作为进一步菌株分型的基础数据。

图1 阪崎克罗诺杆菌分离菌株MALDI-TOF-MS 质谱图
Fig.1 MALDI-TOF-MS mass spectrum of the C.sakazakii isolates
2.3 多位点序列分型分析结果
分离菌株的7 个等位基因(atpD、fusA、glnS、gltB、gyrB、infB、pps)序列经PubMLST 数据库比对分析,发现10 株阪崎克罗诺杆菌分离菌株分为4个ST 型,分别为ST21、ST73、ST60、ST7(表2)。AD钙营养米粉中分离的阪崎克罗诺杆菌菌株型复杂,3 株分离菌株X67、X68、B74 分别为3 种不同的ST 型ST7、ST60、ST21。ST73 型阪崎克罗诺杆菌仅在蚝风味调味料中检出,在所有食品样品中分离率最高的为ST21 型阪崎克罗诺杆菌(n=6)。将7 个等位基因序列串联后利用MEGA4.0 软件构建进化树,可见分离菌株分为4 个独立分支,分析结果与MLST 分型结果一致 (图2)。PubMLST数据库中国分离阪崎克罗诺杆菌聚类分析结果表明,国内阪崎克罗诺杆菌型式复杂,多种型式阪崎克罗诺杆菌流行,以ST21 型为主,临床样品、食品、婴幼儿配方食品中阪崎克罗诺杆菌分离较多。本研究分离的10 株阪崎克罗诺杆菌中,有5 株(X21、B14、B15、B16、X68) 与PubMLST 数据库中发布的临床及食品分离株亲缘关系较近,有3 株(B74、X67、X23)与香料及植物性食物中分离株亲缘关系较近,有1 株(X81)与婴儿配方奶粉中分离株亲缘关系较近(图3)。
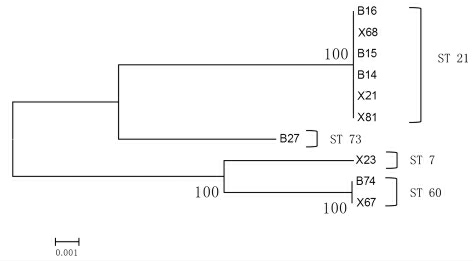
图2 阪崎克罗诺杆菌分离菌株MLST 序列进化树
Fig.2 The phylogenetic tree of the MLST sequences for the isolates

图3 PubMLST 数据库中国阪崎克罗诺杆菌分离菌株来源及分布
Fig.3 The resources and genotyping of the C.sakazakii of China in PubMLST database
表2 阪崎克罗诺杆菌分离菌株MLST 分型结果
Table 2 MLST analysis results of C.sakazakii isolates
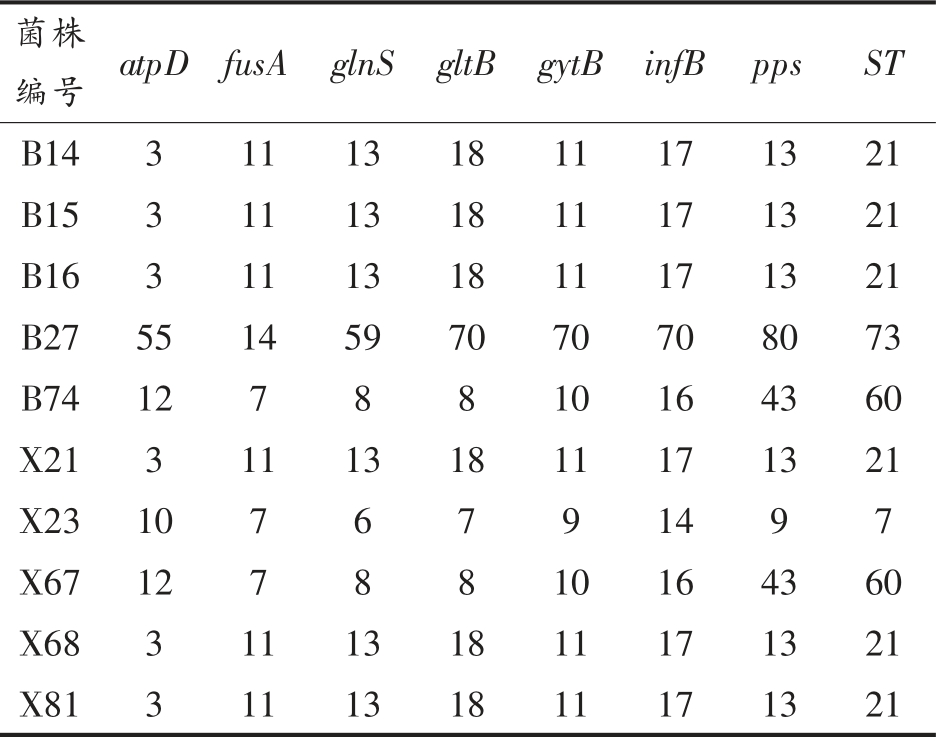
菌株编号 atpD fusA glnS gltB gytB infB pps ST B14 3 11 13 18 11 17 13 21 B15 3 11 13 18 11 17 13 21 B16 3 11 13 18 11 17 13 21 B27 55 14 59 70 70 70 80 73 B74 12 7 8 8 10 16 43 60 X21 3 11 13 18 11 17 13 21 X23 10 7 6 7 9 14 9 7 X67 12 7 8 8 10 16 43 60 X68 3 11 13 18 11 17 13 21 X81 3 11 13 18 11 17 13 21
3 结论与讨论
阪崎克罗诺杆菌污染配方乳粉引起新生儿及婴幼儿感染事件时有发生[17-20],我国GB 10765-2010《食品安全国家标准 婴儿配方食品》[21]中规定婴儿配方食品中阪崎克罗诺杆菌限量标准为“不得检出/100 g”。除感染新生儿及婴幼儿外,阪崎克罗诺杆菌对免疫低下人群依然存在较高的感染风险[8]。该菌被国际食品微生物标准委员会列入“对特定人群产生严重的生命危害、或产生慢性后遗症”的微生物,与肉毒梭菌的A、B 型毒素、单核细胞增生李斯特菌等具有同等的危害[22]。即食食品受众人群广泛,涵盖从幼儿到老人各个年龄段人群,且食用方式简单,一般没有再加工过程,微生物学风险较高,因此其安全性备受关注[23-26]。李远宏等[27]在2016年对100 份谷类食品进行克罗诺杆菌检测,发现谷类食品中克罗诺杆菌的分离率为21%,分离菌株中阪崎克罗诺杆菌占81%。本研究从即食食品样品中分离到164 株疑似克罗诺杆菌,通过MALDI-TOF-MS 快速筛选,VITEK 2和16s rDNA 序列分析进一步鉴定,其中10 株为阪崎克罗诺杆菌,主要分离来源为芝麻酱、米粉等谷物类食品。
MALDI-TOF-MS 技术通过获取核糖体蛋白质质谱图后与数据库中标准菌株质谱图比对,完成待测菌的鉴定。与其它鉴定技术相比,MALDITOF-MS 具有简单、快速、低成本、高通量的优势,分析待测菌质谱图,可获得菌株特异性标志蛋白,有利于深入挖掘菌株特征信息。本研究发现质荷比为5 740 m/z 的蛋白质为阪崎克罗诺杆菌的特征蛋白,质荷比为5 380,6 255 和7 159 m/z 的蛋白质仅在野生分离株中存在,可作为阪崎克罗诺杆菌分型、致病性、耐受性等特征分析的靶蛋白,该结果与赵红阳等[28]的研究结果吻合。
PubMLST 数据库中丰富的数据信息为克罗诺杆菌分型分析提供了基础,本研究采用MLST技术对分离菌株进行分型,同时与PubMLST 数据库中322 株阪崎克罗诺杆菌中国分离株进行亲缘关系分析。发现分离的5 株阪崎克罗诺杆菌与PubMLST 数据库中临床分离株紧密相关,提示即食食品样品中阪崎克罗诺杆菌有潜在的致病风险。已有研究结果表明[14,29],ST4、ST12、ST13、ST14型阪崎克罗诺杆菌能够导致坏死性小肠结肠炎、败血症、脑膜炎,本研究分离菌株主要为ST21 型,未分离到以上4 种ST 型的阪崎克罗诺杆菌。阪崎克罗诺杆菌中国分离株系统发育进化树显示我国阪崎克罗诺杆菌主要分离来源是临床和食品样品、婴儿配方食品,且与本研究ST21 型分离菌株亲缘关系较近,说明ST21 型阪崎克罗诺杆菌是我国的主要流行菌株。
本研究通过对即食食品样品中阪崎克罗诺杆菌进行分离、鉴定、分型,评估阪崎克罗诺杆菌在即食食品中潜在的传播和致病风险,结合PubMLST 数据库中中国分离株进化分析结果,分析了阪崎克罗诺杆菌在我国的主要流行菌株。本研究为分析食品中阪崎克罗诺杆菌可能的污染来源、流行规律及防控措施提供依据。通过了解食品及加工环境中克罗诺杆菌的污染来源,建立有效的预防控制措施,有助于降低克罗诺杆菌在即食食品中的传播风险。
[1]IVERSEN C,WADDINGTON M,FARMER J,et al.The biochemical differentiation of Enterobacter sakazakii genotypes[J].BMC Microbiology,2006,6(1):94.
[2]FARMER J,ASBURY M,HICKMAN F,et al.Enterobacter sakazakii:A new species of “Enterobacteriaceae” isolated from clinical specimens [J].International Journal of Systematic and Evolutionary Microbiology,1980,30(3):569-584.
[3]CAROL I,NIALL M,BARBARA M C,et al.Cronobacter gen.nov.,a new genus to accommodate the biogroups of Enterobacter sakazakii,and proposal of Cronobacter sakazakii gen.nov.,comb.nov.,Cronobacter malonaticus sp.nov.,Cronobacter turicensis sp.nov.,Cronobacter muytjensii sp.nov.,Cron[J].International Journal of Systematic and Evolutionary Microbiology,2008,58(pt6):1442-1447.
[4]IVERSEN C,LEHNER A,MARUGG J,et al.Identification of “Cronobacter” spp.(Enterobacter sakazakii)[J].Journal of Clinical Microbiology,2007,45(11):3814-3816.
[5]IVERSEN C,LEHNER A,MULLANE N,et al.The taxonomy of Enterobacter sakazakii:proposal of a new genus Cronobacter gen.nov.and descriptions of Cronobacter sakazakii comb.nov.Cronobacter sakazakii subsp.sakazakii,comb.nov.,Cronobacter sakazakii subsp.malonaticus subsp.nov.,Cronobacter[J].BMC Evolutionary Biology,2007,7(1):64.
[6]PARRA-FLORES J,CERDA-LEAL F,CONTRERAS A,et al.Cronobacter sakazakii and microbiological parameters in dairy formulas associated with a food alert in Chile[J].Frontiers in Microbiology,2018,9:1708.
[7]JANG H,ADDY N,EWING L,et al.Wholegenome sequences of Cronobacter sakazakii isolates obtained from foods of plant origin and dried-food manufacturing environments[J].Genome Announcement,2018,6(15):e00223-18.
[8]龚燕,顾学章.阪崎肠杆菌肺炎64 例临床及药敏分析[J].上海医学,2005,28(12):1047-1048.
GONG Y,GU X Z.Clinical and drug sensitivity analysis of 64 cases of Enterobacter sakazakii pneumonia[J].Shanghai Medical,2005,28(12):1047-1048.
[9]FEI P,MAN C,LOU B,et al.Genotyping and source tracking of Cronobacter sakazakii and C.malonaticus isolates from powdered infant formula and an infant formula production factory in China[J].Applied and Environmental Microbiology,2015,81(16):5430-5439.
[10]陈万义,任婧,吴正钧,等.生鲜蔬菜中阪崎克罗诺杆菌的分离与鉴定[J].食品科技,2014,39(1):304-308.
CHEN W Y,REN J,WU Z J,et al.Isolation and identification of Cronobacter sakazakii from raw vegetables[J].Food Science and Technology,2014,39(1):304-308.
[11]YE J Z,YU X,LI X S,et al.Antimicrobial resistance characteristics of and disinfectant-resistant gene distribution in Staphylococcus aureus isolates from male urogenital tract infection[J].National Journal of Andrology,2014,20(7):630-636.
[12]陆幸儿,郑悦康,吴灿权.10 类食品污染阪崎肠杆菌状况的调查研究[J].中国医学创新,2011,8(18):3-5.
LU X E,ZHENG Y K,WU C Q.Investigation of 10 kinds of food be poluted by Enterobacter sakazakii[J].Medical Innovation of China,2011,8(18):3-5.
[13]JOSEPH S,SONBOL H,HARIRI S,et al.Diversity of the Cronobacter genus as revealed by multilocus sequence typing[J].Journal of Clinical Microbiology,2012,50(9):3031-3039.
[14]BALDWIN A,LOUGHLIN M,CAUBILLA-BARRON J,et al.Multilocus sequence typing of Cronobacter sakazakii and Cronobacter malonaticus reveals stable clonal structures with clinical significance which do not correlate with biotypes[J].BMC Microbiology,2009,9:223.
[15]中华人民共和国国家卫生和计划生育委员会,国家食品药品监督管理总局. 食品安全国家标准 食品微生物学检验克罗诺杆菌属 (阪崎肠杆菌) 检验:GB 4789.40-2016[S]. 北京:中国标准出版社,2016.
National Health and Family Planning Commission of the People 's Republic of China,China Food and Drug Administration.National food safety standard microbiological examination of food Cronobacter(Enterobacter sakazakii):GB 4789.40-2016[S].Beijing:Standards Press of China,2016.
[16]JARADAT Z,ABABNEH Q,SAADOUN I,et al.Isolation of Cronobacter spp.(formerly Enterobacter sakazakii) from infant food,herbs and environmental samples and the subsequent identification and confirmation of the isolates using biochemical,chromogenic assays,PCR and 16S rRNA sequencing[J].BMC Microbiology,2009,9(1):1-11.
[17]FORSYTHE S,DICKINS B,JOLLEY K.Cronobacter,the emergent bacterial pathogen Enterobacter sakazakii comes of age;MLST and whole genome sequence analysis[J].BMC Genomics,2014,15:1121.
[18]YAN Q,CONDELL O,POWER K,et al.Cronobacter species (formerly known as Enterobacter sakazakii) in powdered infant formula:A review of our current understanding of the biology of this bacterium[J].Journal of Applied Microbiology,2012,113(1):1-15.
[19]CHEN Y,ZHANG Y F,WANG X H,et al.Antibacterial activity and its mechanisms of a recombinant Funme peptide against Cronobacter sakazakii in powdered infant formula[J].Food Research International,2019,116:258-265.
[20]SHUKLA S,HALDORAI Y,BAJPAI V,et al.Electrochemical coupled immunosensing platform based on graphene oxide/gold nanocomposite for sensitive detection of Cronobacter sakazakii in powdered infant formula[J].Biosensors & Bioelectronics,2018,109:139-149.
[21]中华人民共和国卫生部.食品安全国家标准 婴儿配方食品:GB 10765-2010[S]. 北京:中国标准出版社,2010.
Ministry of Health of the People' s Republic of China.National food safety standard:Infant formula:GB 10765-2010[S].Beijing:Standards Press of China,2010.
[22]IVERSEN C.Risk profile of Enterobacter sakazakii,an emergent pathogen associated with infant milk formula[J].Trends Food Science Technology,2003,14(11):443-454.
[23]罗梦幽,贺苏皖,白凤岚,等.成都市市售即食食品中克罗诺杆菌的分离鉴定及药敏检测[J].西南民族大学学报(自然科学版),2018,44(6):602-608.
LUO M Y,HE S W,BAI F L,et al.Isolation,identification and antimicrobial susceptibility detection of Cronobacter spp.from ready-to-eat food bought in Chengdu[J].Journal of Southwest Minzu University(Natural Science Edition),2018,44(6):602-608.
[24]VASCONCELLOS L,CARVALHO C,TAVARES R,et al.Isolation,molecular and phenotypic characterization of Cronobacter spp.in ready-to-eat salads and foods from Japanese cuisine commercialized in Brazil [J].Food Research International,2018,107:353-359.
[25]BERTHOLD-PLUTA A,GARBOWSKA M,STEFANSKA I,et al.Microbiological quality of selected ready-to-eat leaf vegetables,sprouts and non-pasteurized fresh fruit-vegetable juices including the presence of Cronobacter spp[J].Food Microbiology,2017,65:221-230.
[26]XU X K,LI C S,WU Q P,et al.Prevalence,molecular characterization,and antibiotic susceptibility of Cronobacter spp.in Chinese ready-to-eat foods[J].International Journal of Food Microbiol,2015,204:17-23.
[27]李远宏,张逸飞,张庆成,等.谷类食品中克罗诺杆菌的分离与鉴定[J].食品工业科技,2016,37(15):154-158.
LI Y H,ZHANG Y F,ZHANG Q C,et al.Isolation and identification of Cronobacter spp.from cereal foods[J].Science and Technology of Food Industry,2016,37(15):154-158.
[28]赵红阳,吕佳,卢雁,等.基质辅助激光解吸电离飞行时间质谱对阪崎肠杆菌的鉴定[J].中国微生态学杂志,2013,25(5):541-547.
ZHAO H Y,LV J,LU Y,et al.Identification of Enterobacter sakazakii by matrix-assisted laser desorption ionization-time of flight mass spectrometry[J].Chinese Journal of Microecology,2013,25 (5):541-547.
[29]CAUBILLA-BARRON J,HURRELL E,TOWNSEND S,et al.Genotypic and phenotypic analysis of Enterobacter sakazakii strains from an outbreak resulting in fatalities in a neonatal intensive care unit in France[J].Journal of Clinical Microbiology,2007,45(12):3979-3985.