基于三重四极杆的选择反应监测(Selective reaction monitoring,SRM)也称为多反应监测(Multiple reaction monitoring,MRM)是质谱定量的“金标准”,在蛋白质定量中广泛使用,而根据专一性肽段的母离子质量和子离子质量,最大程度地去除干扰离子,监测母离子与子离子形成的离子对信号响应,是目标蛋白验证和绝对定量的有效手段[1-2]。然而,随着定量蛋白质组学的深入发展,样本基质越来越复杂,目标蛋白丰度越来越低,容易受到高丰度蛋白的掩盖,而SRM 由于质量分辨率低,难以有效去除复杂基质背景的干扰,易造成假阳性结果。同时,随着分析通量的要求越来越高,一次分析可能需要监测成千上万个离子对,而由于SRM 灵敏度的局限,使得同时监测的离子对数量有限;此外,离子对、碰撞能量等条件的优化也费时、费力,难以满足目标蛋白质组学高通量发展的需要[3]。四极杆-高分辨串联质谱技术(如QTOF、QOrbitrap) 的发展以及扫描速度的提升,为目标蛋白定量提供了新的途径。数据依赖型采集(Data dependent acquisition,DDA)方式是一种随机选取丰度最高的前几名离子进行二级碎裂检测,在非标记蛋白质组学领域具有非常突出的优势,主要用于过敏原检测[1,4]、蛋白功能分析[5]、生物标志物和差异蛋白鉴别等[6-7]。基于DDA 策略的非标记蛋白质组学定量方法主要有两种:一种是信号强度法,即利用提取一级谱图峰面积或峰强度进行定量;另一种是谱图计数法,即通过统计同一蛋白质所有肽段二级谱图总数来确定蛋白质的相对含量,丰度越高的蛋白质肽段,被检测到的次数越多,其得到的二级谱图张数越多;前者基于一级峰面积或峰强度定量存在共流出肽段干扰定量而影响准确性的问题,后者基于二级谱图张数定量法则存在低丰度蛋白被掩盖而无法定量的情况。
定量蛋白质组学以定性和定量样本中所有蛋白质为目标,可分为标记定量和非标记定量,常用的标记方法为同位素标记试剂法。其中,同位素标记法准确度高,而试剂价格昂贵且可标记的样本数量有限;非标记定量技术无需昂贵的同位素试剂,耗费低,对样品酶解产物直接进行质谱分析,使其最接近原始状态,能够更好地反映出样品中蛋白质的差异性[8]。近年来,肉类掺假事件层出不穷,随着肉类价格差异不断扩大,不法商家为了追求利益,借机用价格低廉的鸡肉、马肉、鸭肉等伪制牛羊肉制品,使得消费者对肉类及其制品的食品安全问题甚为担忧。要想打击“掺假肉”泛滥的问题,首先要制定准确、可靠的检测方法。欧盟等在“马肉风波”后[9],批准了对肉制品进行DNA 检测的分子方法,其灵敏度高,操作简易,检测过程短,特别适用于原料肉中掺假肉的鉴别。然而,食品中DNA 成分复杂,加之该技术的高灵敏度,使得应用扩增方法来鉴定物种具有因非特异性扩增而产生假阳性的结果。同时,由于该方法无法区分同一种属来源的不同产品,如鸡肉和鸡蛋、牛肉和牛奶等,因而常采用质谱法鉴别特征肽质量图谱的结果做对比。本文采用纳流液相色谱-四极杆串联轨道阱高分辨质谱仪(nLC-QE)分析肉和肉丸样品,采用数据依赖型采集模式(DDA)分析筛选肉中的特异性肽段,定量测定肉蛋白,对比不同分离条件下特征肽段测定情况,并根据特征肽鉴别肉的种属以提高鉴别的准确性,为肉类掺假检测提供技术支持。
1 材料与方法
1.1 材料与试剂
试验所用的猪肉、牛肉、鸡肉、鸭肉等购于当地超市,剔除脂肪和筋膜,经绞肉机绞碎备用。市售牛肉丸购于当地市场,自制牛肉丸采用100%牛肉制成。
质谱级乙腈、甲醇、纯水,Fisher 公司;碘代乙酰胺(IAA)、测序级胰蛋白酶、质谱级甲酸、色谱级正己烷、二硫苏糖醇(DDT),Sigma-Aldrich 公司;三羟甲基氨基甲烷(Tris)、尿素、硫脲、牛血清白蛋白、考马斯亮蓝G250、碳酸氢铵、三羟甲基氨基甲烷(Tris),生工生物工程(上海)有限公司;氯化钾、磷酸二氢钾、磷酸氢二钾、浓盐酸、无水乙酸钠、冰乙酸、无水乙醇等均为分析纯级;超纯水(电阻率≥18.2 MΩ·cm),实验室自制。
1.2 仪器及设备
EASY-nLC 1200 纳升级液相色谱、Q Exactive 四极杆轨道阱高分辨质谱仪(QE),Thermo 公司;JY3002 电子天平,梅特勒-托利多仪器有限公司;DZF-6050 型真空干燥箱,巩义市予华仪器有限责任公司;ZX4 漩涡振荡器,意大利VELP 公司;IKA T25 高剪切均质机,IKA 公司;T1901 紫外可见光分光光度计,北京普析通用仪器有限责任公司;Direct-Q5 超纯水机,美国密理博有限公司;SIGMA 2-16P 高速离心机,西格玛有限公司;Scientz-950E 超声波细胞粉碎机,宁波新芝生物科技有限公司;HLB 固相萃取柱(3 mL/200 mg),Waters公司。
1.3 试验方法
1.3.1 原料处理及牛肉丸的制备 选择经卫生检疫合格的黄牛后腿肉1 000 g,剔除脂肪和筋膜,顺着纹路切成5 cm×5 cm×5 cm 的立方体,放入冰箱冷藏至5 ℃。斩拌过程中加入3 次冰,添加冰的总质量为所有原料总质量的15%,以保证肉糜温度在10 ℃以下,加入红薯粉80 g、味精4 g、肉弹素5 g、食盐25 g 等配料,斩拌至胶状肉泥。左手抓肉泥,从拇指和十指间挤出2.5 cm 左右的肉丸,右手用调羹接住后放入35 ℃的温水锅中,缓慢升温,待肉丸中心温度达80 ℃,肉丸浮起、变色,捞起。迅速放入15 ℃水中冷却15 min。放入冰箱-20℃冷冻,备用。
1.3.2 蛋白提取及含量测定 将瘦肉和肉丸样品切碎,各取0.5 g 样品加入2.5 mL 正己烷,超声3 min,涡旋5 min,于4 500 r/min 离心3 min,保留沉淀物。重复以上步骤2 次,用氮气吹干沉淀物。加入4 mL 不同的提取液,旋涡混匀30 s,超声处理3 min,在4 ℃下于12 000 r/min 离心5 min,取上清液采用考马斯亮蓝染色法测定蛋白质的含量,于4 ℃下保存备用。本实验用的蛋白质提取液主要有以下3 种,提取液1:0.3 mol/L KCl、0.15 mol/L KH2PO4、0.15 mol/L K2HPO4;提取液2:6 mol/L 尿素、1 mol/L 硫脲、50 mmol/L Tris-HCl;提取液3:0.1%三氟乙酸。
1.3.3 蛋白酶解与肽段纯化 取200 μL 混合溶液或肉丸蛋白酶解液,加入装有150 μL 碳酸氢铵(0.5 mol/L)的2 mL 离心管中,涡旋30 s,加入10 μL DTT 溶液(500 mmol/L),混匀后置于75 ℃水浴中处理30 min,以使溶液中的蛋白质变性,后将提取物冷却至室温。添加150 mmol/L 碘代乙酰胺使蛋白质烷基化,最终浓度为15 mmol/L,暗处反应30 min。随后,添加50 μL 牛胰蛋白酶溶液(0.5 mg/L,用30 mmol/L NH4HCO3 配制)和10 μL 氯化钙溶液(0.1 mol/L)。立即将酶解液涡旋1 min,置于37 ℃水浴中酶解12 h。向酶解液中加入20 μL甲酸终止反应,混匀后置于室温中15 min,加入水,使得溶液终体积为1 mL。取1 mL 酶解液过活化的HLB 柱,并用0.5 mL 体积分数1%的甲酸冲洗空的离心管,将洗涤液装入小柱,以定量转移样品[10]。然后用2 mL 体积分数1%的甲酸水洗涤小柱。最后,将肽用1 mL 乙腈/水混合液(体积比1∶1,含0.1%甲酸)洗脱到10 mL 离心管中,加入洗脱液后将小柱浸泡10 min,使得肽能完全洗脱。再加1 mL 混合洗脱液重复洗脱1 次。随后,将全部洗脱液在氮气流中吹干,色谱分析前将提取物用0.5 mL 的乙腈/水混合液(体积比3∶97,含有0.1%甲酸)复溶,涡旋30 s,过0.22 μm 有机系滤膜,上机检测。
1.3.4 色谱及质谱分析条件 将酶切肽段用Easy-nLC 1200 和QE 进行LC-HRMS 分析,肽段以800 bar 的压力直接上样到PepMap 100 C18分析柱上 (250 mm×75 μm,2 μm),再用300 nL/min 流速进行洗脱,流动相A 为体积分数0.1%甲酸水溶液,流动相B 为体积分数0.1%甲酸和80%乙腈混合溶液,梯度洗脱程序如表1所示,柱温箱温度为40 ℃。离子源为nano-Flex 纳喷雾离子源;扫描模式:正离子;毛细管电压为2.0 kV;离子传输管温度为320 ℃;RF-lens 为50%;DDA 采集模式,一级质谱分辨率70 000,自动增益目标值(AGC)为1×106,最大注入时间(IT)为100 ms,扫描范围为m/z 300~1 500;二级谱图分辨率设为17 500,最大注入时间为80 ms,扫描范围为m/z 150~2 000,隔离窗口设为1.6 u,归一化碎裂能量(NCE) 设为28,碎裂模式为HCD,自动增益值(AGC)为1×105,采集top N 设为20,其余参数按照默认设置。
表1 纳升级液相色谱分离梯度洗脱程序
Table 1 Gradient elution program employed for separation of peptides by nano LC

梯度1/min分离梯度 流动相A/%梯度2/min流动相B/%梯度3/min 0 0 0 98 2 2 2 2 28 2 40 70 100 68 32 53 83 113 63 37 57 87 117 0 100 60 90 120 0 100
1.3.5 数据库检索及统计分析 试验重复测定3次,结果以“平均值±标准偏差”表示,数据统计分析采用SPSS 17.0 软件进行一维方差分析,差异显著性采用Duncan(邓肯)检验,检验水平P<0.05。肽段DDA 数据使用MaxQuant 软件进行搜库鉴定,检索UniProt _Bovine(包含876 759 条蛋白序列)、_Chicken (包含709 277 条蛋白序列)、_Pig(包含709 277 条蛋白序列)、_Duck(包含842 876 条蛋白序列)数据库,一级质谱中母离子质量误差为:10 ppm,二级质谱中碎片离子质量精度:0.02 u,酶为胰蛋白酶(Trypsin),完全酶切,漏切数目为2 个,固定修饰为半胱氨酸的烷基化(C,+57.02150 u),可变修饰为甲硫氨酸的氧化(M,+15.99492 u),从鉴定结果中挑选4 种肉类的特征肽段。非标记定量使用Proteome Discoverer 2.4(Thermo Scientific)软件的 PWF_QE_Precursor_Quan_and_ LFQ_SequestHT_Percolator 模版搜索和 CWF_Comprehensive_Enhanced Annotation_LFQ_and_ Precursor_Quan 模板组装,检索fasta 数据库文件、一级质谱和二级质谱的参数同上,Percolator 结点进行FDR 值计算,PSM、肽段、蛋白质假阳性率均控制在1%。非标记定量用Minora Feature Detector 结点进行色谱峰提取;Feature Mapper 结点进行色谱峰和肽段鉴定结果的匹配,设置保留时间对齐,最大的保留时间偏差为10 min,最小信噪比为5;Precursor Ions Quantifier 结点进行定量结果输出,用Unique+Razor 肽的母离子信号强度进行定量、蛋白的强度为前三强(top 3)肽段强度的总和,数据输出后导入SIMCA 13.0 进行主成分分析(PCA)和二维正交偏最小二乘判别分析(O2PLS-DA)。
2 结果与分析
2.1 提取剂对蛋白质提取效果的影响
将牛肉、猪肉、鸡肉、鸭肉4 种纯肉样品和纯牛肉丸采用不同蛋白质提取剂,提取得到5 种蛋白质提取液,采用考马斯亮蓝染色法测定蛋白含量,结果如图1所示。
由图1可知,纯肉样品的最优提取剂为提取液3,而提取液2 次之,显著高于提取液1 的提取效果,这主要是由于提取液2 中加入了一定量的尿素,可促进蛋白质变性,有助于蛋白质的提取。纯牛肉丸样品的最优提取剂为提取液2,而提取液3 次之,此结果不同于纯肉样品中蛋白质的提取效果,可能是由于肉丸加工过程中蛋白质的变性程度大,并且受到其它组分的影响,而不利于蛋白质的提取。因此,后续步骤采用提取液2 提取纯肉和肉丸样品中的蛋白质,且在处理肉丸样品时增加搅碎时间3.5 min 和超声强化提取3 min,这与已报道文献[11]的纯肉蛋白提取液不适用于肉制品中蛋白提取的研究结果一致。

图1 提取剂对不同样品中蛋白质提取效果的影响
Fig.1 Effects of extraction buffer on efficiency of proteins from different samples
注:图中同一样品标注不同小写字母表示差异显著,P<0.05。
2.2 蛋白质的分析与鉴别
纯肉样品采用提取缓冲溶液2 提取蛋白,酶解后过HLB 柱,采用nLC-QE 高分辨质谱仪分析,纳升级液相色谱分离系统采用含2.0 μm 填料的纳米色谱柱,具有更出色的分离能力,在更高的压力下缩短了梯度分离时间,提高了蛋白质和多肽的分析通量和识别率,将液相不同洗脱时间分离的蛋白质酶解液的质谱数据进行分析,鉴定结果如表2所示。不同液相洗脱梯度对酶解液中蛋白和特异肽的鉴定有显著影响(P<0.05),在洗脱梯度为120 min 时,鉴定到的蛋白质数以及特异肽段数最多,而不同洗脱梯度对不同种属来源的特征肽数目的鉴定影响不大。本研究建立的数据依赖型采集模式在猪肉、牛肉、鸡肉和鸭肉中分别鉴定到80 554,33 350,40 969,32 923 条特异性多肽,其中序列相同、修饰不同的肽段视为不同的肽段,肽段电荷数为2~7,这些肽段在蛋白水平上分别归属于2 715,889,659,1 143 种蛋白质,与文献[12]采用纳升级液相色谱-四极杆轨道阱质谱法分离鉴别到的蛋白数量相当,而远高于文献[13]中采用超高效液相色谱-飞行时间质谱采集的结果。该方法鉴定到的蛋白质有较高的序列覆盖度,且不同洗脱梯度分离出的特异肽的信号强度分布波动非常小,如图2所示。结果表明该方法的稳定性和重现性较好。由此可知,非标记定量蛋白质组学技术鉴定到的蛋白质比较全面且可靠,可用于后续试验。

图2 不同洗脱梯度分离的肉类特异肽的丰度分布
Fig.2 Abundance distribution of meat marker peptides separated by different elution gradients
表2 不同梯度洗脱分离的肉蛋白酶解物中鉴定到的蛋白数和肽段数
Table 2 Identified protein numbers and peptide numbers from meat protein hydrolysates separated by different gradient elution

注:相同肉类不同梯度下鉴别的蛋白数或肽段数标注的不同小写肩标字母表示具有显著差异,P<0.05。
?
2.3 特征肽的筛选与鉴别
将4 种肉类分别用120 min 梯度洗脱重复测定5 次建库,用MaxQuant 软件鉴别肽段数据后,将鉴定到的肽段进行UniProt 的BLAST 搜索,分别找出不同种属肉中的特征肽段,结果如表3所示。
表3 特征肽来源及其特性
Table 3 Source and characteristics of marker peptides

特征肽段 蛋白来源 UniProt登录号母离子m/z(偏差/ppm)保留时间/min 肉种属AAENTIVVVAGNK 肌球蛋白结合蛋白C,快速型 P16419 643.35922+(1.7) 20.0 鸡(Gallus gallus)AAIAQAGYTDK β-烯醇酶 P07332 554.78582+(0.9) 14.8 AAPQFLTPLVDR 肌球蛋白结合蛋白C,快速型 P16419 664.37212+(1.3) 33.1 AVNEMGEATTECR 肌球蛋白结合蛋白C,快速型 P16419 734.31382+(-6.0) 16.6 EGGALPAEVQTR 肌球蛋白结合蛋白C,快速型 P16419 614.32062+(0.9) 18.9 GLEDQQVVVGDR 肌球蛋白结合蛋白C,快速型 P16419 657.83662+(0.91) 21.8 LGFSEVEQVQMVVDGVK 肌酸激酶M 型 P00565 932.47972+(2.3) 41.5 LSVEALNSLEGEFK 肌酸激酶M 型 P00565 768.4017852+(-0.7) 39.1 LVVELSDPDLPLK 肌球蛋白结合蛋白C,快速型 P16419 719.414162+(1.4) 35.6 NGQLLKPSTK 肌球蛋白结合蛋白C,快速型 P16419 543.31932+(2.3) 9.9 NLTEEMAVLDETIAK 肌球蛋白重链,成牛骨骼肌 P13538 838.92422+(2.5) 41.1 NSVEIGADPK 肌球蛋白结合蛋白C,快速型 P16419 515.26422+(2.1) 14.5
(续表3)

特征肽段 蛋白来源 UniProt登录号母离子m/z(偏差/ppm)保留时间/min 肉种属SFLEELLTTQCDR 肌球蛋白调节轻链2,骨骼肌亚型 P02609 870.43532+(1.3) 41.6 SPWETEQVDETYVK 肌联蛋白 A6BM71 855.89652+(1.5) 28.8 SSEGNVLQAFK 肌球蛋白结合蛋白C,快速型 P16419 590.30392+(1.6) 25.8 VAGAALPCAPAVK 肌球蛋白结合蛋白C,快速型 P16419 612.84212+(0.7) 22.2 VMIEMDGTENK β-烯醇酶 P07322 633.78942+(0.8) 22.1 VVDVPDPPQSVR 肌球蛋白结合蛋白C,快速型 P16419 654.35132+(1.2) 22.9 VVERPGPPQAVR 肌球蛋白结合蛋白C,快速型 P16419 652.87582+(-0.8) 14.6 ALEDQLSELK 肌球蛋白-1 Q9BE40 573.30612+(0.2) 28.8 牛(Bos taurus)ALPAAAIEGPAYNR 热休克蛋白β-1 A0A1C9EIX3 707.37792+(1.7) 26.6 DLADEVALVDVMEDK L-乳酸脱氢酶A 链 P19858 831.40062+(-0.3) 46.5 DQLIQNLLK L-乳酸脱氢酶A 链 P19858 542.82172+(0) 34.5 EYEATLEECCAK 清蛋白 A0A140T897 653.36172+(1.3) 21.9 EYEATLEECCAK 清蛋白 A0A140T897 751.811462+(-0.1) 16.1 HPSDFGADAQAAMSK 肌红蛋白 A0A1K0FUF3 766.84352+(-1.3) 22.2 HPSDFGADAQAAMSK 肌红蛋白 A0A1K0FUF3 774.84102+(0.80) 15.9 LIAPVAEEETR L-乳酸脱氢酶B 链 Q5E9B1 614.33262+(-0.1) 21.9 LYQGAGGPGAGGFGAQGPK 70 ku 热休克蛋白1A Q27975 845.42082+(-0.3) 24.1 NALAHALQSAR 肌球蛋白-7 Q9BE39 576.31762+(0.3) 20.3 NALLQAEVEELR 肌球蛋白重链15 F1MU39 692.87522+(-0.4) 36.1 SSGTSYPDVLK 阳离子胰蛋白酶 P00760 577.29042+(0.1) 23.2 VGGHAAEYGAEALER 血红蛋白α 亚基 P01966 765.37082+(1.2) 22.1 YLEFISDAIIHVLHAK 肌红蛋白 P02192 935.01712+(0) 46.0 GEPGPAGSVGPAGAVGPR I 型胶原α-2 链 F1SFA7 766.89442+(-1.1) 21.4 猪(Sus scrofa)IVDELTIAESNCPR VI 型胶原α-3 链 Q1T7A4 808.90112+(-0.4) 30.6 TSENAVVVVAGNK 肌球结合蛋白C2 F1RH20 644.34382+(-2.3) 21.4 VGGQAGAHGAEALER 血红蛋白α 亚基 P01965 711.85782+(-1.5) 15.1 TSGGAGGLGPLR 肌间线蛋白 P02540 521.78662+(-0.7) 35.1 HFLEELLTTQCDR 未知蛋白 A0A4X1TZM9 831.39912+(1.2) 49.7 GHHEAELTPLAQSHATK 肌红蛋白 P02189 609.64402+(-0.6) 24.3 AGTPSVPELSSGLELLR 肌小节结合蛋白3 A0A287BPK4 863.47482+(-0.3) 82.6 AVEEVEAPPAAVPK 肌联蛋白X6 亚型 A0A480SN35 703.88142+(0.4) 37.2 AITQDLPGLLGSLGLGK 动力型G 结构域蛋白 A0A4X1VRP4 826.98362+(0.5) 95.8 SGEGKPEGAGELDFSGLLK 未知蛋白 A0A4X1VUZ8 945.97932+(1.3) 49.5 SALAHAVQSSR 肌球蛋白-1 Q9TV61 563.80282+(0.7) 25.6 YGVSPPLVSADVVAK 肌联蛋白 R0K6C9 751.41672+(3.9) 27.6 鸭(Anatinae)VNVADCGAEALAR 血红蛋白β 亚基 P02115 673.33012+(0.3) 24.6 VIILDKPGPPVGPIR 肌联蛋白 R0K6C9 524.32752+(0.4) 27.7 VAGHQEEFGSEALQR 血红蛋白α-D 亚基 P07035 829.40012+(0.9) 19.7 TYFPHFDLSHGSGQIK 血红蛋白α-D 亚基 P07035 917.44962+(1.2) 29.1 DILVAPEIDLDVALR 肌联蛋白 R0K6C9 826.46692+(-0.1) 45.3 LADNLDTLGAAAAK 载脂蛋白A-I O42296 672.36192+(-0.8) 22.6
(续表3)

特征肽段 蛋白来源 UniProt登录号母离子m/z(偏差/ppm)保留时间/min 肉种属TYFPHFDLHPGSEQVR 血红蛋白α-D 亚基 P07035 965.46632+(1.3) 68.4 KVAAALVEAVNHIDDIAGALSK 血红蛋白α-A 亚基 P22740 1103.11602+(0.8) 33.4 VAAALVEAVNHIDDIAGALSK 血红蛋白α-A 亚基 P22740 1039.06852+(-0.4) 93.2 VNVADCGAEALAR 血红蛋白β 亚基 P02115 644.81982+(1.9) 67.2 HGVTVLTQLGK 肌红蛋白 U3IJ58 576.84092+(0.5) 38.1 FISLLDELQK 载脂蛋白A-I O42296 603.34302+(0.7) 26.8 GIPQAAEYQAK 载脂蛋白A-I O42296 588.30692+(-0.8) 57.6 DAIAQFEASAVGK 载脂蛋白A-I O42296 653.83602+(1.6) 50.2 FMSAVAAVLAEK α-D-球蛋白 A0A0U2VAM2 683.35832+(0.3) 25.8
肉类掺假鉴别和筛选用的特征肽段要求不易发生修饰,质谱响应强且易被质谱检测到,色谱保留时间适当,氨基酸数在8~20 之间,热稳定性好,不含半胱氨酸(C)和蛋氨酸(M)的特征肽[14],根据条件最终分别筛选出12 条猪肉、15 条牛肉、19 条鸡肉、16 条鸭肉的特征肽段,各特征肽段的质谱信号较强,峰型较好,图3所示为2 条猪源性特征肽的色谱-质谱图。采用纳升级液相色谱-高分辨质谱法筛选到的特征肽段主要来自肌红蛋白、肌球蛋白、清蛋白、载脂蛋白和血红蛋白等,部分特征肽与文献[2]、[4]、[12]报道的结果一致,部分特征肽为首次鉴别报道,具有物种特异性,可用于肉类掺假鉴别和定量分析不同物种的肉类。

图3 猪源性特征肽SALAHAVQSSR(m/z 563.8)和DLADEVALVDVMEDK(m/z 831.4)的质谱图
Fig.3 Mass spectrometry of porcine marker peptides SALAHAVQSSR (m/z 563.8)and DLADEVALVDVMEDK (m/z 831.4)
2.4 色谱洗脱梯度及蛋白提取液对特征肽检出数和强度的影响
由表4可知,不同梯度的色谱分离对4 种肉类中特征肽的检出数量和信号强度影响不大,然而随着色谱分离时间的延长,部分特征肽的信号强度呈下降趋势,这主要是由于流动相线性变化梯度小、色谱峰展宽所引起。DDA 模式根据设定的阈值,每次只选择超过阈值的1 个母离子进行MS/MS 质谱采集,离子归属清晰,然而质谱仪扫描速度有限,会导致离子漏检。同时,多肽鉴别重复性分析研究[15]也表明,可鉴定出新多肽的速率在2次重复样品注射后急剧增加,而在约5 次重复注射后逐渐减少,这主要是由DDA 采集模式中肽类的半随机取样所引起,试验中采用多次重复技术采样分析法探讨不同洗脱梯度对特征肽检出及信号强度的影响,特征肽段的同位素点积值(idot)均大于0.9,且大部分肽段的idotp 值接近于1.0,与谱图库中的匹配度非常高,可信度高。猪肉源特征肽主要来自于肌球蛋白、肌红蛋白、酶蛋白,牛肉源特征肽主要来自于肌球蛋白、血清白蛋白、酶蛋白、热休克蛋白,鸡肉源特征肽主要来自于肌球结合蛋白C、酶蛋白、肌球调控蛋白,鸭肉源特征肽主要来自于载脂蛋白、血红蛋白亚基、球蛋白,综合考虑特征肽的检出情况与信号强度,以及缩短分析时间,提高检测效率等方面,可选择梯度1(60 min)的色谱分离条件进行肉丸中肉蛋白含量的测定及肉种属鉴定。
表4 色谱洗脱梯度对特征肽检出数和强度的影响
Table 4 Effect of elution gradient on identified number and intensity of marker peptides

种属特征肽序列 蛋白来源 母离子m/z(偏差/ppm) idotp 特征肽母离子信号强度(×107)梯度1 梯度2 梯度3猪 GHHEAELTPLAQSHATK 肌红蛋白 913.96082+(0.5) 0.97--0.54±0.00 HFLEELLTTQCDR 未制蛋白 831.40122+(0.6) 0.98 0.71±0.01 1.40±0.10 8.25±0.13 SALAHAVQSSR 肌球蛋白-1 563.80182+(1.1) 1.00 12.48±0.03 18.52±0.82 28.02±5.13 SGEGKPEGAGELDFSGLLK 未知蛋白 945.97582+(1.2) 0.97--1.40±0.00 TSENAVVVVAGNK 磷酸甘油酸激酶 644.34882+(1.0) 0.98 1.70±0.00 1.80±0.07 2.45±0.10 VGGQAGAHGAEALER 异柠檬酸脱氢酶 711.85782+(1.6) 0.98 1.30±0.00 0.78±0.03 1.70±0.03牛 ALEDQLSELK 肌球蛋白-1 573.30612+(1.0) 1.00 17.54±3.51 20.04±4.27 19.82±2.33 DLADEVALVDVMEDK L-乳酸脱氢酶 831.40062+(0.6) 0.98 0.65±0.01 0.18±0.00 0.24±0.00 DQLIQNLLK 肌球蛋白-1 542.82102+(1.6) 0.97 3.89±0.31 3.37±0.21 3.05±0.11 EYEATLEECCAK 血清白蛋白 751.81052+(0.3) 0.96 1.30±0.95 1.36±0.21 1.06±0.09 HPSDFGADAQAAMSK 肌红蛋白 766.84352+(0.4) 0.98 12.58±0.64 1.41±1.31 9.70±0.33 LIAPVAEEETR L-乳酸脱氢酶 614.33262+(1.2) 0.98-0.48±0.02 0.33±0.05 LYQGAGGPGAGGFGAQGPK 热休克蛋白70 ku 845.42082+(2.6) 0.95-0.44±0.00 0.22±0.00 NALAHALQSAR 肌球蛋白-7 576.31762+(1.9) 0.98 1.16±0.00 1.81±0.43 0.16±0.02鸡 AAENTIVVVAGNK 肌球蛋白结合蛋白C,快速型643.35922+(1.3) 1.00 6.00±0.20 3.70±0.20 3.25±0.15 AAIAQAGYTDK β-烯醇酶 554.78532+(1.1) 1.00 17.03±0.82 14.95±0.02 11.32±1.33 AAPQFLTPLVDR 肌球蛋白结合蛋白C,快速型664.37212+(0.5) 0.91 2.80±0.13 1.80±0.10 1.55±0.15 AVNEMGEATTECR 肌球蛋白结合蛋白C,快速型734.31382+(0.6) 0.97 2.80±0.09 2.20±0.00 1.60±0.00 EGGALPAEVQTR 肌球蛋白结合蛋白C 614.32002+(1.5) 0.97 4.00±0.52 3.00±0.00 2.20±0.00 GLEDQQVVVGDR 肌球蛋白结合蛋白C,快速型657.83602+(0.5) 0.97 5.00±0.26 2.80±0.20 2.70±0.10 LGFSEVEQVQMVVDGVK 肌酸激酶M-型 932.47972+(2.3) 0.91-0.82±0.02 0.29±0.00 LSVEALNSLEGEFK 肌酸激酶M-型 768.40122+(1.3) 0.98 13.04±0.61 10.02±0.03 10.04±0.51 LVVELSDPDLPLK 肌球蛋白结合蛋白C,快速型719.41362+(0.1) 0.91 0.12±0.01 0.78±0.00 0.66±0.04 NLTEEMAVLDETIAK 肌球蛋白重链 838.92422+(0.9) 0.98 22.03±057 9.00±0.00 6.50±0.10 NLTEEMAVLDETIAK 肌球蛋白重链 846.92172+(0.8) 0.98 0.66±0.02--NSVEIGADPK 肌球蛋白结合蛋白C,快速型515.26422+(1.5) 0.98 2.70±0.19 1.65±0.05 1.21±0.29
(续表4)

注:-.未检出。
种属特征肽序列 蛋白来源 母离子m/z(偏差/ppm) idotp 特征肽母离子信号强度(×107)梯度1 梯度2 梯度3 SFLEELLTTQCDR 肌球蛋白轻链2 806.38782+(1.4) 0.99 23.03±1.54 2.55±0.25 2.15±0.45 SSEGNVLQAFK 肌球蛋白结合蛋白C,快速型590.30392+(2.2) 0.96 0.90±0.03 0.51±0.01 0.45±0.01 VAGAALPCAPAVK 肌球蛋白结合蛋白C,快速型612.84212+(0.7) 0.98 4.00±0.05 2.45±0.05 2.05±0.05 VMIEMDGTENK β-烯醇酶 633.78892+(0.8) 0.99 62.04±3.21 44.01±2.03 38.02±0.04 VMIEMDGTENK β-烯醇酶 641.78632+(0.7) 0.96 1.00±0.22 1.20±0.00 0.46±0.00 VMIEMDGTENK β-烯醇酶 641.78632+(0.5) 0.94 1.25±0.09 0.78±0.08 0.60±0.00 VVDVPDPPQSVR 肌球蛋白结合蛋白C,快速型654.35132+(1.1) 0.94 8.20±0.40 4.70±0.30 1.15±0.05 VVERPGPPQAVR 肌球蛋白结合蛋白C,快速型652.87532+(1.7) 0.98 0.74±0.03 0.63±0.01 0.58±0.08鸭 LADNLDTLGAAAAK 载脂蛋白A-I 672.36192+(0) 0.98 2.80±0.37 3.07±0.33 2.17±0.39 TYFPHFDLHPGSEQVR 血红蛋白α-D 亚基 965.46572+(0.6) 1.00 0.31±0.03 0.25±0.02 0.16±0.03 VAAALVEAVNHIDDIAGALSK 血红蛋白α-A 亚基 1039.06802+(0.8) 0.91 0.24±0.00--VAGHQEEFGSEALQR α-D-球蛋白 829.40012+(1.0) 0.98 0.20±0.04 1.93±0.17 1.45±0.07 VNVADCGAEALAR 血红蛋白β 亚基 673.33012+(0.5) 0.90 1.73±0.13 1.50±0.00 1.08±0.17
2.5 非标记定量蛋白组学法检测肉丸中蛋白的含量及鉴定肉种属
非标记定量蛋白组学不需要经过任何标记,直接酶解后经过质谱分析采集数据,利用Proteome Discoverer 软件对谱图进行归一化后,可用肽段离子峰强度或峰面积来鉴定和定量蛋白质,这主要利用了肽段的峰强度或峰面积与其来源的蛋白质的浓度呈正比,且其定量具有重复性和准确性的优点,该方法常用于定量分析肉类样品中蛋白质丰度的变化[16]。采用非标记定量蛋白法鉴别的不同价格牛肉丸样品的结果如图4和图5所示。纯肉和牛肉丸样品的质谱测定结果经PD 软件预处理,导出蛋白的丰度值进行PCA 聚类分析,PCA 属于无监督的判别分析,试验中的前2 个主成分的累计可信度较高,图4a 中横坐标为每个样本的第1 主成分得分值,纵坐标为每个样本的第2 主成分得分值,牛肉和纯牛肉丸样品聚合度很好,紧密的分布在第4 象限,而且不同样品间的差别不大;猪肉、鸡肉、鸭肉样品的聚合度也很好,紧密的分布在第1、2、3 象限,掺有猪肉和鸡肉的牛肉丸样品分别在第1、2 象限均匀分布,部分样品间的聚集度较高,而部分牛肉丸样品在PCA 得分图中的区分效果一般,出现部分混淆情况,这表明PCA 方法可以在很大程度上将数据降维,且降维后的数据能够最大程度地表达原始变量的数据特征而不丢失有效信息,然而对部分样品的区分效果一般。O2PLS-DA 是常用的有监督判别分析,是一种多因变量对多自变量的回归建模方法,在一个算法下实现回归建模(多元线性回归)、数据结构简化(主成分分析)及两组变量之间的相关性分析(典型相关性分析)[17-18]。在PCA 基础上进行O2PLS-DA 分析的纯肉和不同价格牛肉丸样品的结果如图5所示。从图5a 得分图可知,猪肉、牛肉、鸡肉、鸭肉和13 批不同价格的牛肉丸样品可以分成7 大类,纯牛肉丸和掺有猪肉或鸡肉的牛肉丸样品可以很好的根据O2PLS-DA 模型区分开,且PCA 中聚集度较差的样品也能可被分开,13 批样品中共有4 批为纯牛肉丸样品,且在检出的掺有其它肉类的样品中有明确标识的仅占22.2%。由图5b 载荷图可知,每1 个点代表1种潜在蛋白标志物,离中心点和坐标轴越远,对模型的影响越大,即对不同样本的分组贡献率越高。

图4 不同价格牛肉丸样品的蛋白PCA 得分图(a)和载荷图(b)
Fig.4 Protein PCA scores (a) and load (b) of beef meatball samples with different prices

图5 不同价格牛肉丸样品的蛋白O2PLS-DA 得分图(a)和载荷图(b)
Fig.5 Protein O2PLS-DA scores (a) and load (b) of beef meatball samples with different prices
从不同种属的肉类样品中筛选出的特征肽段(如表4) 可作为进一步确证肉制品中肉类的来源,从两种牛肉丸样品中鉴别出猪源和鸡源特征肽的色谱-质谱结果分别如图6和图7所示。牛肉丸样品W1 中共鉴别出猪源特征肽3 条,信号强度(NL)非常大,碎片离子中共测得多个y 离子和b 离子与理论图谱库匹配程度高,这表明在该牛肉丸样品中混有猪肉成分。牛肉丸样品W3 中共鉴别出鸡源特征肽3 条,信号强度(NL)非常大,碎片离子中共测得多个y 离子和b 离子与理论图谱库匹配程度高,这表明在该牛肉丸样品中混有鸡肉成分,因此非标记蛋白定量法结合多元统计分析可用于区分样品中肉的种属与分类,特征肽定量法可用于进一步确认鉴别结果与定量分析。

图6 牛肉丸样品W1 中鉴别出的猪源特征肽色谱-质谱图
Fig.6 Chromatography-mass spectrometry of porcine-specific peptides identified in beef meatball sample W1

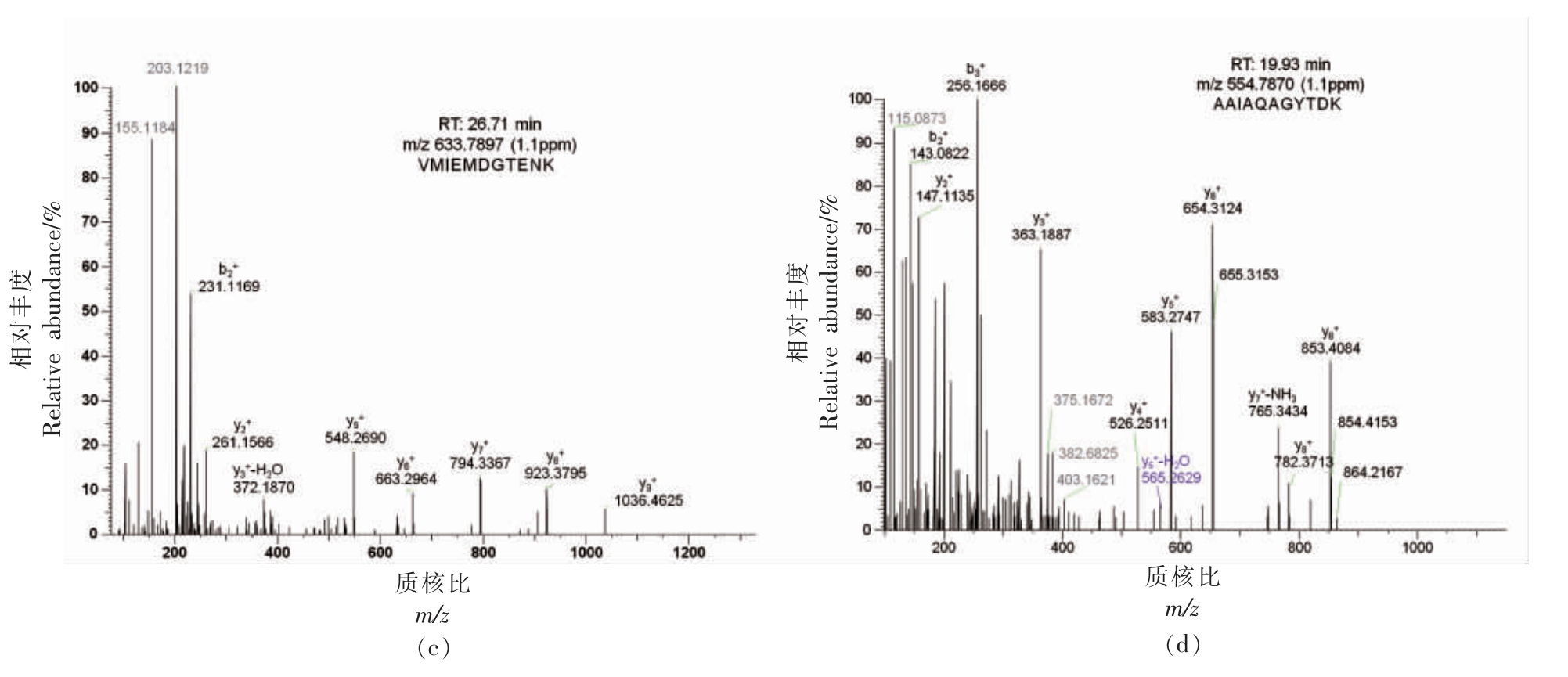
图7 牛肉丸样品W3 中鉴别出的鸡源特征肽色谱-质谱图
Fig.7 Chromatography-mass spectrometry of chicken-specific peptides identified in beef meatball sample W3
3 结论
不同蛋白质提取缓冲液对纯肉和牛肉丸样品中蛋白质的提取效果影响显著(P<0.05),且由于牛肉丸在加工过程中蛋白质的变性程度大,不利于蛋白质的提取,因此在处理牛肉丸样品时需增加搅碎时间和超声强化提取时间。不同液相洗脱梯度对肉中蛋白数和肽段数的鉴定结果有显著影响(P<0.05),在蛋白水平上,DDA 采集模式分别从猪肉、牛肉、鸡肉和鸭肉样品中鉴定出2 715,889,659,1 143 种蛋白质,不同洗脱梯度分离出的特异肽的信号强度分布波动非常小,表明该方法的稳定性和重现性较好。UniProt 蛋白数据库的BLAST算法搜索结果表明,在猪肉、牛肉、鸡肉、鸭肉中分别鉴定出12,15,19,16 条不同种属肉类的特征肽段,不同梯度的色谱分离对4 种肉类特征肽的检出数量和信号强度影响不大,在DDA 采集模式下特征肽的匹配度非常好,可信度高,然而部分特征肽的重现性较差。非标记定量结果表明,纯牛肉丸样品与掺假肉丸样品能很好的鉴别,所测定的牛肉丸样品中有69.2%的样品掺有猪肉或鸡肉,有明确标识的仅占22.2%,O2PLS-DA 作为一种有监督的判别分析方法较PCA 法更适合用于非标蛋白定量法中用于区分牛肉丸样品,特征肽定量法可进一步确认鉴别结果与定量分析。
[1]ORTEA I,O'CONNOR G,MAQUET A,et al.Review on proteomics for food authentication[J].Journal of Proteomics,2016,147(9):212-225.
[2]WATSON A D,GUNNING Y,RIGBY N M,et al.Meat authentication via multiple reaction monitoring mass spectrometry of myoglobin peptides[J].Analytical Chemistry,2015,87(20):10315-10322.
[3]VIDOVA V,SPACIL Z.A review on mass spectrometry-based quantitative proteomics:Targeted and data independent acquisition[J].Analytica Chimica Acta,2017,964(4):7-23.
[4]KORTE R,MONNEUSE J M,GEMROT E,et al.New high-performance liquid chromatography coupled mass spectrometry method for the detection of lobster and shrimp allergens in food samples via multiple reaction monitoring and multiple reaction monitoring cubed[J].Journal of Agricultural and Food Chemistry,2016,64(31):6219-6227.
[5]POLETI M D,MONCAU C T,SILVA-VIGNATO B,et al.Label-free quantitative proteomic analysis reveals muscle contraction and metabolism proteins linked to ultimate pH in bovine skeletal muscle[J].Meat Science,2018,145(11):209-219.
[6]GALLEGO M,MORA L,CONCEPCIÓN ARISTOY M,et al.The use of label-free mass spectrometry for relative quantification of sar coplasmic proteins during the processing of dry-cured ham[J].Food Chemistry,2016,196(4):437-444.
[7]MONTOWSKA M,FORNAL E.Detection of peptide markers of soy,milk and egg white allergenic proteins in poultry products by LC-Q-TOF-MS/MS[J].LWT,2018,87(1):310-317.
[8]NEILSON K A,ALI N A,Muralidharan S,et al.Less label,more free:Approaches in label-free quantitative mass spectrometry[J].Proteomics,2011,11(2):535-553.
[9]PREMANANDH J.Horse meat scandal-A wakeup call for regulatory authorities[J].Food Control,2013,34(2):568-569.
[10]BONNET M,SOULAT J,BONS J,et al.Quantification of biomarkers for beef meat qualities using a combination of parallel reaction monitoring-and antibody-based proteomics[J].Food Chemistry,2020,317(2):126376.
[11]NALAZEK-RUDNICKA K,KŁOSOWSKA-CHOMICZEWSKA I,WASIK A,et al.MRM-MS of marker peptides and their abundance as a tool for authentication of meat species and meat cuts in single-cut meat products[J].Food Chemistry,2019,283(6):367-374.
[12]VON BARGEN C,DOJAHN J,WAIDELICH D,et al.New sensitive high-performance liquid chromatography-tandem mass spectrometry method for the detection of horse and pork in halal beef[J].Journal of Agricultural and Food Chemistry,2013,61(49):11986-11994.
[13]SARAH S A,FARADALILA W N,SALWANI M S,et al.LC-QTOF-MS identification of porcinespecific peptide in heat treated pork identifies candidate markers for meat species determination [J].Food Chemistry,2016,199(10):157-164.
[14]HOFFMANN B,MÜNCH S,SCHWÄGELE F,et al.A sensitive HPLC-MS/MS screening method for the simultaneous detection of lupine,pea,and soy proteins in meat products[J].Food Control,2017,71(1):200-209.
[15]BATEMAN N W,GOULDING S P,SHULMAN N J,et al.Maximizing peptide identification events in proteomic workflows using data-dependent acquisition(DDA)[J].Molecular & Cellular Proteomics,2014,13(1):329-338.
[16]GALLEGO M,MORA L,ARISTOY M C,et al.Optimisation of a simple and reliable label-free methodology for the relative quantitation of raw pork meat proteins[J].Food Chemistry,2015,182(9):74-80.
[17]FONVILLE J M,RICHARDS S E,BARTON R H,et al.The evolution of partial least squares models and related chemometric approaches in metabonomics and metabolic phenotyping[J].Journal of Chemometrics,2010,24(11/12):636-649.
[18]刘晔,王远兴.基于UPLC-QTOF-MS 技术对庐山云雾茶成分分析及真伪判别[J].中国食品学报,2020,20(7):269-277.
LIU Y,WANG Y X.Analysis of Lu Mountain Clouds-Mist tea's composition and authenticity discrimination based on UPLC-QTOF-MS[J].Journal of Chinese Institute of Food Science and Technology,2020,20(7):269-277.