克罗诺杆菌(Cronobacter,原称阪崎肠杆菌)是一种食源性条件致病菌,可导致各年龄段人群感染,尤其是新生儿、早产儿、老年人和免疫力低下的人群。感染后可引起败血症、神经炎、脑脊髓炎、脑脓肿、脑积水和坏死性小肠结肠炎[1-2]。目前,已报道在婴幼儿配方乳粉和谷类辅助食品、奶酪、蔬菜、谷物等食品中检出克罗诺杆菌[3-4]。食品污染克罗诺杆菌后会对人体健康构成危害,然而,不是所有克罗诺杆菌都可导致人患病。研究发现,阪崎克罗诺杆菌(Cronobacter Sakazakii)、丙二酸盐阳性克罗诺杆菌(Cronobacter malonaticus)和苏黎世克罗诺杆菌(Cronobacter turicensis)是引起新生儿和婴儿脑膜炎、败血症和坏死性小肠结肠炎的常见类型[5-7]。摄入被阪崎克罗诺杆菌污染的婴幼儿配方乳粉是导致新生儿和婴儿感染的主要原因[8],感染引起的死亡率为40%~80%[9-10]。对婴儿配方乳粉中阪崎克罗诺杆菌进行检测和追溯,对保障新生儿和婴儿食品安全至关重要。
对致病菌分型是追溯其来源的关键。应用于阪崎克罗诺杆菌的分型方法主要有表型和分子分型,其中分子分型技术被认为是追溯微生物来源的最有力工具。目前已有脉冲场凝胶电泳(Pulse field gel electrophoresis,PFGE)、随机扩增多态性DNA (Random amplified polymorphic DNA,RAPD)、多位点序列分型 (Multilocus sequence typing,MLST)、多位点可变数目串联重复序列分析(Multiple locus variable-number tandem repeat analysis,MLVA)、肠杆菌间保守重复序列PCR(Enterobacterial repetitive intergenic consensus-PCR,ERIC-PCR)和全基因测序(Whole genome sequencing,WGS)等分型方法被用于阪崎克罗诺杆菌分型[6,11],然而,不同方法有着各自的特点和局限。PFGE 分辨率高,是细菌分型的“金标准”,也是流行病爆发调查和食源性病原菌监测中最为常用的方法[12]。然而,PFGE 不能对基因组上缺乏XbaⅠ等限制性内切酶酶切位点的菌株进行分型,也不能用于阪崎克罗诺杆菌鉴定,不能确切反映菌株间的系统发育关系等缺陷[6]。MLST 分型重复性好、分辨率高,可根据fusA 等管家基因对克罗诺杆菌进行鉴定,分型结果也可以上传至MLST数据库(http://pubmlst.org/cronobacter/),进行长期流行学研究,然而,MLST 分型方法不适用于管家基因序列高度一致的菌株[6,11]。ERIC-PCR 具有快速、简便、廉价的特点,可用于管家基因序列相似及不同克罗诺杆菌菌株的流行病学和遗传多样性研究[13]。
分型方法分型能力的高、低对病原菌溯源和流行病学调查有着重要意义。本研究分别采用PFGE、MLST 和ERIC-PCR 分型方法对分离于不同地区,不同品牌婴幼儿配方乳粉、米粉和辅食中的阪崎克罗诺杆菌进行分型,比较3 种分型方法的分型能力,以确定更适于阪崎克罗诺杆菌的分型方法。
1 材料与方法
1.1 菌株来源
供试阪崎克罗诺杆菌共37 株,分离于2010年7月至2012年7月间采集自陕西省宝鸡市、蔡家坡、杨凌、西安市、眉县、岐山县、周至县、扶风县、武功县、兴平市和乾县部分超市、母婴专卖店的婴幼儿配方乳粉、米粉和辅食(表1)。
1.2 试剂与仪器
1.2.1 培养基和试剂 Luria-Bertani(LB)营养琼脂、Luria-Bertani (LB) 肉汤、胰蛋白大豆琼脂(Tryptic Soy Agar,TSA)购自北京陆桥技术股份有限公司。
Taq DNA 聚合酶、ExTaq DNA 聚合酶、dNTPmix、10×PCR 缓冲液、DL2000 DNA Ladder、蛋白酶K(含缓冲液)和限制性内切酶XbaⅠ均购于宝生物工程(大连)有限公司(TaKaRa);DNA 提取试剂盒购于北京康为试剂科技有限公司;十二烷基磺酸钠(SDS)、三羟甲基氨基甲烷盐酸(Tris-HCl)、乙二胺四乙酸 (EDTA)、苯甲基磺酰氟(PMSF) 等均购自Sigma 公司;PFGE 专用低熔点琼脂糖(SeaKem Gold Agarose)购自美国Cambrex Bio Science 公司。
1.2.2 主要仪器与设备 GelDocXR 凝胶成像系统、170-3672 脉冲场凝胶电泳系统、MyCyclerPCR仪,Bio-Rad 公司;超NU-425-400E 净工作台,苏州净化设备有限公司;GNP-9080 隔水式恒温培养箱,北京科伟实验仪器有限公司;MDF-U5411 高压灭菌锅,上海申安高压仪器设备有限公司。
1.3 试验方法
1.3.1 PFGE 分型 PFGE 分型方法主要按照美国疾病预防控制中心 (the Centers for Disease Control and Prevention,CDC)颁布的应用于食源性疾病监测网PulseNet 中的PFGE 标准方法。400 μL 菌悬液中加入20 μL 蛋白酶K 后与400 μL 含SDS 的Seakem Gold 琼脂糖快速混匀、冷却凝固。加入5 mL 细胞裂解缓冲液 (Cell lysis buffer,CLB)和25 μL 蛋白酶K 裂解细菌,于54 ℃、150~175 r/min 恒温水浴摇床中裂解1.5~2 h。用无菌超纯水和TE 缓冲液洗涤裂解细胞后,用200 μL 含有限制性内切酶XbaⅠ及其缓冲液的混合液于37℃恒温水浴中孵育1.5~2 h。吸出酶切混合液,加入200 μL 0.5×TBE 缓冲液,终止反应。PFGE 电泳结束后,溴化乙锭染色30 min;去离子水洗剂2 次,TE 缓冲液洗剂3 次,每次15 min。脱色的琼脂糖凝胶用凝胶成像系统拍照。利用BioNumerics 3.0软件对PFGE 结果进行聚类分析。
1.3.2 MLST 分型 用基因组DNA 提取试剂盒抽提阪崎克罗诺杆菌基因组DNA 作为模板,参考克罗诺杆菌MLST 分型官方数据库(http://pubmlst.org/cronobacter/)公布的阪崎克罗诺杆菌MLST 分型用管家基因atpD、fusA、glnS、gltB、gyrB、infB 和pps 种类、扩增和测序引物序列进行PCR 扩增和测序。PCR 扩增反应总体系为50 μL,包括5 μL 10×Pfu 缓冲液,5 μL dNTP Mix(2 mmol/L),1 μL Pfu DNA 聚合酶,引物各0.5 μL(10 μmol/L),36 μL 超纯水,2 μL 模板DNA;反应条件为94 ℃10 min;94 ℃1 min,60 ℃1 min,72 ℃1 min,35 个循环;72 ℃10 min。除gltB 扩增的退火温度为63℃外,其余6 个基因均为60 ℃。PCR 产物由上海桑尼生物技术有限责任公司纯化后测序,测序结果与克罗诺杆菌PubMLST 数据库(http://pubmlst.org/cronobacter/)比对获得Sequence Type(ST)型。
1.3.3 ERIC-PCR 分型 用1.3.2 节中抽提的基因组DNA 作为模板,上游引物:5'-ATGTAAGCTCCTGGGGATTCAC-3',下游引物:5'-AAGTAAGTGACTGGGGTGAGCG-3',PCR 扩增总体系为25 μL,包括13.15 μL 超纯水,1.5 μL Mg-Cl2(25 mmol/L),2 μL dNTP(2.5 mmol/L),2.5 μL 10×PCR 缓冲液,引物各0.3 μL(50 pmol/L),0.25 μL Taq DNA 聚合酶,5 μL 模板。PCR 扩增条件为95 ℃ 5 min;94 ℃ 45 s,52 ℃ 1 min,72 ℃ 5 min,35 个循环;72 ℃10 min。PCR 扩增后取5 μL PCR 产物进行琼脂糖凝胶电泳,使用凝胶成像系统照相。采用“1”“0”记带法,同一位点有扩增带的记为“1”,无扩增带记为“0”。使用R 软件包(版本3.4.4)和Pheatmap 软件包(版本3.0.1)进行聚类分析。
1.4 辛普森多样性指数(DI 值)
根据辛普森多样性指数方程计算3 种分子分型方法的DI 值,根据DI 值判断分型方法分辨率高低。DI 值越大,分型能力越强[14]。
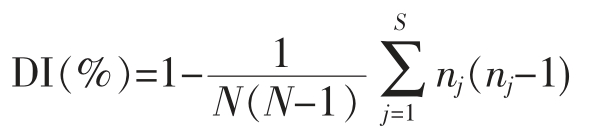
式中,N——菌株总数;S——被描述的类型总数;nj——j 型的菌株总数。
2 结果与分析
2.1 阪崎克罗诺杆菌PFGE 分型
PFGE 分型结果如图1所示。37 株阪崎克罗诺杆菌可被分为35 个PFGE 带型,分别命名为PT01~PT35,其中PT28 包含3 株菌,其余34 个PT型各含1 株菌。菌株PFGE 带谱相似度范围为92%~100%,分型后没有明显的优势带谱。菌株14-4(2)和14-4(1)分离于陕西省兴平市同一品牌的婴幼儿配方乳粉,菌株12-12(2)分离于陕西省西安市的婴儿辅食奶糕粉,3 株菌带谱相同,为PT28 型。菌株15-17(2)、15-8(2)和菌株14-20(2)PFGE 带谱相似性为99%,其中15-17 (2)和15-8(2)来源于同一地区采集的米粉,而14-20(2)来自其它地区的婴幼儿配方奶粉。PFGE 分型方法分型能力较高,可对不同来源的菌株进行相似性和溯源分析。

图1 阪崎克罗诺杆菌PFGE 分型及聚类分析
Fig.1 PFGE typing and cluster analysis of Cronobacter sakazakii
2.2 阪崎克罗诺杆菌MLST 分型
MLST 分型结果表明,37 株菌株可被分为23种ST 型,其中ST1、ST4、ST93 型菌株各有4 株,ST444 型3 株菌,ST12、ST451 和ST449 各2 株,其余16 种ST 型各1 株。23 种ST 型中,有13 个ST型(ST441、ST442、ST443、ST444、ST445、ST446、ST447、ST448、ST449、ST450、ST451、ST452 和ST453)为克罗诺杆菌MLST 数据库没有记录的ST 型。2-9(1)、35-15-1、36-8-1、6-16(1)为与新生儿脑膜炎相关的ST4 型。其中,36-8-1 或6-16(1)分别来源于同一地区的米粉和婴幼儿配方奶粉,2-9(1)、35-15-1 和6-16(1)为来源于不同地区的婴幼儿配方奶粉,且35-15-1 和6-16(1)是为同一品牌。MLST 方法也具有较高的分型能力,可用于追溯食品中阪崎克罗诺杆菌污染源(表1,图2)。
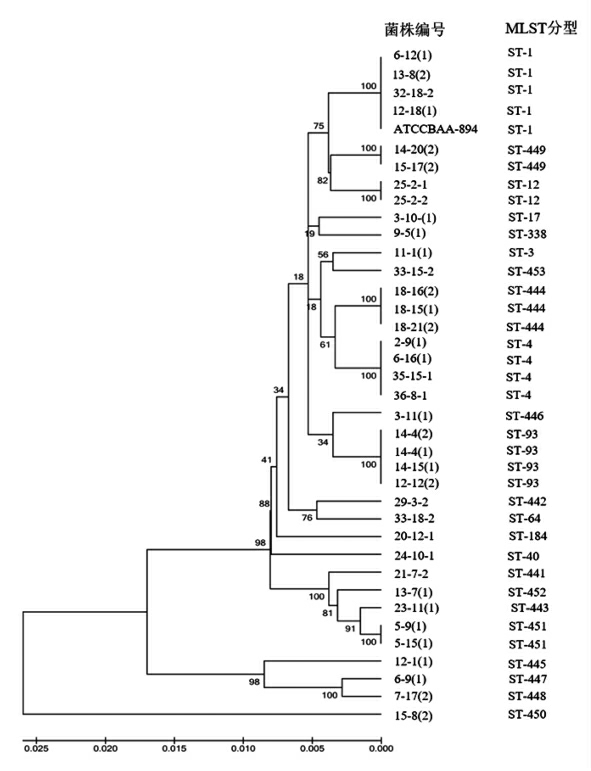
图2 阪崎克罗诺杆菌MLST 分型结果及聚类分析
Fig.2 MLST subtyping and cluster analysis of Cronobacter sakazakii
注:6-12-1 为菌株编号,ST-1 表示该菌株的ST 分型。
表1 阪崎克罗诺杆菌来源及MLST 分型结果
Table 1 The source and MLST sutyping of Cronobacter sakazakii
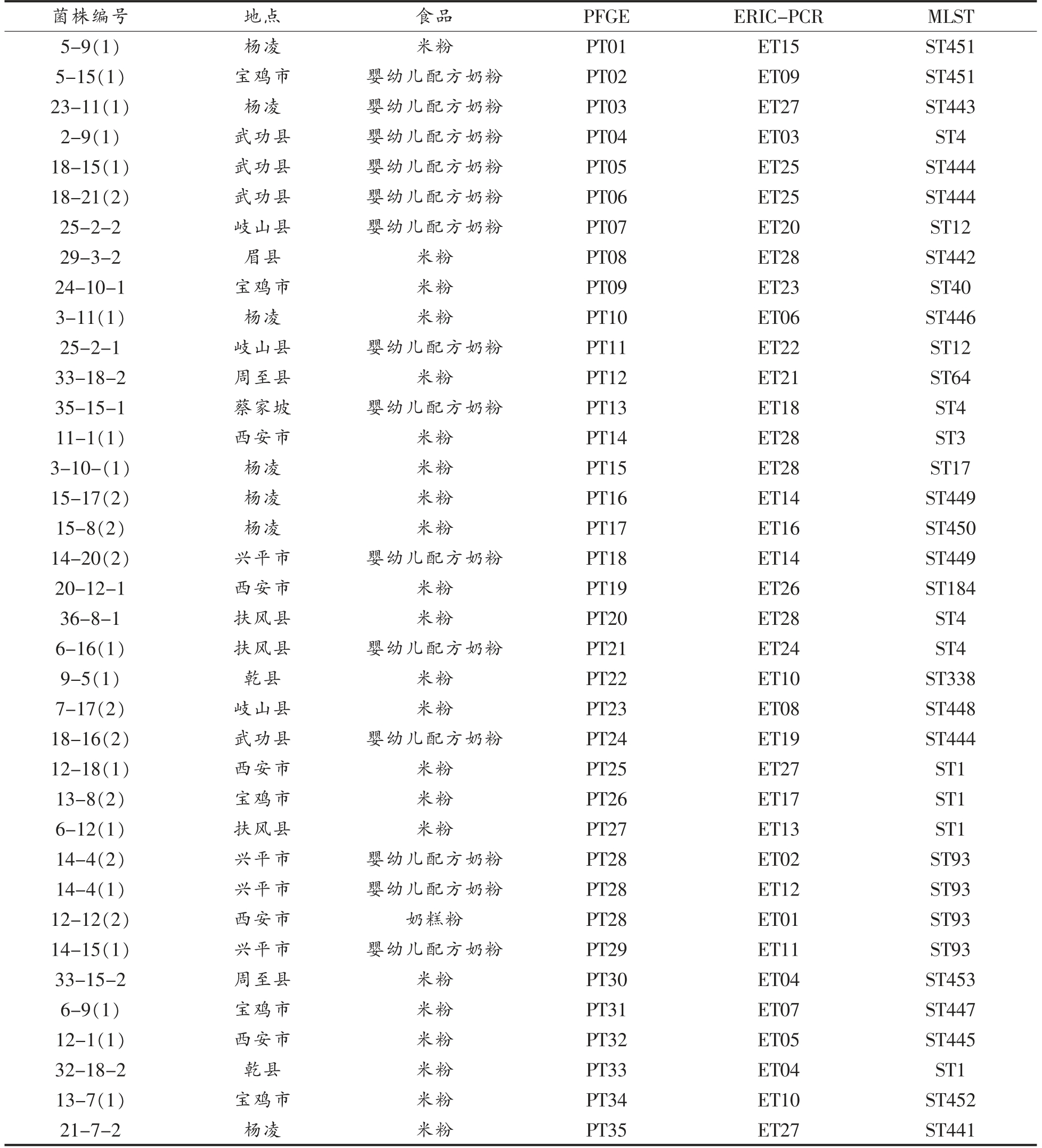
?菌株编号 地点 食品 PFGE ERIC-PCR MLST 5-9(1) 杨凌 米粉 PT01 ET15 ST451 5-15(1) 宝鸡市 婴幼儿配方奶粉 PT02 ET09 ST451 23-11(1) 杨凌 婴幼儿配方奶粉 PT03 ET27 ST443 2-9(1) 武功县 婴幼儿配方奶粉 PT04 ET03 ST4 18-15(1) 武功县 婴幼儿配方奶粉 PT05 ET25 ST444 18-21(2) 武功县 婴幼儿配方奶粉 PT06 ET25 ST444 25-2-2 岐山县 婴幼儿配方奶粉 PT07 ET20 ST12 29-3-2 眉县 米粉 PT08 ET28 ST442 24-10-1 宝鸡市 米粉 PT09 ET23 ST40 3-11(1) 杨凌 米粉 PT10 ET06 ST446 25-2-1 岐山县 婴幼儿配方奶粉 PT11 ET22 ST12 33-18-2 周至县 米粉 PT12 ET21 ST64 35-15-1 蔡家坡 婴幼儿配方奶粉 PT13 ET18 ST4 11-1(1) 西安市 米粉 PT14 ET28 ST3 3-10-(1) 杨凌 米粉 PT15 ET28 ST17 15-17(2) 杨凌 米粉 PT16 ET14 ST449 15-8(2) 杨凌 米粉 PT17 ET16 ST450 14-20(2) 兴平市 婴幼儿配方奶粉 PT18 ET14 ST449 20-12-1 西安市 米粉 PT19 ET26 ST184 36-8-1 扶风县 米粉 PT20 ET28 ST4 6-16(1) 扶风县 婴幼儿配方奶粉 PT21 ET24 ST4 9-5(1) 乾县 米粉 PT22 ET10 ST338 7-17(2) 岐山县 米粉 PT23 ET08 ST448 18-16(2) 武功县 婴幼儿配方奶粉 PT24 ET19 ST444 12-18(1) 西安市 米粉 PT25 ET27 ST1 13-8(2) 宝鸡市 米粉 PT26 ET17 ST1 6-12(1) 扶风县 米粉 PT27 ET13 ST1 14-4(2) 兴平市 婴幼儿配方奶粉 PT28 ET02 ST93 14-4(1) 兴平市 婴幼儿配方奶粉 PT28 ET12 ST93 12-12(2) 西安市 奶糕粉 PT28 ET01 ST93 14-15(1) 兴平市 婴幼儿配方奶粉 PT29 ET11 ST93 33-15-2 周至县 米粉 PT30 ET04 ST453 6-9(1) 宝鸡市 米粉 PT31 ET07 ST447 12-1(1) 西安市 米粉 PT32 ET05 ST445 32-18-2 乾县 米粉 PT33 ET04 ST1 13-7(1) 宝鸡市 米粉 PT34 ET10 ST452 21-7-2 杨凌 米粉 PT35 ET27 ST441
2.3 阪崎克罗诺杆菌ERIC-PCR 分型
ERIC-PCR 分型后,各菌株扩增得到的DNA条带数目为2~6 条,扩增片段长度均在100~2 000 bp 之间。37 株菌株可被分为28 个亚型,分别命名为ET01~ET28。其中,ET28 型有4 株菌,ET27 型有3 株菌,ET4、ET10、ET14 和ET25 各有2 株菌,其余ET 型均有1 株菌。同属ET28 型的4株菌分离于不同地区采集的米粉。23-11(1)、12-18 (1) 和21-7-2 的ERIC-PC 分型结果为ET27型,菌株23-11(1)和21-7-2 分离于陕西杨凌采集婴幼儿配方奶粉和米粉,12-18(1)分离于西安采集米粉。ERIC-PCR 分型结果聚类分析表明,分离于不同地区不同食品的阪崎克罗诺杆菌可能具有相同基因型,可用于阪崎克罗诺杆菌溯源分析。
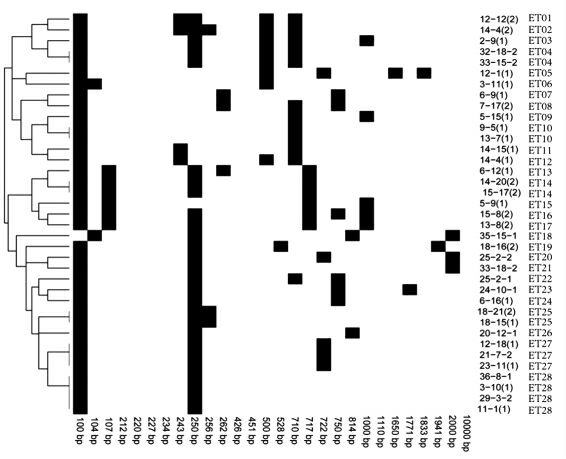
图3 ERIC-PCR 结果聚类分析
Fig.3 Cluster analysis of ERIC-PCR results
2.4 PFGE、MLST 和ERIC-PCR 分型比较
对37 株菌的PFGE 和MLST 分型结果进行比较,以相似度100%为临界值,PFGE 和MLST 的DI 值分别为99.55%和96.40%。DI 值越高,对菌株的鉴别能力越强,因此PFGE 的分型能力高于MLST 分型。总体而言,PFGE 与MLST 分型结果相似,具有相同ST 型菌株的PFGE 分型相似度不低于94%。菌株14-4(2)、14-4(1)和12-12(2)MLST分型为ST93,PFGE 带型为PT28。然而,也有菌株具有相同ST 型而PFGE 分型略有差异的情况,如菌株14-20 (2) 和15-17 (2)MLST 型为ST449,PFGE 图谱相似度为98%;菌株2-9(1)、35-15-1、36-8-1 和6-16(1)为ST4 型,PFGE 条带也存在差异。
以相似性100%为临界值,PFGE 和ERICPCR 分型的DI 值分别为99.55%和98.05%,PFGE对阪崎克罗诺杆菌分辨率略高于ERIC-PCR。ERIC-PCR 分型中菌株32-18-2 和33-15-2 同为ET04 型,而PFGE 分型相似性为92%;菌株15-17(2)与14-20(2)同为ET14 型,PFGE 分型相似性为99%。菌株14-4(2)、14-4(1)和12-12(2)PFGE分型为PT28,ERIC-PCR 电泳图谱存在一定差异,这表明PFGE 和ERIC-PCR 对阪崎克罗诺杆菌均有较高分型能力,2 种方法分型结果略有差异。
MLST 和ERIC-PCR 分别将37 株菌分为23个ST 型和28 个不同的类群,且ERIC-PCR 的DI值大于MLST 分型,表明ERIC-PCR 对阪崎克罗诺菌的分型能力强于MLST 分型。ERIC-PCR 与MLST 分型结果间存在一定的相似性,如菌株18-21(2)和18-15(1)均为ST444,ERIC-PCR 指纹图谱也相同。然而两种分型结果也存在差异,如菌株36-8-1、3-10(1)、29-3-2 和11-1(1)的ERICPCR 分型相似性为100%,而ST 型各不相同。
3 讨论与结论
传统的表型分型方法如血清学分型不利于对菌株进行亲缘性分析和溯源,基于细菌DNA 的分子分型技术则在研究菌株间关系、确定感染源等方面表现出明显的优势,在食品安全检测和病原菌分型研究中起到关键作用。本研究通过比较PFGE、ERIC-PCR 和MLST 3 种分子分型方法对分离于婴幼儿食品源的37 株阪崎克罗诺杆菌的分型能力,以确定3 种分型方法在食源性阪崎克罗诺杆菌风险监测和溯源中的优势和局限。
PFGE 分型是针对菌株基因组,通过不同的限制性内切酶将细菌基因组酶切为不同片段,再外加变化的电场,从而产生不同的基因指纹图谱,以分析菌株相似性和亲缘关系[15]。周厚德等[4]采用PFGE 将24 株来源于市售婴幼儿食品的克罗诺杆菌分成22 个PFGE 基因指纹图谱PFGE 型别高度多样性。张彩霞等[16]对68 株食品源阪崎克罗诺杆菌进行PFGE 分型,可分为58 个PFGE 带型,且带型较分散。本研究的37 株婴幼儿食品源阪崎克罗诺杆可分为35 个PFGE 带型,且DI 值为99.55%,表明PFGE 对阪崎克罗诺杆菌具有较高的分型能力。PFGE 分辨率高的优势,有利于菌株之间的亲缘性分析和溯源,从而快速分析出感染源,减少致病菌造成的危害。然而,由于PFGE具有较低的重现性、无法识别酶切位点外的差异和不具备鉴定菌株的能力[6],因此不利于长期的流行病学监测或研究菌株之间的进化和种系关系。
ERIC-PCR 是以肠杆菌间保守重复序列设计引物,PCR 扩增后产生不同的DNA 图谱,从而对细菌进行分型和遗传分析。Babak 等[17]采用ERICPCR 对从364 个婴幼儿配方奶粉样品中分离的25 株阪崎克罗诺杆菌进行分子分型,菌株被分为8 种不同的类型且具有高度的遗传多样性。Ye等[18]利用ERIC-PCR 分型方法将22 株来源于婴儿配方奶粉的阪崎肠杆菌分为16 个ERIC-PCR指纹图谱,且DI 值为93.3%,表明ERIC-PCR 具有较高的区分能力,并显示了阪崎肠杆菌的遗传多样性,同时作者认为ERIC-PCR 分型方法可用于追踪食物链中阪崎肠杆菌的来源。本研究中37株阪崎克罗诺杆菌具有28 种ERIC-PCR 指纹图谱,DI 值为98.05%,这也表明ERIC-PCR 同样具有较强的分型能力。此外,ERIC-PCR 具有快捷、简便等优势可使其迅速分析菌株之间遗传多样性和亲缘性,从而可快速展开流行病学调查,然而PCR 扩增结果影响因素多,需不断优化试验。
MLST 是通过扩增菌株的管家基因,比较管家基因的核苷酸序列差异从而对菌株进行分型,已广泛用于即食食品、婴幼儿配方奶粉、奶粉等食品中分离阪崎克罗诺杆菌分型[19-20]。同时,已有研究发现阪崎克罗诺杆菌ST 型与特定感染相关,如ST4 型与新生儿脑膜炎有关[21]。本研究中37 株阪崎克罗诺杆菌具有23 个ST 型,DI 值为96.04%,表明MLST 同样具有较高的分辨率,其中有4 株阪崎克罗诺杆菌为ST4 型,对婴幼儿健康造成威胁。MLST 分型可用于评估菌株潜在致病性。此外,已建立在线数据库可实现数据的全球共享和比对,有利于长期流行病学调查。
PFEG、MLST 和ERIC-PCR 3 种分子分型方法均有较高的分型能力,已有文献比较这3 种分型方法对空肠弯曲菌和大肠杆菌的分型能力,结果也表明3 种方法均有高度的辨识度,其中PFGE分辨率最高,其次是ERIC-PCR 和MLST[22]。然而,目前未有文献比较这3 种分型方法对阪崎克罗诺杆菌的分型能力。本研究结果表明PFGE 对阪崎克罗诺杆菌的分型能力最强,而MLST 分型的能力相对最弱,这与已报道PFGE 分型方法对阪崎克罗诺杆菌、阪崎肠杆菌、气单胞菌、大肠埃希菌、空肠弯曲菌、沙门氏菌、李斯特菌和铜绿假单胞菌等菌株的分型能力均略强于ERIC-PCR 结果一致[23-28]。此外,目前虽尚未有研究比较MSLT 和ERIC-PCR 2 种分型方法对阪崎克罗诺杆菌的分型效果,但ERIC-PCR 对肺炎克雷伯菌、空肠弯曲菌等致病菌的分型能力高于MLST[29-30]。根据对菌株的分型能力而言,3 种分型方法中PFGE 分辨率最高。
细菌分型在食品安全监控和流行病学调查中发挥着重要作用,传统的表型方法和基于特定基因序列的分子分型方法已不能满足对菌株的精准溯源分析。基于细菌基因组分型技术可进一步对菌株进行精准分型,且具有重复性好、稳定等优点,同时随着基因组测序技术的发展以及测序费用降低,该技术可在风险监测和溯源中广泛应用。
[1]STRYSKO J,COPE J R,MARTIN H,et al.Food safety and invasive cronobacter infections during early infancy,1961-2018[J].Emerging Infectious Diseases,2020,26(5):857-865.
[2]韩冉.阪崎克罗诺杆菌间毒力比较与致病因子研究[D].天津:天津科技大学,2014.
HAN R.Virulence comparison and pathogenic factors screening of Cronobacter sakazakii[D].Tianjin:Tianjin University of Science and Technology,2014.
[3]JARADAT Z W,AL MOUSA W,ELBETIEHA A,et al.Cronobacter spp.-opportunistic food-borne pathogens.A review of their virulence and environmental-adaptive traits[J].Journal of Medical Microbiology,2014,63(8):1023-1037.
[4]周厚德,彭思露,刘成伟,等.2018年江西省婴幼儿食品中克罗诺杆菌污染状况及分子分型和耐药特征分析[J].中国食品卫生杂志,2019,31(4):335-339.
ZHOU H D,PENG S L,LIU C W,et al.Analysis of the contamination,molecular typing and drug resistance of Cronobacter in infants and young children foods in Jiangxi province in 2018[J].Chinese Journal of Food Hygiene,2019,31(4):335-339.
[5]刘咪,杨保伟,夏效东,等.阪崎克罗诺肠杆菌致病性机理研究进 展[J].食品 科学,2014,35(9):329-333.
LIU M,YANG B W,XIA X D,et al.Progress in understanding the pathogenic mechanism of Cronobacter sakazakii[J].Food Science,2014,35(9):329-333.
[6]费鹏,杨同香,姜亦超,等.克罗诺杆菌分型技术研究进展[J].食品科学,2017,38(21):308-312.
FEI P,YANG T X,JIANG Y C,et al.Progress in typing methods for Cronobacter spp.[J].Food Science,2017,38(21):308-312.
[7]CHAUHAN R,SINGH N,PAL G K,et al.Trending biocontrol strategies against Cronobacter sakazakii:A recent updated review[J].Food Research International,2020,137:109385.
[8]YAN Q Q,CONDELL O,POWER K,et al.Cronobacter species (formerly known as Enterobacter sakazakii) in powdered infant formula:A review of our current understanding of the biology of this bacterium[J].Journal of Applied Microbiology,2012,113(1):1-15.
[9]FORSYTHE S J.Updates on the Cronobacter genus[J].Annual Review of Food Science and Technology,2018,9(1):23-44.
[10]FRIEDEMANN M.Epidemiology of invasive neonatal Cronobacter (Enterobacter sakazakii) infections[J].European Journal of Clinical Microbiology&Infectious Diseases,2009,28(11):1297-1304.
[11]田雪,王娉,陈颖,等.克罗诺杆菌 (阪崎肠杆菌)分子分型技术研究进展[J].卫生研究,2014,43(5):862-866.
TIAN X,WANG P,CHEN Y,et al.Progress in molecular typing for Cronobacter spp.(Enterobacter sakazakii)[J].Journal of Hygiene Research,2014,43(5):862-866.
[12]MELLES D C,VAN LEEUWEN W B,SNIJDERS S V,et al.Comparison of multilocus sequence typing (MLST),pulsed-field gel electrophoresis(PFGE),and amplified fragment length polymorphism (AFLP) for genetic typing of Staphylococcus aureus[J].J Microbiol Methods,2007,69(2):371-375.
[13]AWADALLAH M A I,AHMED H A,MERWAD A M A,et al.Molecular characterization of Cronobacter sakazakii in Egypt,survival and thermoresistance at different temperatures:A potential public health risk[J].Vector Borne&Zoonotic Diseases,2018,18(2):101-107.
[14]HUNTER P.Numerical index of the discriminatory ability of typing systems:An application of Simpson's index of diversity[J].Journal of Clinical Microbiology,1988,26(11):2465-2466.
[15]何卓琳,唐敏嘉,蒲万霞.大肠杆菌分子分型方法研究进展[J].中国草 食动物 科学,2021,41(5):57-60.
HE Z L,TANG M J,PU W X.Advance in molecular typing of Eschericia coli [J].China Herbivore Science,2021,41(5):57-60.
[16]张彩霞,陈颖,胡安妥,等.食品中阪崎克罗诺杆菌的药物敏感性及分子分型研究[J].中国农业科技导报,2020,22(6):123-129.
ZHANG C X,CHEN Y,HU A T,et al.Drug sensitivity analysis and molecular typing of Cronobacter sakazaakii isolated from food[J].Journal of Agricultural Science and Technology,2020,22(6):123-129.
[17]BABAK P,WOLFRAM M B,SAMANEH A,et al.Antibiotic resistance and molecular characterization of Cronobacter sakazakii strains isolated from powdered infant formula milk[J].Foods,2020,11(8):1093.
[18]YE Y,WU Q,XU X,et al.The phenotypic and genotypic characterization of Enterobacter sakazakii strains from infant formula milk[J].Journal of Dairy Science,2010,93(6):2315-2320.
[19]陈启明,刘战民,陆兆新.克罗诺杆菌检测方法的研究进展[J].食品安全质量检测学报,2020,11(24):9281-9287.
CHEN Q M,LIU Z M,LU Z X,et al.Research progress of detection methods of Cronobacter spp.[J].Journal of Food Safety&Quality,2020,11(24):9281-9287.
[20]FEI P,MAN C X,LOU B B,et al.Genotyping and source tracking of Cronobacter sakazakii and C.malonaticus isolates from powdered infant formula and an infant formula production factory in China[J].Applied&Environmental Microbiology,2015,81(16):5430-5439.
[21]JOSEPH S,FORSYTHE S J.Predominance of Cronobacter sakazakii sequence type 4 in neonatal infections[J].Emerging Infectious Diseases,2011,17(9):1713-1715.
[22]WARDAK S,JAGIELSKI M.Evaluation of genotypic and phenotypic methods for the differentiation of Campylobacter jejuni and Campylobacter coli clinical isolates from Poland.II.PFGE,ERIC-PCR,PCRflaA-RFLP and MLST[J].Medycyna Dowiadczalna I Mikrobiologia,2009,61(1):63-77.
[23]张博.源于羊奶粉生产过程各工序的阪崎克罗诺杆菌基因分型研究[D].杨凌:西北农林科技大学,2017.
ZHANG B.Study on genotyping of Cronobacter Sakazakll in production processes of infant formula goat milk powder factories[D].Yangling:Northwest A&F University,2017.
[24]PROUDY I,BOUGLE D,COTON E,et al.Genotypic characterization of Enterobacter sakazakii isolates by PFGE,BOX-PCR and sequencing of the fliC gene[J].Journal of Applied Microbiology,2010,104(1):26-34.
[25]黄玉萍,邓玉婷,姜兰,等.复合水产养殖环境中气单胞菌耐药性及其同源性分析[J].中国水产科学,2014(4):777-785.
HUANG Y P,DENG Y T,JIANG L,et al.Antimicrobial resistance and homology analysis in Aeromonas isolated from integrated fish farms [J].Journal of Fishery Sciences of China,2014 (4):777-785.
[26]CASAREZ E A,PILLAI S D,DI GIOVANNI G D.Genotype diversity of Escherichia coli isolates in natural waters determined by PFGE and ERIC-PCR[J].Water Research,2007,41(16):3643-3648.
[27]LONDERO A,COSTA M,SUCARI A,et al.Comparison of three molecular subtyping techniques for Listeria monocytogenes[J].Revista Argentina De Microbiologia,2019,51(4):359-362.
[28]SILBERT S,PFALLER M A,HOLLIS R J,et al.Evaluation of three molecular typing techniques for nonfermentative Gram-negative bacilli [J].Infection Control&Hospital Epidemiology,2004,25(10):847-851.
[29]李丽娟,陈丹娜,谢小燕,等.广东省东莞市56 株高毒力肺炎克雷伯菌感染分布及分子流行病学研究[J].中国热带医学,2020,20(10):967-971,983.
LI L J,CHEN D N,XIE X Y,et al.Distribution and molecular epidemiology of 56 strains of hypervirulent Klebsiella pneumoniae in Dongguan,Guangdong[J].China Tropical Medicine,2020,20(10):967-971,983.
[30]王寒.四川部分地区禽源空肠弯曲菌的分离鉴定、分子分型及毒力相关基因检测[D].雅安:四川农业大学,2017.
WANG H.Isolation,identification,molecular typing and virulence related gene detection of Campylobacter jeiuni isolated poultry in some areas of Sichuan province[D].Yanan:Sichuan Agricultural University,2017.