在追求美好生活的新时代,人们对于食品的需求已经从“保障供给”向“营养健康”转变[1]。今年“两会”期间,习总书记指出:“发展生物科技、生物产业,向动物、植物、微生物要热量、要蛋白”。蛋白质作为生命活动的主要承担者,是人体健康与生长发育最重要的营养素[2]。长期以来,人们主要通过畜牧业获取肉、蛋、奶等产品作为蛋白质的主要来源[3]。然而,畜牧业占用了全球约1/3 的土地和约1/4 的淡水,并释放了约15%的温室气体,难以满足人们日益增长的蛋白质需求[4]。随着合成生物学、代谢工程等新兴生物技术的发展,对微生物细胞进行理性设计与改造,构建微生物细胞工厂,利用可再生资源生产氨基酸,实现了向微生物要氨基酸[5-7],合成单细胞蛋白实现了向微生物要蛋白[8-9]。
氮代谢不仅是微生物细胞的基本代谢过程,也是氨基酸和蛋白质生物合成的关键瓶颈[10-11]。在微生物细胞中,NH4+在谷氨酸脱氢酶与谷氨酰胺合成酶等相关酶的作用下合成谷氨酸和谷氨酰胺,经转氨作用合成其它氨基酸,掺入蛋白质合成[12-13]。本文重点关注铵的生物合成途径,即向自然界要铵;介绍利用铵合成蛋白质减量替代的氨基酸,即向微生物要氨基酸;利用氨基酸合成蛋白质,即向微生物要蛋白,并展望未来利用微生物细胞工厂生产氨基酸与蛋白质的发展方向。
1 微生物利用氮源生产铵盐
氮元素是生物合成蛋白质、核酸的关键元素[10]。氮在自然界中以气态氮(N2、NO、N2O)、硝态氮(NO3-、NO2-)、联氨(N2H4)、羟胺(NH2OH)等形式存在,微生物通过生物固氮、硝酸盐同化等氮代谢途径,将自然界中不同形式的氮转变为NH4+,从而掺入蛋白质、核酸等有机氮的合成中(图1)。
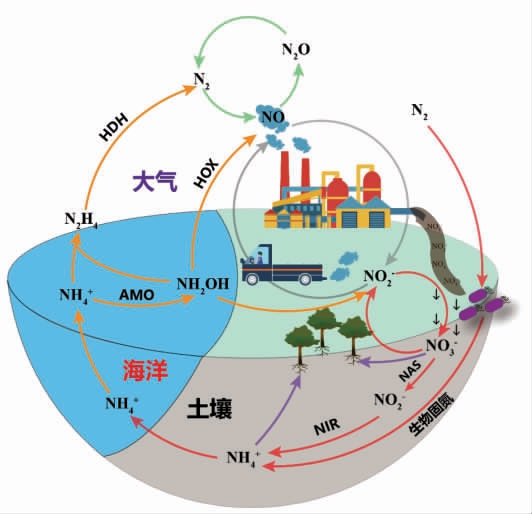
图1 自然界中的氮循环
Fig.1 Schematic diagram of the nitrogen cycle in nature
1.1 微生物利用氮气生成铵盐
固氮生物将空气中氮气还原为氨的过程,称为生物固氮,每年固定的氮素量约占全球作物所需氮量的75%[14]。生物固氮依赖于固氮酶,在生物固氮过程中,铁蛋白将电子传递给钼铁蛋白,钼铁蛋白再将电子传递给N2 和质子,生成NH3 和H2[15-16]。提高微生物固氮效率的关键在于:在固氮微生物中提高固氮酶活性[17],在非固氮微生物中引入固氮酶[18]。
通过对比分析5 种来源的固氮酶基因,发现棕色固氮菌(Azotobacter vinelandii)钼依赖型固氮酶是由14 个基因组成,位于基因组上2 个未连接区 域:包 含nifHDKTY、nifENX、nifSUVP、nifWZ、nifM 和nifF 的29 kb 的区域以及包含nifBQ 和nifAL 的6 kb 的区域[15],其中nifH 是检测环境中固氮微生物的标记基因,nifDK 编码活性中心含钼的催化部分[19],nifH、nifEN、nifQ、nifV 和nifB 负责铁钼辅因子(FeMoco)的生物合成,nifF 编码黄素氧还原蛋白,nifUS 参与Fe-S 簇的形成,nifAL 则是调控基因转录水平的调控基因[20]。在解析固氮酶基因功能的基础上,发展了一系列提高固氮酶活性的方法,如改造转录因子nifL 和nifA。棕色固氮菌固氮基因簇在转录水平上受双组份调控因子nifLA 调控,敲除nifL 或者强化nifA 的表达,能有效增加胞内nifH 的mRNA 水平,增强固氮酶活性,使培养液中铵浓度增加到35 mmol/L[20]。另一个例子是在褐球固氮菌(Azotobacter chroococcum)中,利用组成型启动子控制正调控因子NifA表达,并通过部分敲除失活负调控因子NifL,提高固氮酶活性,将工程菌株应用于小麦的生产,使小麦产量增加60%[17]。
如何借助合成生物学的方法,将固氮途径引入非固氮微生物,提高非固氮微生物的固氮能力成为研究热点。1972年,科研人员首次将肺炎克雷伯氏菌(Klebsiella pneumoniae)的固氮基因组簇nif 克隆到大肠杆菌(Escherichia coli),获得具有生物固氮能力的大肠杆菌[21]。由于存在固氮酶活性低、能量供应不足等问题,因此MIT 的Christopher A Voigt 团队将产酸克雷伯氏杆菌(Klebsiella oxytoca)固氮酶系统的16 个基因分为103 个部分,通过改变启动子占有率、基因顺序和基因方向等方法,在大肠杆菌中从头设计了122个突变体,并根据设计-构建-检测-学习循环优化了固氮酶系统,最终使其固氮效率达到对照产酸克雷伯氏杆菌的57%[7]。微生物固氮酶系统每固定一个氮气分子需要消耗16 个ATP 和8 个高能电子[22],造成微生物细胞内能量供应严重不足。为此,将来源于类芽孢杆菌WLY78(Paenibacillus sp.WLY78)的固氮酶导入大肠杆菌,其固氮酶活性仅为原菌株的10%。在大肠杆菌中将来源于类芽孢杆菌WLY78 的电子传递链蛋白(pfoABfldA)和产酸克雷伯氏杆菌来源的铁-硫原子簇蛋白(nifSU)进行重组,使固氮酶的活性提高了5 倍[23]。为了进一步强化固氮酶系统的能量供给,将固氮酶系统分为电子传递链模块、金属原子簇模块及固氮酶核心模块3 个模块。针对电子传递链模块,将固氮酶系统中负责电子传递的模块替换植物叶绿体的电子传递链模块,同时将固氮酶系统电子传递链与线粒体来源的电子传递链组成功能性杂合的电子传递链模块,均有效提高了能量供应[18]。
1.2 微生物利用硝态氮生成铵盐
微生物利用硝酸盐的途径主要包括硝酸盐同化途径和硝酸盐异化还原途径,将硝酸盐还原为亚硝酸盐。硝酸盐同化途径主要存在于藻类、蓝细菌和植物中,其过程是硝酸盐在ATP 依赖的硝酸盐转运蛋白的作用下转运到细胞质中,再由细胞质中的硝酸盐还原酶(NAS)还原为亚硝酸盐,亚硝酸盐通过电子传递链进一步被还原为铵[24-25]。然而,在富铵环境下,NAS 的表达会受到显著抑制。若NAS 在其它细胞器中,则不存在NAS 表达受到抑制这个问题,如:在结核分枝杆菌(Mycobacterium tuberculosis)中发现了膜锚定硝酸盐还原酶系统(NAR),可以在周质空间中进行硝酸盐的高效同化[26]。硝酸盐异化还原途径主要存在兼性厌氧菌中,硝酸盐作为呼吸链的最终氢受体,被还原为亚硝酸盐,从而用于呼吸作用。膜锚定硝酸盐还原酶系统(NAR) 和周质空间硝酸盐还原酶系统(NAP)都可以催化硝酸盐异化还原反应[24]。有研究发现,脱氮副球菌(Paracoccus denitrifican)同时具有NAR 和NAP 两种酶系统[24]。NAR 系统在细胞质中还原硝酸盐,并将质子释放进细胞周质中,产生的质子驱动力直接促进能量的生成;NAP 系统还原硝酸盐为亚硝酸盐的过程发生在细胞质周质中,不会发生质子转移。硝酸盐异化还原途径可与甲烷、硫化物和氢气等电子供体的氧化耦合生成亚硝酸盐。
微生物细胞能将亚硝酸盐还原为铵和一氧化氮。由于还原为铵对微生物生长具有促进作用,因此它是微生物利用亚硝酸盐的主要途径。亚硝酸盐异化还原为铵是硝酸盐异化还原为铵(DNRA)途径(NO3-→NO2-→NH4+)的关键反应,当环境中的铁、氢气、硫化物等和硝酸盐相关的电子供体大量存在时,微生物可以利用DNRA 作用耦合电子供体的氧化来生长[27-28]。这一过程由位于周质空间中细胞色素c 亚硝酸盐还原酶(ccNIR)、八面体血红素亚硝酸盐还原酶(ONR)[29]和八面体血红素连四硫酸盐还原酶(OTR)[30] 3 个酶所催化。此外,在胎儿弯曲菌(Campylobacter fetus)中,发现只含羟胺氧化酶(HAO)而不含亚硝酸盐还原酶,也可以将亚硝酸盐还原为铵,推测这一反应是由HAO 催化[31]。
1.3 微生物利用其它形式氮生产铵盐
一氧化氮、一氧化二氮、羟胺和联氨等形式的氮是氮循环的重要组成部分,微生物可以通过多种氮代谢途径将这些形式的氮转变为氮气,再利用生物固氮过程将这些形式的氮转变铵:1) 一氧化氮被一氧化氮还原酶还原为一氧化二氮和氮气,一氧化氮还原酶以多种形式存在于多种微生物中,如在脱硫弧菌(Desulfovibrio gigas)中是黄铜二铁-一氧化氮还原酶[32],在大肠杆菌中是杂合蛋白HCP[33],在尖孢镰刀菌(Fusarium oxysporum)中是NADH 依赖型黄素蛋白P450[34]。2)羟胺氧化还原酶(HAO)将羟胺转化为一氧化氮,再借助一氧化氮转化途径和固氮途径生成铵盐。在消化螺菌(Nitrospira spp)及其它氨氧化细菌中,发现10余种羟胺还原酶类似蛋白[35],其中一种羟胺氧化酶[36](HOX)可催化羟胺氧化为一氧化氮。然而,最近研究表明,HAO 并非将羟胺直接催化为一氧化氮,而是将其催化为亚硝酸盐,而后在未知酶的作用下生成一氧化氮[37]。3)联氨脱氢酶(HDH)将联氨氧化成氮气[38-39],氮气在微生物固氮途径的作用下生成铵盐。进一步分析HDH 的氨基酸序列发现,其与羟胺氧化还原酶(HAO) 和羟胺氧化酶(HOX)具有相似的序列[39]。
2 微生物生产饲用氨基酸
随着人民生活水平不断提高,动物产品的人均消费量逐年增加,2021年我国人均肉类、禽蛋、奶类消费量分别为70,24,42 kg,导致2021年我国养殖业消耗4.5 亿t 饲料,其中豆粕(蛋白)消耗量为6 800 万t,占比15.3%;导致2021年我国大豆进口量为9 652 万t,对外依存度为85.66%,严重威胁养殖行业的健康发展。在中美贸易摩擦的背景下,如何减少豆粕使用,对保障养殖行业健康发展和人民生活水平提高具有重要意义。2021年3月,农业农村部推荐减少豆粕用量的重要举措之一是:在饲粮中补充必需氨基酸,降低动物日料中蛋白质水平。小品种氨基酸的添加能有效提高动物生长性能,改善肠道微生物,提高免疫力、减缓应激,在降本增效中发挥着重要作用。
2.1 饲用氨基酸高产菌株构建技术
基于基因-蛋白-生化反应以及组学数据,将细胞代谢网络和调控网络标准化、数字化、模型化,搭建了基因组规模代谢网络模型(GSMM)[40],可在计算机中模拟细胞代谢,预测氨基酸的最佳合成途径,设计菌种改造策略,指导氨基酸高产菌株构建[41]。如在大肠杆菌代谢模型iML1515[42]的基础上,利用GECKO 方法[43]整合酶学动力参数kcat值,构建大肠杆菌酶约束模型(ec_iML1515)。利用酶约束模型ec_iML1515,预测赖氨酸合成过程中的关键靶点,发现前体积累、产物合成路径和能量供给是制约赖氨酸合成的关键靶点。在此基础上。利用模块化工程策略对预测的关键靶点进行调控,使赖氨酸产量增加到193.6 g/L[41](图2a)。
为了平衡氨基酸高产菌株中代谢流分配,研究人员建立了一系列代谢流静态调控方法,实现氨基酸生产菌株的代谢流精细调控。例如,采用启动子工程平衡L-色氨酸前体磷酸烯醇式丙酮酸、赤藓糖-4-磷酸和丝氨酸的供给,将L-色氨酸产量提高到52.1 g/L[5]。为了同时调控多个基因表达,研究人员开发了具有切割DNA 和RNA 的双重功能的CRISPR/Cpf1,Cpf1 可以切割gRNA 阵列使其成为单个成熟的gRNA,同时靶向多个靶基因[44]。利用DNA 切割功能失活的dCpf1 和gRNA阵列,将赖氨酸合成途径中的柠檬酸合酶(gltA)、磷酸烯醇式丙酮酸羧激酶(pck)、葡萄糖-6-磷酸异构酶(pgi)和高丝氨酸脱氢酶(hom)同时弱化不低于90%,将赖氨酸产量提高了4 倍[44]。然而,静态调控策略会造成细胞代谢负担,引起微生物细胞生长缓慢与代谢失衡等[45-46]。为此,开发了一系列基于生物传感器的动态调控策略:物理信号(光、温度)、化学信号(pH 值、溶氧)和生物信号(细胞生长浓度)[45-46]。例如,在大肠杆菌中利用温度敏感型元件(cIts-pR-pL)和调节蛋白(tetRPLtetO-1),开发了基于温度敏感型的动态调控技术,动态调节丙酮酸羧化酶(PYC),平衡代谢流在细胞生长和苏氨酸合成之间的平布。在发酵起始阶段,通过温度控制关闭PYC 表达,使代谢流进入TCA 循环用于生长;当细胞生长到一定阶段,通过升高温度开启PYC 表达,使代谢流经丙酮酸羧化为草酰乙酸用于苏氨酸合成,最终使L-苏氨酸产率提高到124.03%[47](图2a)。此外,由于氨基酸高产菌株复杂的代谢调控网络,仅依靠已知靶点的叠加调控,无法实现目标氨基酸代谢流的最大化。为此,在蛋氨酸合成菌株中,建立了包含中心代谢路径和氨基酸合成路径中80 个候选靶点的基因库,并利用CRISPRi 技术对候选靶点基因进行调控,获得影响蛋氨酸合成的关键靶点。根据模块化工程策略,将蛋氨酸生物合成途径分为:碳模块、硫模块和一碳模块,并利用CRISPRi 技术对蛋氨酸合成模块进行多层次调控,最终使蛋氨酸的产量提升到16.86 g/L[48]。
2.2 饲用氨基酸高产菌株高通量筛选技术
为了快速获得生产性能显著提升的氨基酸高产菌株,研究人员开发了与目标氨基酸智能适配的生物传感器,将胞内或胞外的氨基酸浓度信号转化为易于检测的荧光信号[49]或细胞生长信号[50],从而提高高产菌种筛选效率。如在L-赖氨酸高产菌株筛选中,构建了由响应赖氨酸的启动子元件pA 和绿色荧光蛋白(GFP)组成的表达元件pAG,将胞内赖氨酸浓度转化为易于检测的绿色荧光蛋白信号。利用pAG 元件和绿色荧光蛋白强度的变化,从1 000 万个突变库中筛选到186 株有益突变体,进一步通过96 孔板筛选到大肠杆菌MU-1和大肠杆菌MU-2 菌株,L-赖氨酸产量达到136.51 g/L 和133.29 g/L[49]。由于基于RNA 适配器具有折叠复杂结构的灵活性,开发了响应L-色氨酸的RNA 适配器,并利用黄色荧光蛋白建立了高产L-色氨酸菌株的高通量筛选平台,成功获得1 株L-色氨酸产量提高165.9%的高产菌株大肠杆菌K3mu1[51]。此外,研究人员根据培养环境氨基酸匮乏时,稀有密码子tRNAs 无法完全翻译的“密码子偏好性”规律,建立了基于富含稀有密码子的氨基酸高产菌株筛选系统,该系统通过将报告蛋白或耐药标记中的密码子替换为稀有密码子,提高蛋白翻译过程中对胞内氨基酸的“门槛值”,实现了亮氨酸、精氨酸和丝氨酸等氨基酸高产菌株的高通量筛选[50](图2b)。
2.3 饲用氨基酸高产菌株环境适应性提升技术
提升高产菌种对工业环境的环境适应性,能显著提高菌种生产性能[52]。利用诱变育种与适应性进化策略,获得具有优良生产性状的氨基酸高产菌株,再通过反向代谢工程策略解析耐受机制,指导高产菌株的选育与构建[52]。例如,通过常压室温等离子体(ARTP)诱变L-苏氨酸生产菌株大肠杆菌TRFC,获得了抗噬菌体的L-苏氨酸高产菌株大肠杆菌TRFC-AP,使L-苏氨酸产量增加到159.3 g/L(提高了10.9%)。对高产菌株大肠杆菌TRFC-AP 的基因分析,发现噬菌体侵染基因(fhuA)的缺失是高产菌株产生噬菌体抗性的根本原因[53]。为了进一步提高氨基酸生产菌种工业环境耐受性,开发了由胁迫诱导突变模块组成的基因组复制工程辅助连续进化系统(GREACE),在胁迫环境下实现了“边突变边筛选” [54]。在GREACE 的基础上,引入突变的DNA 聚合酶校对元件(DnaQ),使突变率增加了317 倍,并利用GREACE 技术筛选到突变菌株大肠杆菌RS3,将L-赖氨酸产量提高到155 g/L。研究人员进一步利用基因组学和代谢组学技术,解析突变菌株大肠杆菌RS3 耐受机制,发现大肠杆菌RS3 通过前蛋白转位酶(SecYM145V)、ATP 合酶亚基(AtpBS165N)和瓜丁胺酶(SpeBA302V)的突变,提高了大肠杆菌细胞在胁迫条件的完整性并强化了合成目标化学品赖氨酸代谢流[52](图2c)。
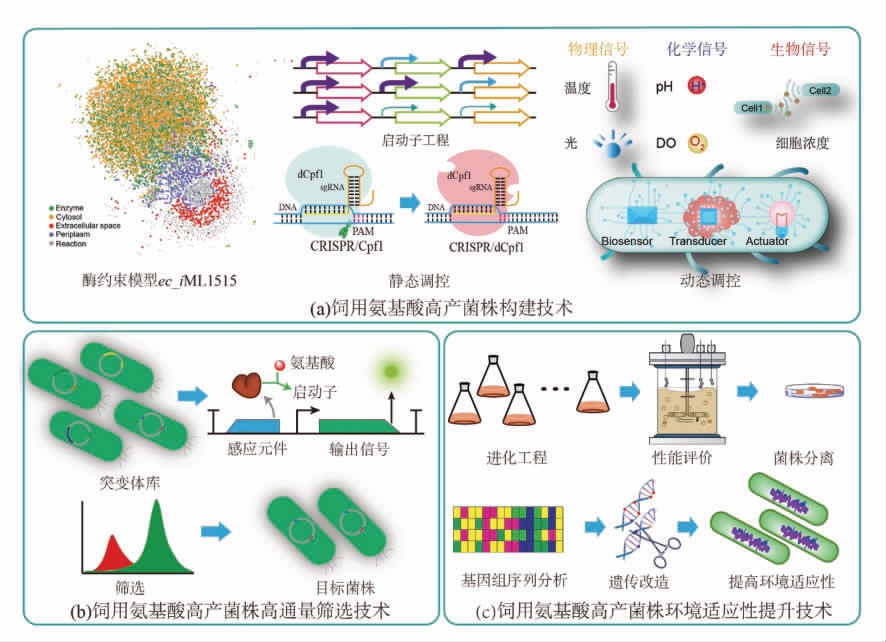
图2 代谢工程改造微生物细胞工厂生产饲用氨基酸
Fig.2 Enhancing feed amino acid production by metabolic engineering in microbial cell factories
3 微生物生产单细胞蛋白
蛋白质是生命活动的主要承担者,在人体健康与生长发育过程中具有不可替代的作用。长期以来,人们主要通过畜牧业获取肉、蛋、奶等产品作为蛋白质的主要来源。基于传统畜牧业的蛋白生产能力,难以满足人们日益增长的蛋白质需求。随着合成生物学、系统代谢工程等新兴生物技术的发展,人们开发了利用非致病和非产毒的酵母菌、真菌、藻类和细菌,生产营养价值高,原料来源广,培养周期短的单细胞蛋白(SCP),实现了向微生物要蛋白[9,55-56](图3)。
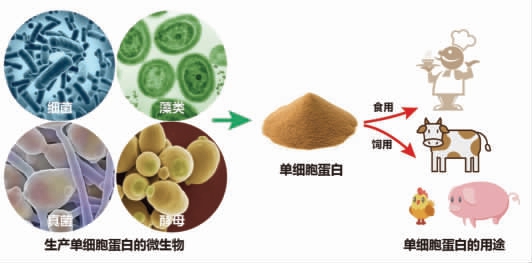
图3 微生物细胞工厂生产单细胞蛋白
Fig.3 Production of single cell protein by microbial cell factories
3.1 生产单细胞蛋白的微生物细胞
能生产单细胞蛋白的微生物有细菌、藻类、真菌和酵母。其中,细菌具有倍增时间短、生长迅速,营养需求简单、底物谱广等优点[55-56]。细菌单细胞蛋白含量约占细胞干重的40%~80%,且在氨基酸组成上优于酵母或真菌单细胞蛋白。例如嗜甲基菌属(Methylophilus spp)倍增时间仅为2 h,用其生产的单细胞蛋白能替代动物饲料蛋白。此外,细菌在利用废水中的碳、氮、硫、磷营养物质生产单细胞蛋白方面展现巨大的应用潜力,如紫色光合细菌(Purple photosynthetic bacteria)可用于回收环境废弃物中的有机物、氮和磷,用于生产单细胞蛋白。然而,细菌通常体积小,分离困难,所获得的蛋白质不易消化。
藻类单细胞蛋白具有蛋白含量高(占细胞干重的50%~70%),富含脂肪酸、维生素、色素、多肽,核酸含量低(占细胞干重的3%~8%)等优点[57]。藻类能利用太阳光和CO2 进行生长和转化为碳水化合物,加上具有蛋白含量高、生长速度快、栽培简单的优点,被广泛应用于饲料生产,如小球藻(Chlorella)和栅藻(Senedessmus)被用作鱼类饲料[58]。藻类单细胞蛋白的缺点在于藻类细胞具有纤维质的细胞壁,不易被人体消化;同时藻类培养过程中能富集一定量的重金属,限制了其进一步应用。
真菌单细胞蛋白含量在30%~50%之间,含有丰富的苏氨酸和赖氨酸,甲硫氨酸含量较低[57]。真菌单细胞蛋白还可以提供核黄素、烟酸、硫胺素、生物素、泛酸等多种维生素。如1985年Marlow Foods(GB)公司推出的威尼斯镰刀菌(Fusarium venenatum)单细胞蛋白,可以替代动物肉类,形成肉类替代品QuornTM,进一步研究发现,摄食威尼斯镰刀菌单细胞蛋白后,人体的胰岛素和血糖水平显著降低[59]。此外,芬兰科学家以糖、木材水解物或亚硫酸盐废料上生长的丝状真菌宛氏拟青霉(Paecilomyces varioti) 作为单细胞蛋白用于动物饲料[60]。真菌单细胞蛋白存在的问题是真菌生产速度较慢、核酸含量高,从而引起痛风或风湿性关节炎。
酵母细胞生产单细胞蛋白具有以下几个优点:1)含有45%左右的优质全价蛋白质,包括儿童所必需的组氨酸在内的人体必需的9 种氨基酸,满足人类营养需求;2)酵母单细胞蛋白易消化,无致敏成分,无豆腥味,适用人群广泛;3)酵母发酵工艺成熟,适合于规模化制造;4) 酵母细胞体积大,易于分离回收。例如:酿酒酵母作为酿酒工业的副产品,可用于生产酵母提取物以及马麦酱、维吉麦酱等酵母咸味酱。2019年,欧洲食品安全局(EFSA) 接受经干燥和灭活处理的解脂耶氏酵母为新型食品[61]。
3.2 用于生产单细胞蛋白的微生物工程改造
对生产单细胞蛋白的微生物菌株进行改造,主要涉及提高底物利用效率,提高细胞环境耐受性,提高细胞生长速度和蛋白质的含量。在利用复杂原料生产单细胞蛋白过程中,原料中有毒组分会抑制细胞生长,从而降低细胞浓度,降低单细胞蛋白的生产效率[62]。通过表达外转运蛋白和工程改造转录因子,可以减少微生物细胞对这些有毒组分的摄取,从而提高细胞的耐受性和生长速度[63]。而过表达转运蛋白可以提高其吸收底物的速度,提高底物利用率,进一步促进细胞生长[64-65]。在以多种糖作为原料时,表达多种糖摄取转运蛋白能显著改善细胞的生长性能,如在蓝藻PCC 7002(Synechococcus sp PCC 7002)中过表达天然Na+依赖性碳转运蛋白(SbtA 和BicA),显著提高了细胞生长速率和干细胞质量[65]。在以甘油(工业过程中的关键副产品)为原料生产单细胞蛋白时,在产甘油假丝酵母(Candida glycerinogenes)中过量表达甘油转运蛋白(STL1 和STL2),显著增加了产SCP 的微生物细胞浓度[66]。
适应性进化(ALE)、化学诱变和基因组工程也被广泛用于改善微生物细胞的生长性能和生物量积累。如:借助适应性进化技术,筛选获得能在一种或多种抑制剂存在的情况下生长速率提高3.3 倍,而延滞期缩短56%的菌株马克斯克鲁维酵母(Kluveronymces maxianus)。将化学诱变与适应性进化相结合,则能显著提高大肠杆菌甲基营养菌株的甲醇耐受性[67]。
生产单细胞蛋白的微生物细胞浓度也可以通过协同生长的方式得到提高[68-69]。工程化改造的方法包括:调节细胞表观遗传因素,微生物群落的结构,共存菌株的营养需求,可利用营养物质,副产品的途径。相关策略包括快速基因组测序/重新设计,多组学数据生成和分析,计算机建模,提高生产量和生物制造自动化等技术的发展。
提高蛋白质含量的方法策略包括:减少mRNA 和蛋白质降解,增强蛋白质折叠和重折叠,减少蛋白质的降解。mRNA 降解与核糖核酸酶(RNase)在mRNA 上的结合概率有关,为了减少总mRNA 降解维持胞内总mRNA 水平,一种直接的方法是开发和使用核糖核酸酶(RNase)缺陷菌株[70]。分子伴侣(如大肠杆菌GroEL/ES 和DnaK/J/GrpE)可以指导翻译蛋白质的正确折叠,对错误折叠或聚集的蛋白质进行重新折叠。单个或多个伴侣的共表达是增强微生物细胞中蛋白质含量的有效方法[71]。提高蛋白质产量的另一方法是开发和使用缺乏蛋白酶的微生物细胞,减少蛋白质降解。如敲除枯草芽孢杆菌中8 个细胞外蛋白酶基因,可以获得具有更高稳定性的异源蛋白,然而,很少有文献报道蛋白酶基因的缺失会提高SCP 中的总蛋白质产量[72]。
3.3 用于生产单细胞蛋白的原料
单细胞蛋白生产所需的原料包括碳源、氮源、无机盐、磷盐等。常用碳源包括气态碳氢化合物、正烯烃、乙醇、甲醇、碳氧化物、多糖、糖蜜、各种啤酒厂的流出物和其它固体化合物[57]。原料占单细胞蛋白生产总成本的45%~75%,碳源占据大部分原料成本,而氮源仅占原料成本的7%~15%[73]。
以碳为主要成分的碳水化合物在自然界中分布广泛,易于获取,且易被微生物所利用,如糖蜜、乳清、淀粉和烷烃等碳水化合物。生产单细胞蛋白的其它碳源包括二氧化碳、正烯烃、乙醇、甲醇、碳氧化物等[57],而在藻类的培养过程中需要高耗能的搅拌促进二氧化碳的溶解。利用来源丰富且价格低廉的农业和工业废弃物生产单细胞蛋白,不仅可以减少近80%的生物需氧量,而且还可最大限度地减少处置它们所需的处理成本[74]。然而,使用前需要对其特殊处理,增加了生产成本,而工业废弃物在使用过程具有高水平的生物需氧量,如果不进行处理就会造成环境污染。碳氢化合物及其衍生物也可用作生产单细胞蛋白的底物,约占总生产成本的30%~70%[61]。碳氢化合物通常从天然气或石油中获得,在本质上属于不可再生资源。
微生物在生长和生产单细胞蛋白的过程中,需要氨、铵盐或硝酸盐等提供的氮源,而且在生长过程中培养基需要保持合适的碳氮比[60]。研究表明,微生物中存在相似的碳氮比,培养基中碳氮比维持在10∶1 最优。也有研究表明,不同微生物之间,培养基碳氮比不同。当碳氮比较高时,导致培养基中的氨先被消耗完,影响后期单细胞蛋白的合成。当碳氮比在1∶1 时,氨大部分被浪费掉,不能进入细胞[56]。除碳水化合物和氮源外,生产单细胞蛋白的培养基中还需补充一定量的微生物生长所需的维生素、矿物质等营养物质,以最大限度地获得最大蛋白产量。
4 展望
利用合成生物学、系统代谢工程等新兴生物技术构建的微生物细胞工厂,实现了向自然界要铵,向微生物要氨基酸和向微生物要蛋白,在满足人体营养需求的前提下,有效减少土地资源、淡水资源的利用和温室气体的排放。氮素不仅是氨基酸与蛋白质的重要组成部分,也是动植物生长的必需元素之一。围绕生物固氮等氮代谢途径,开发新型生物固氮技术,克服天然固氮体系缺陷,创制新一代人工高效固氮技术是未来的研究方向。目前,微生物细胞主要以“粮食生物质”淀粉作为原料的第1 代生物炼制技术进行微生物氨基酸和微生物蛋白质合成,存在“与人争粮,与粮争地”等问题。为了解决这个问题,可以开发以木质纤维素等非粮生物质作为原料的第2 代生物炼制技术。CO2作为自然界含量丰富的碳源,预计到2050年CO2排放量将增加到500 亿t/年。利用CO2 作为微生物细胞工厂的原料,不仅可以实现“不与人争粮,不与粮争地”的目标,而且可以减少CO2 排放,为实现碳达峰与碳中和提供一条有前景的发展路线。虽然通过科研人员的不断努力,赋予或者强化了微生物细胞工厂的固定CO2 的能力,但是微生物细胞工厂大规模利用CO2 生产目标化学品还缺乏市场竞争力,迫切需要捕碳元件与能量供给方面的突破。
[1]陈坚.中国食品科技:从2020 到2035[J].中国食品学报,2019,19(12):1-5.
CHEN J.Food science and technology in China:from 2020 to 2035[J].Journal of Chinese Institute of Food Science and Technology,2019,19(12):1-5.
[2]郭春雷,苏畅,王志宏.2015年中国十五省(区、市)18~64 岁居民膳食蛋白质摄入状况[J].营养学报,2018,40(5):428-433.
GUO C L,SU C,WANG Z H,et al.Dietary protein intake of adults aged 18-64 in 15 provinces of China in 2015[J].Acta Nutrimenta Sinica,2018,40(5):428-433.
[3]贺志雄,周传社,王敏.草食畜牧业乳肉功能性产品发展的战略思考[J].中国科学院院刊,2021,36(6):685-691.
HE Z X,ZHOU C S,WANG M,et al.Developing strategies of functional milk and meat productsfrom herbivorous animal husbandry[J].Bulletin of the Chinese Academy of Sciences,2021,36(6):685-691.
[4]MEKONNEN M M,HOEKSTRA A Y.A global assessment of the water footprint of farm animal products[J].Ecosystems,2012,15(3):401-415.
[5]GUO L,DING S,LIU Y,et al.Enhancing tryptophan production by balancing precursors in Escherichia coli[J].Biotechnology and Bioengineering,2022,119(3):983-993.
[6]LI M,LIU C,YANG J,et al.Common problems associated with the microbial productions of aromatic compounds and corresponding metabolic engineering strategies[J].Biotechnology Advances,2020,41:107548.
[7]DO CARMO FELIX F K,JUNIOR LETTI L A,DE MELO PEREIRA G V,et al.L-lysine production improvement:a review of the state of the art and patent landscape focusing on strain development and fermentation technologies[J].Critical Reviews in Biotechnology,2019,39(8):1031-1055.
[8]LI Y,WEI H,WANG T,et al.Current status on metabolic engineering for the production of L-aspartate family amino acids and derivatives[J].Bioresource Technology,2017,245:1588-1602.
[9]MATASSA S,BOON N,PIKAAR I,et al.Microbial protein:future sustainable food supply route with low environmental footprint[J].Microbial Biotechnology,2016,9(5):568-575.
[10]VAN HEESWIJK W C,WESTERHOFF H V,BOOGERD F C.Nitrogen assimilation in Escherichia coli:Putting molecular data into a systems perspective[J].Microbiology and Molecular Biology Reviews,2013,77(4):628-695.
[11]VO J,INWOOD W,HAYES J M,et al.Mechanism for nitrogen isotope fractionation during ammonium assimilation by Escherichia coli K12[J].Proceedings of the National Academy of Sciences of the United States of America,2013,110(21):8696-8701.
[12]MARCHANT H K,KARTAL B.The microbial nitrogen-cycling network[J].Nature Reviews Microbiology,2018,16(5):263-276.
[13]VICENTE E J,DEAN D R.Keeping the nitrogenfixation dream alive[J].Proceedings of the National Academy of Sciences of the United States of America,2017,114(12):3009-3011.
[14]燕永亮,田长富,杨建国,等.人工高效生物固氮体系创建及其农业应用[J].生命科学,2021,33(12):1532-1543.
YAN Y L,TIAN C F,YANG J G,et al.Establishment of artificial efficiency biological nitrogen fixation system and its agricultural application [J].Chinese Bulletin of Life Sciences,2021,33(12):1532-1543.
[15]EADY R R.Structure-function relationships of alternative nitrogenases[J].Chemical Reviews,1996,96(7):3013-3030.
[16]BUREN S,JIMENEZ-VICENTE E,ECHAVARRIERASUN C,et al.Biosynthesis of nitrogenase cofactors[J].Chemical Reviews,2020,120(12):4921-4968.
[17]BAGESHWAR U K,SRIVASTAVA M,PARDHASARADHI P,et al.An environmentally friendly engineered Azotobacter strain that replaces a substantial amount of urea fertilizer while sustaining the same wheat yield[J].Applied and Environmental Microbiology,2017,83(15):e00590-17.
[18]YANG J,XIE X,YANG M,et al.Modular electron-transport chains from eukaryotic organelles function to support nitrogenase activity[J].Proceedings of the National Academy of Sciences of the United States of America,2017,114(12):E2460-E2465.
[19]ZEHR J P,JENKINS B D,SHORT S M,et al.Nitrogenase gene diversity and microbial community structure:a cross-system comparison[J].Environmental Microbiology,2003,5(7):539-554.
[20]BREWIN B,WOODLEY P,DRUMMOND M.The basis of ammonium release in nifL mutants of Azotobacter vinelandii[J].Journal of Bacteriology,1999,181(23):7356-7362.
[21]DIXON R A,POSTGATE J R.Genetic transfer of nitrogen fixation from Klebsiella pneumoniae to Escherichia coli[J].Nature,1972,237(5350):102-103.
[22]DIXON R,KAHN D.Genetic regulation of biological nitrogen fixation[J].Nature Reviews Microbiology,2004,2(8):621-631.
[23]LI X X,LIU Q,LIU X M,et al.Using synthetic biology to increase nitrogenase activity[J].Microbial Cell Factories,2016,15:43.
[24]MORENO-VIVIAN C,CABELLO P,MARTINEZLUQUE M,et al.Prokaryotic nitrate reduction:Molecular properties and functional distinction among bacterial nitrate reductases[J].Journal of Bacteriology,1999,181(21):6573-6584.
[25]KAMP A,DE BEER D,NITSCH J L,et al.Diatoms respire nitrate to survive dark and anoxic conditions[J].Proceedings of the National Academy of Sciences of the United States of America,2011,108(14):5649-5654.
[26]MALM S,TIFFERT Y,MICKLINGHOFF J,et al.The roles of the nitrate reductase NarGHJI,the nitrite reductase NirBD and the response regulator GlnR in nitrate assimilation of Mycobacterium tuberculosis[J].Microbiology-SGM,2009,155(4):1332-1339.
[27]ETTWIG K F,ZHU B L,SPETH D,et al.Archaea catalyze iron-dependent anaerobic oxidation of methane[J].Proceedings of the National Academy of Sciences of the United States of America,2016,113(45):12792-12796.
[28]MAIA L B,MOURA J J G.How biology handles nitrite[J].Chemical Reviews,2014,114(10):5273-5357.
[29]TIKHONOVA T V,SLUTSKY A,ANTIPOV A N,et al.Molecular and catalytic properties of a novel cytochrome c nitrite reductase from nitrate-reducing haloalkaliphilic sulfur-oxidizing bacterium Thioalkalivibrio nitratireducens[J].Biochimica Et Biophysica Acta -Proteins and Proteomics,2006,1764(4):715-723.
[30]ATKINSON S J,MOWAT C G,REID G A,et al.An octaheme c -type cytochrome from Shewanella oneidensis can reduce nitrite and hydroxylamine[J].FEBS Letters,2007,581(20):3805-3808.
[31]HAASE D,HERMANN B,EINSLE O,et al.Ep silonproteobacterial hydroxylamine oxidoreductase(epsilon Hao):characterization of a 'missing link' in the multihaem cytochrome c family[J].Molecular Microbiology,2017,105(1):127-138.
[32]SARAIVA L M,VICENTE J B,TEIXEIRA M.The role of the flavodiiron proteins in microbial nitric oxide detoxification[M]// Advances in Microbial Physiolgy.London:Elsevier,2004,49:77-129.
[33]WANG J,VINE C E,BALASINY B K,et al.The roles of the hybrid cluster protein,Hcp and its reductase,Hcr,in high affinity nitric oxide reduction that protects anaerobic cultures of Escherichia coli against nitrosative stress[J].Molecular Microbiology,2016,100(5):877-892.
[34]SHOUN H,FUSHINOBU S,JIANG L,et al.Fungal denitrification and nitric oxide reductase cytochrome P450nor[J].Philosophical Transactions of the Royal Society b-Biological Sciences,2012,367(1593):1186-1194.
[35]KARTAL B,DE ALMEIDA N M,MAALCKE W J,et al.How to make a living from anaerobic ammonium oxidation[J].FEMS Microbiology Reviews,2013,37(3):428-461.
[36]MAALCKE W J,DIETL A,MARRITT S J,et al.Structural basis of biological NO generation by octaheme oxidoreductases[J].Journal of Biological Chemistry,2014,289(3):1228-1242.
[37]CARANTO J D,LANCASTER K M.Nitric oxide is an obligate bacterial nitrification intermediate produced by hydroxylamine oxidoreductase[J].Proceed ings of the National Academy of Sciences of the United States of America,2018,115(35):EB325-EB325.
[38]KARTAL B,MAALCKE W J,DE ALMEIDA N M,et al.Molecular mechanism of anaerobic ammonium oxidation[J].Nature,2011,479(7371):127-159.
[39]MAALCKE W J,REIMANN J,DE VRIES S,et al.Characterization of anammox hydrazine dehydrogenase,a key N-2-producing enzyme in the global nitrogen cycle[J].Journal of Biological Chemistry,2016,291(33):17077-17092.
[40]YIZHAK K,BENYAMINI T,LIEBERMEISTER W,et al.Integrating quantitative proteomics and metabolomics with a genome-scale metabolic network model[J].Bioinformatics,2010,26(12):255-260.
[41]YE C,LUO Q,GUO L,et al.Improving lysine production through construction of an Escherichia coli enzyme-constrained model[J].Biotechnology and Bioengineering,2020,117(11):3533-3544.
[42]MONK J M,LLOYD C J,BRUNK E,et al.iML1515,a knowledgebase that computes Escherichia coli traits[J].Nature Biotechnology,2017,35(10):904-908.
[43]SANCHEZ B J,ZHANG C,NILSSON A,et al.Improving the phenotype predictions of a yeast genome-scale metabolic model by incorporating enzymatic constraints[J].Molecular Systems Biology,2017,13(8):935.
[44]LI M,CHEN J,WANG Y,et al.Efficient multiplex gene repression by CRISPR -dCpf1 in Corynebacterium glutamicum[J].Frontiers in Bioengineering and Biotechnology,2020,8:1-9.
[45]XU P.Production of chemicals using dynamic con trol of metabolic fluxes[J].Current Opinion in Biotechnology,2018,53:12-19.
[46]SHEN X,WANG J,LI C,et al.Dynamic gene expression engineering as a tool in pathway engineering[J].Current Opinion in Biotechnology,2019,59:122-129.
[47]FANG Y,WANG J,MA W,et al.Rebalancing microbial carbon distribution for L-threonine maximization using a thermal switch system[J].Metabolic Engineering,2020,61:33-46.
[48]HUANG J F,SHEN Z Y,MAO Q L,et al.Systematic analysis of bottlenecks in a multibranched and multilevel regulated pathway:the molecular fundamentals of L-methionine biosynthesis in Escherichia coli[J].ACS Synthetic Biology,2018,7(11):2577-2589.
[49]WANG Y,LI Q,ZHENG P,et al.Evolving the L-lysine high-producing strain of Escherichia coli using a newly developed high-throughput screening method[J].Journal of Industrial Microbiology and Biotechnology,2016,43(9):1227-1235.
[50]ZHENG B,MA X,WANG N,et al.Utilization of rare codon-rich markers for screening amino acid overproducers[J].Nature Communications,2018,9(1):1-11.
[51]LIU Y,YUAN H,DING D,et al.Establishment of a biosensor-based high-throughput screening platform for tryptophan overproduction[J].ACS Synthetic Biology,2021,10(6):1373-1383.
[52]WANG X,LI Q,SUN C,et al.GREACE-assisted adaptive laboratory evolution in endpoint fermentation broth enhances lysine production by Escherichia coli[J].Microbial Cell Factories,2019,18:1-13.
[53]CHENG L K,WANG J,ZHAO X B,et al.An antiphage Escherichia coli mutant for higher production of L-threonine obtained by atmospheric and room temperature plasma mutagenesis[J].Biotechnology Progress,2020,36(6):1-11.
[54]ZHU L,LI Y,CAI Z.Development of a stress-induced mutagenesis module for autonomous adaptive evolution of Escherichia coli to improve its stress tolerance[J].Biotechnology for Biofuels,2015,8:1-10.
[55]OVERLAND M,TAUSON A H,SHEARER K,et al.Evaluation of methane-utilising bacteria products as feed ingredients for monogastric animals [J].Archives of Animal Nutrition,2010,64(3):171-189.
[56]SEZNEC J F.British petroleum and global oil 1950-1975:The challenge of nationalism[J].Middle East Journal,2001,55(2):343-345.
[57]NASSERI A T,RASOUL-AMINI S,MOROWVAT M H,et al.Single cell protein:production and process[J].American Journal of Food Technology,2011,6(2):103-116.
[58]RAJA R,HEMAISWARYA S,KUMAR N A,et al.A perspective on the biotechnological potential of microalgae[J].Critical Reviews in Microbiology,2008,34(2):77-88.
[59]LANG V,BELLISLE F,ALAMOWITCH C,et al.Varying the protein source in mixed meal modifies glucose,insulin and glucagon kinetics in healthy men,has weak effects on subjective satiety and fails to affect food intake[J].European Journal of Clinical Nutrition,1999,53(12):959-965.
[60]UGALDE U O,CASTRILLO J I.Single cell proteins from fungi and yeasts[J].Agriculture and Food Production,2002,2:123-149.
[61]RITALA A,HAKKINEN S T,TOIVARI M,et al.Single cell protein-state-of-the-art,industrial landscape and patents 2001-2016[J].Frontiers in Microbiology,2017,8:2009.
[62]GUPTA J K,RAI P,JAIN K K,et al.Overexpression of bicarbonate transporters in the marine cyanobacterium Synechococcus sp.PCC 7002 increases growth rate and glycogen accumulation[J].Biotechnology for Biofuels,2020,13(1):17.
[63]FEINBERG L F,MARX C J.Methylotrophs for aquaculture and animal feed:US 10 920 230 B2[P].2021-04-03.
[64]HARA K Y,KOBAYASHI J,YAMADA R,et al.Transporter engineering in biomass utilization by yeast[J].FEMS Yeast Research,2017,17(7):1-14.
[65]JI H,LU X Y,ZONG H,et al.Functional and expression studies of two novel STL1 genes of the osmotolerant and glycerol utilization yeast Candida glycerinogenes[J].Journal of General and Applied Microbiology,2018,64(3):121-126.
[66]ZHOU H,CHENG J S,WANG B L,et al.Xylose isomerase overexpression along with engineering of the pentose phosphate pathway and evolutionary engineering enable rapid xylose utilization and ethanol production by Saccharomyces cerevisiae[J].Metabolic Engineering,2012,14(6):611-622.
[67]BENNETT R K,GREGORY G J,GONZALEZ J E,et al.Improving the methanol tolerance of an Escherichia coli methylotroph via adaptive laboratory evolution enhances synthetic methanol utilization[J].Frontiers in Microbiology,2021,12:638426.
[68]KERCKHOF F M,SAKARIKA M,VAN GIEL M,et al.From biogas and hydrogen to microbial protein through Co-cultivation of methane and hydrogen oxidizing bacteria[J].Frontiers in Bioengineering and Biotechnology,2021,9:1-17.
[69]VETHATHIRRI R S,SANTILLAN E,WUERTZ S.Microbial community-based protein production from wastewater for animal feed applications[J].Bioresource Technology,2021,341:125723.
[70]ROUX C,ETIENNE T A,HAJNSDORF E,et al.The essential role of mRNA degradation in understanding and engineering E.coli metabolism[J].Biotechnology Advances,2022,54:107805.
[71]JUNG H,LING H,TAN Y Q,et al.Heterologous expression of cyanobacterial gas vesicle proteins in Saccharomyces cerevisiae[J].Biotechnology Journal,2021,16(12):e2100059.
[72]JEONG H J D,PARK S H,KIM S J,et al.Complete genome sequence of Bacillus subtilis strain WB800N,an extracellular protease-deficient derivative of strain 168[J].Microbiology Resource Announcements,2018,7(18):1380-1318.
[73]KAM S,KENARI A A,YOUNESI H.Production of single cell protein in stickwater by Lactobacillus acidophilus and Aspergillus niger[J].Journal of Aquatic Food Product Technology,2012,21(5):403-417.
[74]RAVINDER R,RAO L V,RAVINDRA P.Studies on Aspergillus oryzae mutants for the production of single cell proteins from deoiled rice bran[J].Food Technology and Biotechnology,2003,41(3):243-246.