食品安全是关乎人民生命健康和社会和谐的重大战略问题。婴幼儿配方食品是部分婴幼儿的唯一食物来源,对婴幼儿健康及生命安全有重要影响。克罗诺杆菌为革兰氏阴性菌,是一种普遍存在于大自然中的条件致病菌。一旦感染该菌,会对婴幼儿的健康造成严重危害,可引发坏死性小肠结肠炎、菌血症和新生儿脑膜炎等疾病,甚至可能留下严重的神经系统后遗症,死亡率高达40%~80%[1-3]。
在婴幼儿配方乳粉中存在克罗诺杆菌属高污染风险[4-8]。在生产过程中控制克罗诺杆菌污染是婴儿配方食品安全控制的关键环节,而快速精确检测是其质量保证的重要手段。
CRISPR 是指成簇的、规律间隔的短回文重复序列(Clustered regularly interspaced short palindromic repeats),是大多数细菌及古细菌中抵御病毒入侵的一种获得性免疫方式,该序列是许多原核生物的免疫系统[9]。CRISPR/Cas 系统的发现可以追溯到1987年。ISHINO 等[10]首次在大肠杆菌的基因组中发现串联的间隔重复序列,然而,当时未引起学术界的关注。2002年,Jansen 等[11]将这一奇特的重复间隔序列正式命名为串联间隔短回文重复序列(CRISPR)。CRISPR/Cas 系统本质上是一系列由RNA 引导的核酸内切酶 [12-14]。近年来,CRISPR 技术在基因组编辑领域表现出巨大的应用价值,在分子检测、病原菌检测,包括最近的新冠检测等领域也开始有重要的突破性应用[15-22]。本研究使用的Cas12b 属于第2 类CRISPR/Cas 系统中的Ⅴ型,具有附带切割活性[23-24]。在靶标核酸存在的情况下,Cas 蛋白在crRNA 引导下特异性结合靶标核酸并被激发出单链核酸非特异性切割活性,从而将体系中单链核酸报告分子切开并释放出可检测的信号。
本研究基于CRISPR-Cas12b 蛋白,利用团队原创建立的CRISPR-Cas12b 检测技术来建立重要食源性致病菌的分子检测方法,对婴幼儿配方食品中致病菌克罗诺杆菌属进行快速定性筛选检测,这对提升我国婴幼儿配方食品企业质量控制能力和民众关切的婴幼儿食品安全水平具有重要意义。
1 材料与方法
1.1 材料与试剂
选择8 株克罗诺杆菌属参比菌株和31 株实验室分离株,以及30 株非克罗诺杆菌属的纯化标准菌株进行特异性检测(表1、表2)。
表1 克罗诺杆菌属阳性菌株名称及其编号
Table 1 The name and number of positive strains of Cronobacter

编号 菌株名称 菌株号 菌种来源1阪崎克罗诺杆菌 ATCC25944 中国工业微生物菌种保藏管理中心2普遍克罗诺杆菌 NCTC 9529 上海海关3都柏林克罗诺杆菌 DSM 18705 上海海关4丙二酸盐阳性克罗诺杆菌 DSM 18702 上海海关5苏黎世克罗诺杆菌 DSM 18703 上海海关6都柏林克罗诺杆菌 DSM 18706 上海海关7穆汀斯克罗诺杆菌 ATCC 51329 上海海关8都柏林克罗诺杆菌 DSM 18707 上海海关9克罗诺杆菌属1-31 奶粉 分离自样品
表2 克罗诺杆菌属阴性菌株名称及其编号
Table 2 The name and number of negative strains of Cronobacter
编号 菌株名称 菌株号 菌种来源1 铜绿假单胞菌 ATCC10104 中国工业微生物菌种保藏管理中心2 奇异变形杆菌 ATCC33420 中国工业微生物菌种保藏管理中心3 大肠杆菌 ATCC25922 中国工业微生物菌种保藏管理中心4 金黄色葡萄球菌 ATCC6538 中国工业微生物菌种保藏管理中心5 粪肠球菌 ATCC29212 中国工业微生物菌种保藏管理中心6 产气肠杆菌 ATCC13048 中国工业微生物菌种保藏管理中心7 成团泛菌 ATCC27155 中国工业微生物菌种保藏管理中心8 单核细胞增生李斯特氏菌 ATCC19114 中国工业微生物菌种保藏管理中心9 蜡样芽孢杆菌 ATCC11778 中国工业微生物菌种保藏管理中心10 EHEC(O157:H7) CICC21530 中国工业微生物菌种保藏管理中心11 宋内氏志贺氏菌 ATCC 25931 中国工业微生物菌种保藏管理中心12 甲型副伤寒沙门氏菌 ATCC 9150 中国工业微生物菌种保藏管理中心13 鼠伤寒沙门氏菌 ATCC 13311 中国工业微生物菌种保藏管理中心14 猪霍乱沙门氏菌 ATCC 7001 中国工业微生物菌种保藏管理中心15 肠炎沙门氏菌 ATCC 13076 中国工业微生物菌种保藏管理中心16 鸭沙门氏菌 ATCC 9270 中国工业微生物菌种保藏管理中心17 阴沟肠杆菌 ATCC 35030 中国工业微生物菌种保藏管理中心18 肺炎克雷伯氏菌 ATCC 700603 中国工业微生物菌种保藏管理中心19 大肠杆菌 ATCC 11229 中国工业微生物菌种保藏管理中心20 婴儿双歧杆菌 CICC 6069 中国工业微生物菌种保藏管理中心21 德氏保加利亚亚种 CICC 6097 中国工业微生物菌种保藏管理中心22 动物双歧杆菌 DSMZ 10140 德国微生物菌种保藏中心23 植物乳杆菌 ATCC 8014 中国工业微生物菌种保藏管理中心24 痢疾志贺氏菌 CMCC(B) 51105 中国工业微生物菌种保藏管理中心25 福氏志贺氏菌 CMCC(B) 51572 中国工业微生物菌种保藏管理中心26 ETEC CICC10413 中国工业微生物菌种保藏管理中心27 EPEC CICC10372 中国工业微生物菌种保藏管理中心28 弗氏柠檬酸杆菌 ATCC 43864 中国工业微生物菌种保藏管理中心29 粘质沙雷氏菌 ATCC 14041 中国工业微生物菌种保藏管理中心30 痢疾志贺氏菌 CMCC(B) 51105 中国工业微生物菌种保藏管理中心
1.2 仪器与设备
ABI7500PCR 扩增仪、BIO-RADPCR 扩增仪,美国BIO-RAD;Vortex Genie2 漩涡振荡器,美国SI;移液枪,美国GILSON;SW-CJ-2D 超净工作台,苏州净化;ESCO LA2-4A1 生物安全柜,新加坡艺思高。
1.3 主要试剂与材料
Phanta ® Max Super -Fidelity DNA Polymerase,南京诺唯赞Vazyme,P505-d1;NEBuffer 3.1(10×),NEB,B7203S;UltraPureTM DNase/RNase-Free Distilled Water,ThermoFisher,10977023;RNA 酶抑制剂,Takara,2313B;Cas12b 蛋白,吐露港生物;细菌DNA 提取试剂盒DP302-02,天根生化科技(北京)有限公司,U8110。
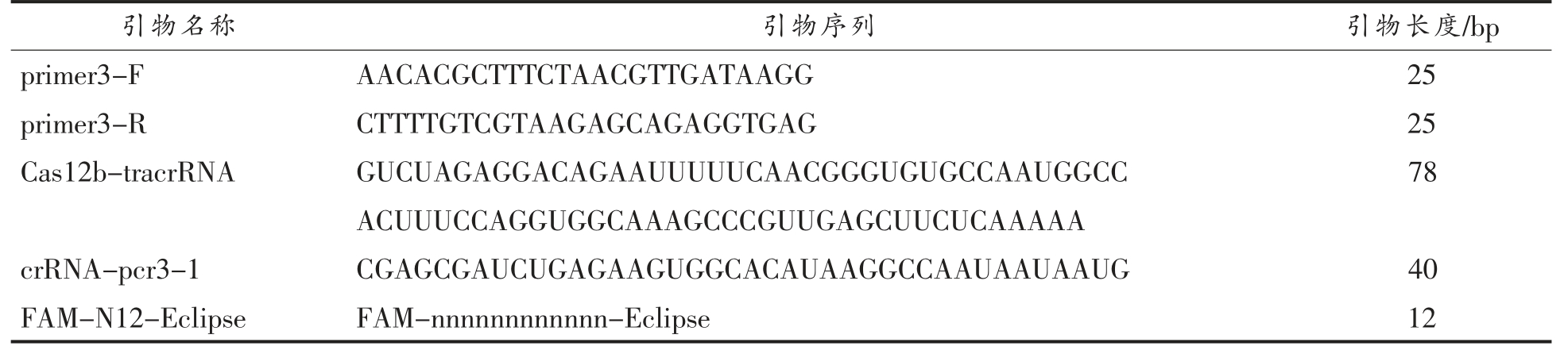
PCR 引物与探针见表3。
表3 引物与探针
Table 3 Primers and probes
?
2 试验方法
2.1 引物设计
通过基因组比较和候选靶点选择,以16S rRNA 基因保守序列、克罗诺杆菌菌株特异性关键基因序列、编码致病性相关基因序列及其他文献报道可区分菌株的特异性序列候选区域,针对每个区域设计2~3 对扩增引物和向导RNA 靶向序列进行生物信息学测试,挑选效率最高和特异性最好的组合,通过NCBI 在线工具进行序列分析和比对,利用Prime Express 软件V3.0(ABI,Foster City,CA,USA)设计出4 对引物和向导RNA组合,经过特异性及灵敏度测试,最终筛选出1对,序列见表3。CRISPR 反应的报告探针为12 个随机序列的单链DNA 序列,在探针的5' 端标记FAM,3' 端标记Eclipse。所有引物/探针均由上海生工生物有限公司合成,所有RNA 引物序列由南京金斯瑞生物科技有限公司合成。
2.2 核酸提取方法
2.2.1 标准菌株核酸提取方法 将标准菌株接种于营养肉汤中,36 ℃培养12 h,吸取1 mL 培养液,加入2 mL 无菌离心管中,13 000 r/min,离心5 min,去除上清,重复上述步骤1 次,即共吸取培养液2 mL 取沉淀。然后根据试剂盒进行提取。
2.2.2 试验样品核酸提取方法 样品进行前处理:取样品100 g 加入到900 mL 的BWP 中,混匀,36 ℃培养12 h。取2 mL 的样品培养液加入2 mL 离心管中13 400 g 离心5 min,而后提取核酸。
2.3 体系及条件
2.3.1 PCR 反应 反应体系包括25 μL PCR 反应混合液,上、下游引物(10 μmol/L)各2 μL,dNTP 1 μL,DNA 聚合酶1 μL,DNA 模板2 μL,用超纯水补足总体积为50 μL。扩增反应条件:95 ℃预变性5 min;95 ℃20 s,60 ℃20 s,72 ℃15 s 40 个循环;4 ℃保存。
2.3.2 CRISPR 反应 20 μL 的CRISPR 反应体系包括:10×NEBuffer 3.1 2 μL,Cas12b(5 μmol/L)1 μL,RNA 酶抑制剂(40 U/μL)0.25 μL,crRNA(10 μmol/L),tracrRNA(10 μmol/L) 和探针(10 μmol/L)各1 μL,待测DNA 模板2 μL,用超纯水补足总体积为20 μL。于ABI7500 荧光PCR 仪(美国,ABI 公司)上进行扩增,设置48 ℃程序,每1 min 检测一次FAM 荧光,持续15 min。
2.4 PCR 扩增条件优化
以克罗诺杆菌属(ATCC 25944)标准菌株核酸为模板,优化扩增温度。退火温度设置为54,56,58,60,62 ℃共5 个温度梯度,选择最适合的退火温度。在此基础上分别设置25,30,35,40,45 个循环数。选择最适宜的循环数。
2.5 特异性检测
特异性测试采用8 株克罗诺杆菌属菌株,31株实验室分离株和30 株非克罗诺杆菌属菌株。将表1,表2中所列克罗诺杆菌属标准菌株以及其它常见食源性致病菌接种于营养肉汤中,36 ℃培养12 h,取培养菌悬液2 mL 提取DNA。对提取的核酸用超微量分光光度计进行浓度和纯度的测定,确保提取核酸的A260/A280 在1.8~2.0 之间。利用PCR 对菌株DNA 进行扩增,使用CRISPR 反应检测。
2.6 灵敏度试验
以克罗诺杆菌属(ATCC 25944)标准菌株核酸为模板,对核酸质量浓度分别为100,10,1,0.1,0.01 ng/μL 进行检测。纯菌液检测限10 倍梯度稀释克罗诺杆菌属(ATCC 25944)纯菌液(107~102 CFU/mL),分别吸取2 mL 提取DNA 进行检测。
2.7 人工污染样品的制备
选择经传统方法和CRISPR 方法验证克罗诺杆菌属的奶粉样品作为添加基质。取克罗诺杆菌属(ATCC 25944)的过夜培养菌悬液分别进行10倍梯度稀释并平板计数。同时取<10~104 CFU/mL稀释度菌液各1 mL 分别添加到盛有100 g 奶粉样品的均质袋中,另取1 份100 g 奶粉样品不添加菌液作为空白对照,上述样品中加入900 mL BPW 缓冲蛋白胨水,用拍击式均质器拍击2 min,制成系列含有不同浓度的人工污染样品。经过12 h 的36 ℃增菌,取各样品匀液2 mL 提取DNA,进行CRISPR 检测。
2.8 实际样品检验
市场上购买婴幼儿配方乳粉共51 份。根据2.2.2 节进行前处理,提取样品核酸进行CRISPR检测。并用传统方法GB 4789.40 进行验证。
3 试验结果
3.1 扩增温度和循环数的优化结果
对温度优化扩增结果如图1显示,58~62 ℃对于检测的结果影响较小,60 ℃的退火温度检测效果最好,选择60 ℃作为扩增的退火温度。
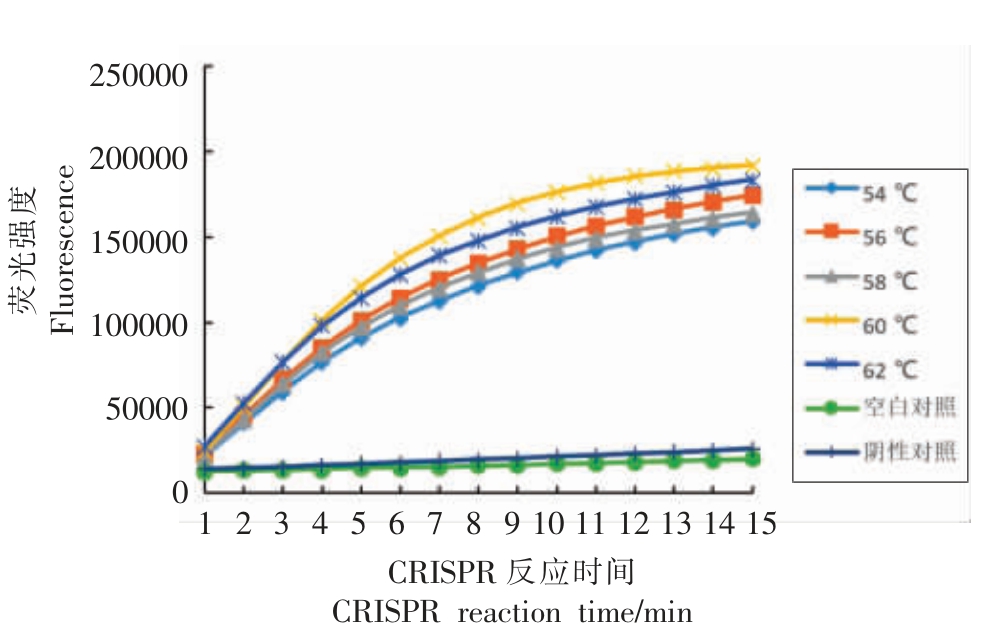
图1 退火温度优化结果图
Fig.1 Result of TM optimization test
对反应循环数的优化结果图如图2所示,可以看出,25,30 循环数扩增效果不太明显;35~45个循环有更为明显的CRISPR 反应效果。后续试验选择40 循环作为试验参数。

图2 反应循环优化结果图
Fig.2 Result of reaction cycle optimization test
3.2 特异性结果
特异性检测结果如图3、图4所示,结果发现所有克罗诺杆菌属出现阳性扩增信号,而其它常见食源性致病菌检测均没有明显扩增信号。表明引物探针组具有良好的特异性,方法适用于克罗诺杆菌属的特异性检测。
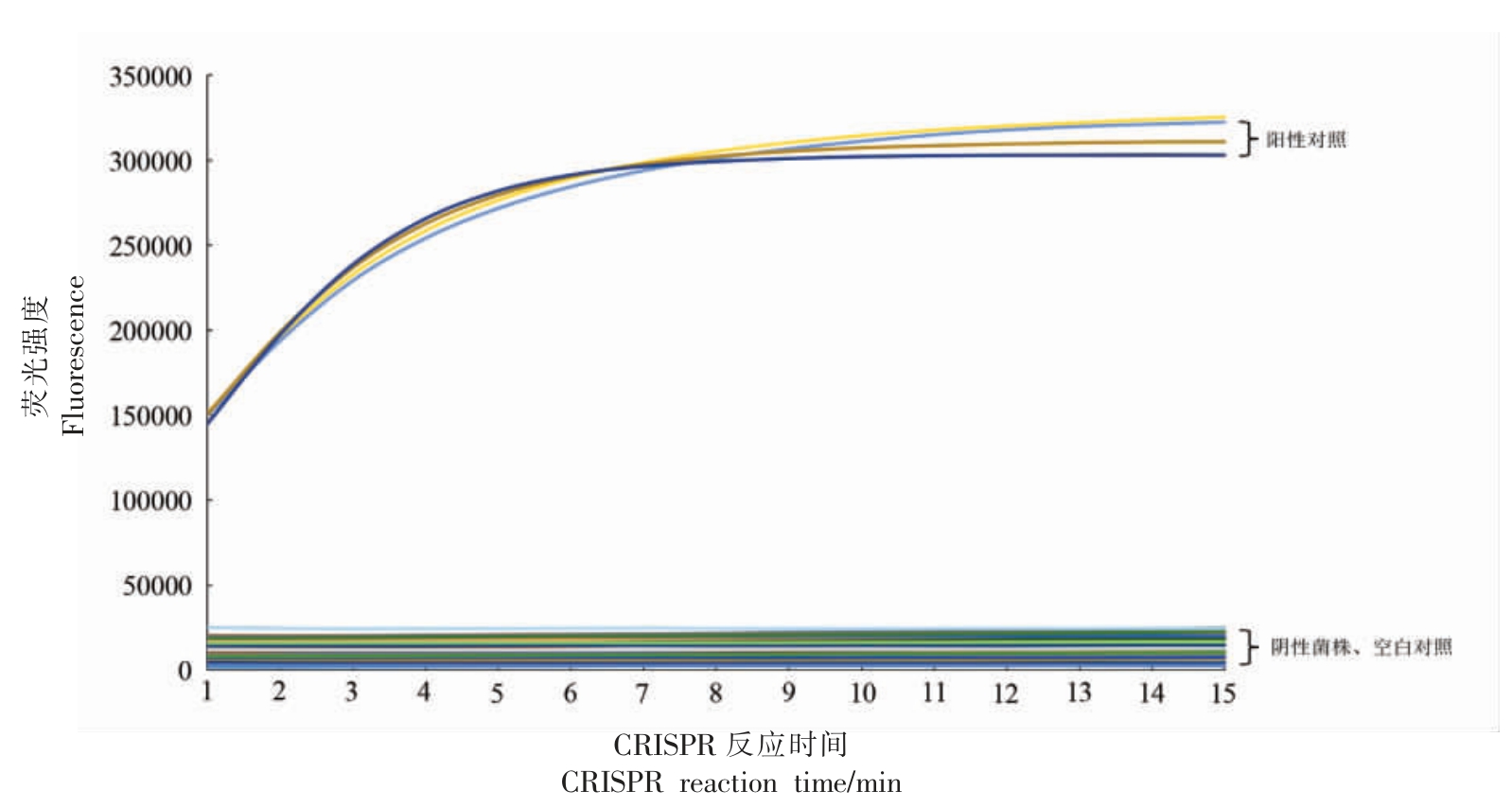
图3 非克罗诺杆菌属CRISPR 检测结果
Fig.3 CRISPR detection results of non Cronobacteria
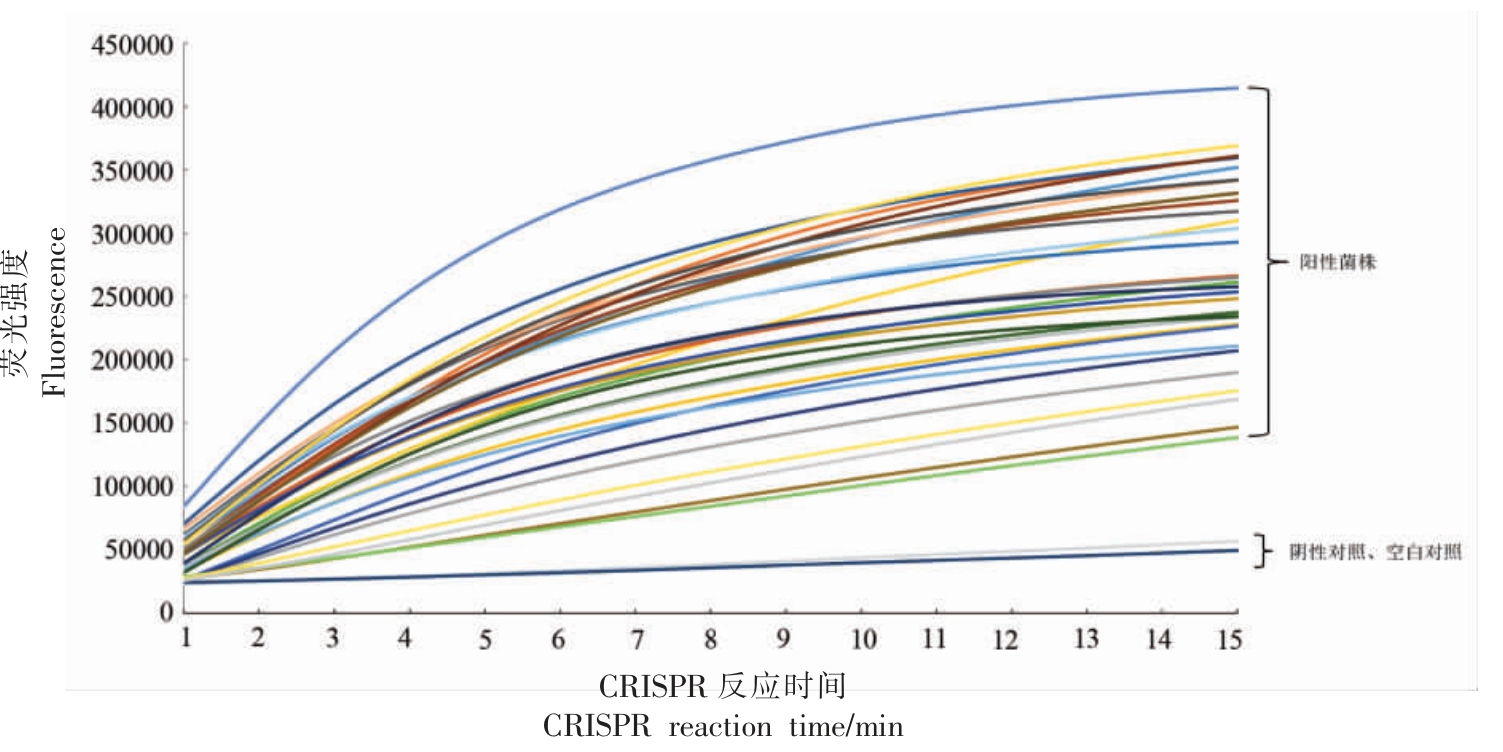
图4 克罗诺杆菌属与分离菌株CRISPR 检测结果
Fig.4 CRISPR detection results of Cronobacteria and its isolatest
3.3 CRISPR 结果判定
观察阳性菌株和阴性菌株的检测结果发现,阳性菌株的荧光信号随着反应进行有明显的增长趋势,而阴性菌株的荧光信号没有明显增长,通过增长趋势的差异可以判定检测结果。根据crispr检测相关的文献[17],定义判定值N 来判断检测结果。
计算公式:判定值N=(10 min 样品荧光值/10 min 对照荧光值)/(2 min 样品荧光值/2 min 对照荧光值)
式中:SF——样品荧光值;CF——对照荧光值;对照荧光值为水的荧光值。
统计所有结果,在这69 个标准菌株的结果中,发现阳性菌株及分离菌株判定值N 均≥1.40,并且有明显增长趋势,阴性菌株结果均≤1.20,没有明显增长趋势。对于1.40~1.20 之间的样品进行重复测试,如果N 值>1.20 判定样品CRISPR 检测筛选阳性,N 值≤1.20 时,则判定样品CRISPR 检测筛选阴性。通过传统方法及RT-qPCR 验证结果均符合预期。并且当检测低浓度样品的时候(如图5、图6、表4)CRISPR 方法更容易判定结果,阳性样品的抬头更明显。
表4 CT 值与N 值
Table 4 CT value and N value

?
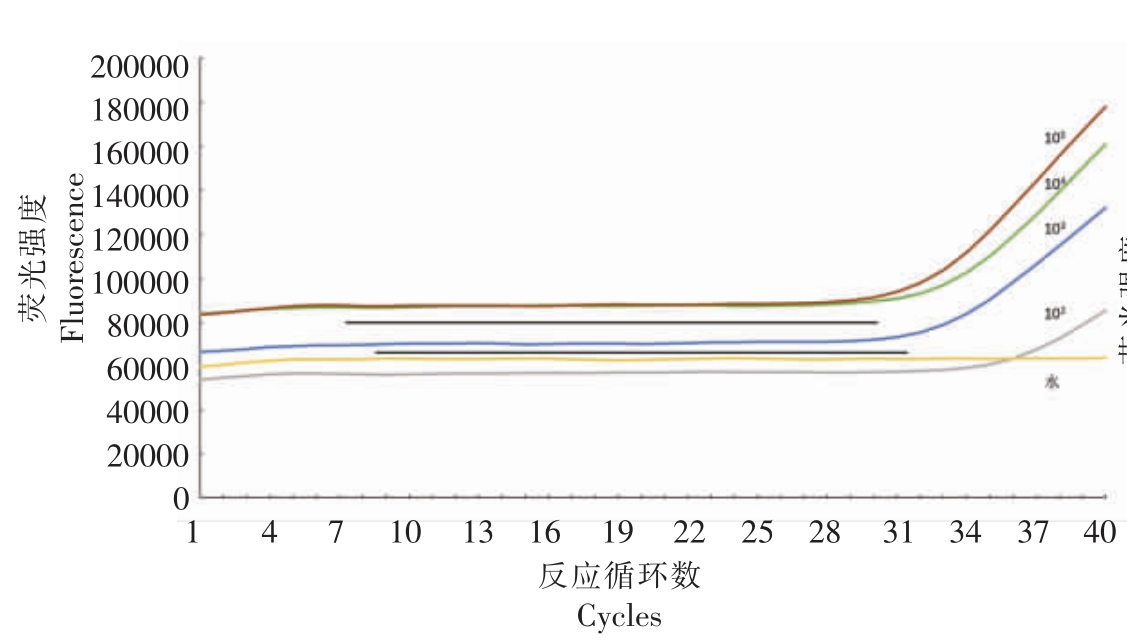
图5 RT-qPCR 结果
Fig.5 Results of RT-qPCR
3.4 灵敏度检测
核酸水平灵敏度检测结果如图7、表5所示,核酸质量浓度在100~0.01 ng/μL 之间判定值N均>1.2,判定为阳性。纯菌液检测结果如图6、表6所示,除102 CFU/g 的纯菌液检测结果≤1.2 以外,重复检测结果均>1.2,可以稳定检测到103 CFU/g的纯菌液。
表5 CRISPR 检测灵敏度判定值结果
Table 5 CRISPR sensitivity test N value results

核酸质量浓度/ng·μL-1 判定值N 100 1.85 10 1.80 1 1.86 0.1 1.82 0.01 1.56
表6 CRISPR 检测纯菌液判定值结果
Table 6 Pure bacterial solution test N value results

纯菌液浓度/CFU·g-1 判定值N 107 1.62 106 1.49 105 1.67 104 1.55 103 1.39 102 1.13
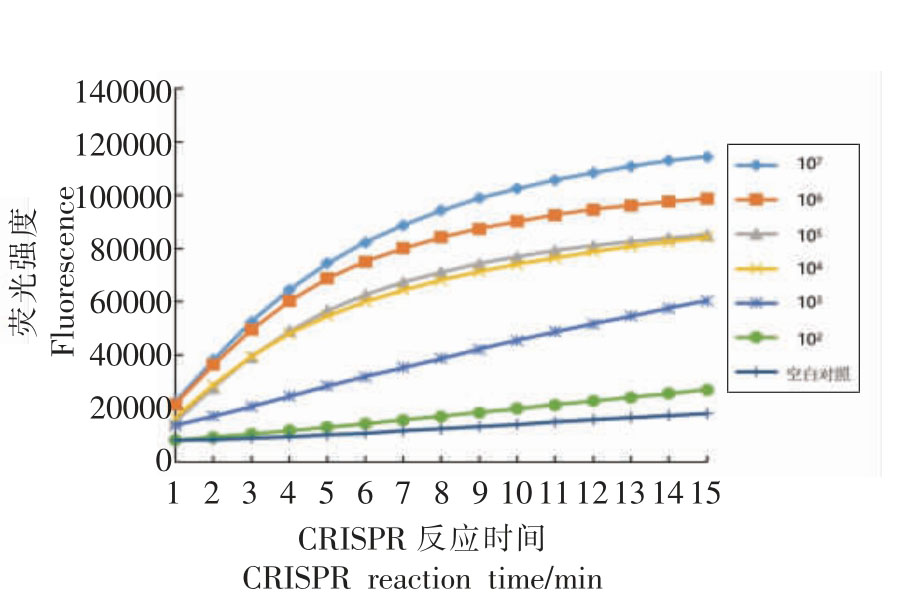
图6 纯菌液CRISPR 检测结果
Fig.6 CRISPR test results of pure bacterial solution

图7 CRISPR 检测灵敏度结果
Fig.7 CRISPR sensitivity test results
3.5 人工污染样品的检测
根据添加菌液的平板计数结果,估计人工污染样品中克罗诺杆菌属初始含量为<10~104 CFU/g。经过12 h 的增菌,将人工污染样品中提取的DNA进行扩增检测。结果发现4 个不同污染水平的奶粉样品均呈阳性扩增(图8、表7)。
表7 CRISPR 方法对人工污染样品的检测结果
Table 7 Results of artificially contaminated samples
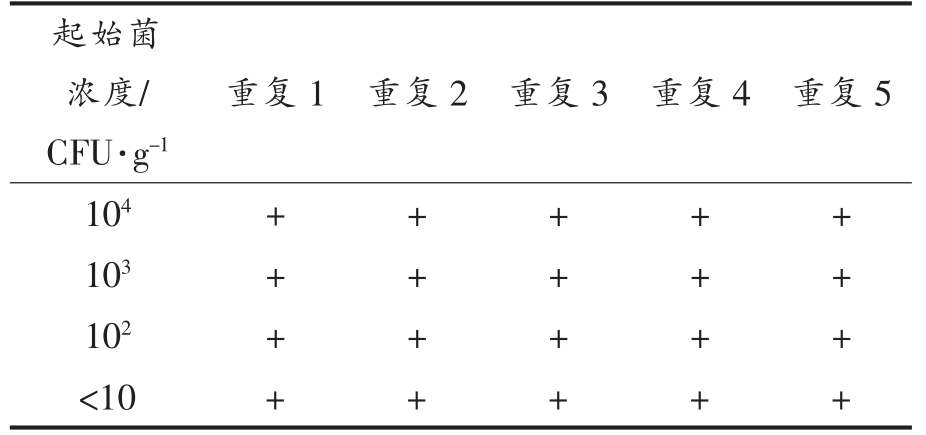
起始菌浓度/CFU·g-1重复1 重复2 重复3 重复4 重复5 104 + + + + +103 + + + + +102 + + + + +<10 + + + + +
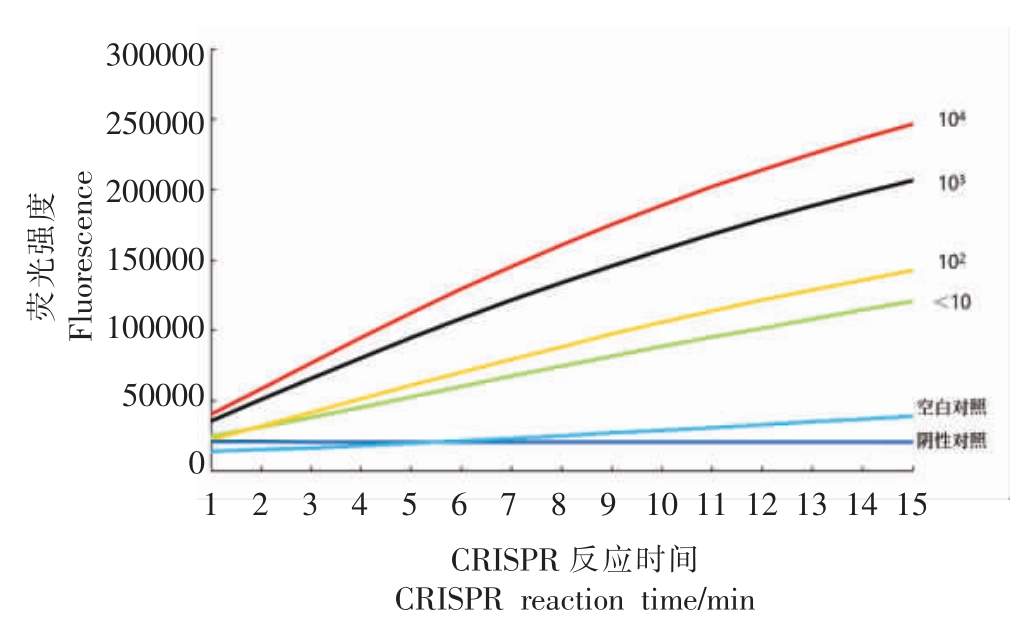
图8 CRISPR 方法对人工污染样品的检测结果
Fig.8 Results of artificially contaminated samples
3.6 实际样品检测
对市售的51 份样品进行CRISPR 检测,结果显示全部样品的判定值N≤1.20,均为阴性,未检出克罗诺杆菌属与传统方法检测结果一致。
4 讨论与结论
婴幼儿配方乳粉中克罗诺杆菌的污染风险控制是乳粉生产质量控制中的一个重要环节,目前,食品克罗诺杆菌属检测方法主要是常规的培养法、荧光PCR 法、环等温扩增法等检测方法。这些技术在灵敏度、特异性、简便性、速度和价格上各有优劣,但仍有容易污染、需要大型扩增设备等缺陷[25-30]。
目前主要使用的传统检测方法如GB 4789.40-2016 《食品安全国家标准 食品微生物学检验克罗诺杆菌属(阪崎肠杆菌)检验》能得到食品样本中克罗诺杆菌的定性和定量测定结果,但都需经富集培养、选择性分离、形态特征观察、生理生化鉴定等过程,操作复杂、耗时费力,检测过程至少需要4~7 d,而且灵敏度低,容易发生错检和漏检,无法对人工难以培养的致病菌进行检测。
本研究使用的CRISPR 的检测技术被誉为“下一代的分子检测技术”,能做到“快速、灵敏、高特异、简便、低价”的特性,所建立的检测方法特异性高,方法检出限为<10 CFU/100 g,实际样品检验结果与预期结果相符。本研究也是CRISPR 检测技术在国际食品安全领域方面的创新性技术开拓和应用探索,是国际首次将该技术在食品安全领域进行的创新性研究及应用,将对我国食品安全检测能力提升产生深远影响,提高我国在食品安全检测领域的国际话语权,具有重要意义,因此具有良好的应用前景。在之后的研究中,可以使用该技术与其它检测技术联合应用,进一步提升其检测速度,灵敏度,提升我国在快速检测领域的能力。
[1]JARADAT Z W,AL MOUSA W,ELBETIEHA A,et al.Cronobacter spp.- opportunistic food-borne pathogens.A review of their virulence and environmental-adaptive traits[J].Journal of Medical Microbiology,2014,63(8):1023-1037.
[2]NAZAROWEC-WHITE M,FARBER J M.Enterobacter sakazakii:A review[J].International Journal of Food Microbiology,1997,34(2):103-113.
[3]GURTLER J B,KORNACKI J L,BEUCHAT L R.Enterobacter sakazakii:A coliform of increased concern to infant health[J].International Journal of Food Microbiology,2005,104(1):1-34.
[4]徐湾,姜华,张选飞,等.我国克罗诺杆菌污染现状与预防控制措施研究进展[J].中国病原生物学杂志,2015,10(9):848-851.
XU W,JIANG H,ZHANG X F,et al.Advances in research on Cronobacter spp.:Current contamination status and preventive measures in China [J].Journal of Pathogen Biology,2015,10(9):848-851.
[5]陈万义,任婧,刘振民,等.婴幼儿配方粉中克罗诺杆菌属菌株检测方法研究进展[J].乳业科学与技术,2015,38(2):23-28.
CHEN W Y,REN J,LIU Z M,et al.A review of detection methods for Cronbacter spp.in power infant formula[J].Journal of Dairy Science and Technology,2015,38(2):23-28.
[6]梁兰英,许娟,林福全.佛山市三水区销售婴幼儿配方粉中阪崎肠杆菌污染状况调查[J].食品安全导刊,2015(3):31-32.
LIANG L Y,XU J,LIN F Q.Investigation on Enterobacter Sakazakii contamination in infant formula powder sold in Sanshui district of Foshan [J].China Food Safety Magazine,2015(3):31-32.
[7]朱凤,李维克,俞慕华,等.深圳乳制品和环境中阪崎肠杆菌污染状况调查[J].现代预防医学,2012,39(16):4107-4109.
ZHU F,LI W K,YU M H,et al.Investigation on contamination by Enterobacter sakazakii in dairy products and environment in Shenzhen[J].Modern Preventive Medicine,2012,39(16):4107-4109.
[8]姚雪婷,赵鹏,蒋玉艳,等.2011-2016年广西壮族自治区市售婴幼儿食品食源性致病菌监测结果分析[J].中国食品卫生杂志,2020,32(3):288-293.
YAO X T,ZHAO P,JIANG Y Y.Analysis of monitoring results of foodborne pathogens in infant food on the market of Guangxi Zhuang Autonomous Region in 2011-2016[J].Chinese Journal of Food Hygiene,2020,32(3):288-293.
[9]BARRANGOU R,FREMAUX C,DEVEAU H,et al.CRISPR provides acquired resistance against viruses in prokaryotes[J].Science,2007,315(5819):1709-1712.
[10]ISHINO Y,SHINAGAWA H,MAKINO K,et al.Nucleotide sequence of the iap gene,responsible for alkaline phosphatase isozyme conversion in Escherichia coli,and identification of the gene product[J].Journal of Bacteriology,1987,169(12):5429-5433.
[11]JANSEN R,EMBDEN J D A V,GAASTRA W,et al.Identification of genes that are associated with DNA repeats in prokaryotes[J].Molecular Microbiology,2002,43(6):1565-1575.
[12]CHERTOW,DANIEL S.Next-generation diagnostics with CRISPR[J].Science,2018,360(6387):381-382.
[13]BARRANGOU,RODOTPHE,FREMAUX,et al.CRISPR provides acquired resistance against viruses in prokaryotes[J].Science,2007,315(5819):1709-1712.
[14]MOLLA K A,YANG Y.CRISPR/Cas-mediated base editing:Technical Considerations and Practical Applications[J].Trends in Biotechnology,2019,37(10):1121-1142.
[15]张爱霞,朱庆锋,陈沛,等.基于CRISPR/Cas13 的RNA 编辑系统及其在核酸检测中的应用[J].广东农业科学,2020,47(11):273-281.
ZHANG A X,ZHU Q F,CHEN P,et al.RNA editing system based on CRISPR/Cas13 and its application in nucleic acid detection[J].Guangdong Agricultural Sciences,2020,47(11):273-281.
[16]PARDEE K,GREEN A,TAKAHASHI M,et al.Rapid,low-cost detection of zika virus using programmable biomolecular components[J].Cell,2016,165(5):1255-1266.
[17]WANG X,ZHONG M,LIU Y,et al.Rapid and sensitive detection of COVID -19 using CRISPR/Cas12a -based detection with naked eye readout,CRISPR/Cas12a-NER[J].Science Bulletin,2020,65(17):1436-1439.
[18]CHEN J S,MA E,HARRINGTON L B,et al.CRISPR-Cas12a target binding unleashes indiscriminate single -stranded DNase activity [J].Science,2018,360(6387):6245.
[19]HUANG W,YU L,WEN D,et al.A CRISPRCas12a-based specific enhancer for more sensitive detection of SARS-CoV-2 infection[J].EBioMedicine,2020,61:103036.
[20]HIDALGO - CANTABRANA C,O'FLAHERTY,SARAH,et al.CRISPR-based engineering of nextgeneration lactic acid bacteria[J].Current Opinion in Microbiology,2017,37:79-87.
[21]CHEN K,WANG Y,RUI Z,et al.CRISPR/Cas genome editing and precision plant breeding in agriculture[J].Annual Review of Plant Biology,2019,70(1):667-697.
[22]SELLE K,BARRANGOU R.CRISPR-based technologies and the future of food science[J].Journal of Food Science,2015,80(11):2367-2372.
[23]LI L,LI S,WU N,et al.HOLMESv2:a CRISPR-Cas12b-assisted platform for nucleic acid detection and DNA methylation quantitation[J].ACS Synthetic Biology,2019,8(10):2228-2237.
[24]TENG F,GUO L,CUI T,et al.CDetection:CRISPR-Cas12b-based DNA detection with sub-attomolar sensitivity and single-base specificity[J].Genome biology,2019,20(1):132.
[25]常超,王凌,伍金娥.基于ATP 再生体系快速检测乳品中微生物[J].食品科学,2018,39(4):320-324.
CHANG C,WANG L,WU J E.Development of a bioluminescence method combined with ATP amplification for detection of Bacteria in dairy products[J].Food Science,2018,39(4):320-324.
[26]PREECHAKASEDKIT P,PINWATTANA K,DUNGCHAI W,et al.Development of a one-step immunochromatographic strip test using gold nanoparticles for the rapid detection of Salmonella typhi in human serum[J].Biosensors & Bioelectronics,2012,31(1):562-566.
[27]林碧莲,柯振华,陈筱婷,等.多重实时荧光定量PCR 快速检测婴幼儿奶粉中沙门氏菌、克罗诺杆菌和金黄色葡萄球菌[J].食品安全质量检测学报,2017,8(11):4375-4381.
LIN B L,KE Z H,CHEN X T,et al.Simultaneous detection of Salmonella spp.,Staphylococcus aureus and Cronobacter in infant milk powder by multiplex quantitative real-time PCR[J].Journal of Food Safety & Quality,2017,8(11):4375-4381.
[28]李莉,陈泽辉.婴幼儿食品中阪崎克罗诺杆菌的快速检测[J].中国卫生标准管理,2019,10(18):4-7.
LI L,CHEN Z H.Rapid detection of Cronobacter Sakazakii in infant food[J].China Health Standard Management,2019,10(18):4-7.
[29]陈纯阳,张宸宁,史爱莹,等.重组酶等温扩增试纸条快速检测阪崎克罗诺杆菌[J].食品科学,2019,40(24):306-312.
CHEN C Y,ZHANG C N,SHI A Y,et al.Rapid detection of Cronobacter sakazakii using recombinase polymerase amplification combined with lateral flow strip[J].Food Science,2019,40(24):306-312.
[30]ZHANG Y,YANG Q,LI C,et al.Sensitive and visual detection of Cronobacter spp.in powdered infant formula by saltatory rolling circle amplification method[J].LWT,2019,107:41-48.