腐败希瓦氏菌(Shewanella putrefaciens)是一种革兰氏阴性运动杆菌,在自然界分布广泛,是水产品的一种特定腐败菌[1]。腐败希瓦氏菌具有较强的低温适应能力,能在冷藏水产品中生长繁殖成为优势腐败菌,同时产生腐胺、尸胺等生物胺以及三甲胺、H2S 等异味物质,并导致水产品表面黏滑、肌肉松散、品质恶化,是引起冷藏水产品发生腐败的重要因素之一[1]。据报道,腐败希瓦氏菌是海鲈鱼[2]、大黄鱼[3]、凡尔纳对虾[4]、斑点叉尾鮰[5]等水产品的优势腐败菌。鉴于此,腐败希瓦氏菌逐渐成为水产品保鲜领域内的研究焦点,尤其是在冷藏条件下致腐败的分子机制研究。
微生物对不同环境进行感知和响应一般是通过双组分信号转导系统(Two-component signal transduction system,TCSs)完成的[6]。TCSs 是一种信号转导系统,目前只在细菌、古生菌和植物中发现[7]。TCSs 可协助细菌适应复杂多变的外部环境,可以提高细菌在不同环境下的生存能力[8]。TCSs使细菌能够检测环境信号,并将其与应激响应所需的基因表达和生化反应过程联系起来[9]。细菌中的TCSs 包含一个组氨酸激酶(Histidine protein kinase,HK)和一个同源的反应调节蛋白(Response regulator protein,RR),这是简单的组氨酸-天冬氨酸组合形式。HK 在感知信号时发生自身磷酸化,并将磷酸基团转移到RR 的保守天冬氨酸残基上,RR 接收磷酸基团后自身构象发生变化,从而执行输出功能[10-12]。TCS 基因在细菌基因组中所占比例通常低于5%,然而具有众多重要的功能[8]。根据现有报道,在食源性细菌中,MalNO[13]和RevSR[14]可分别影响产气荚膜梭状芽孢杆菌对麦芽糖的利用水平以及毒力调节。CesKR 和AgrAC 可影响单增李斯特菌对抗生素的抗性[15]。VraSR[16]和SrrAB[17]可分别影响金色葡萄球菌的耐药性与致病性。
目前,关于腐败希瓦氏菌TCSs 的研究较少。本文从NCBI 数据库获得来自水产品的腐败希瓦氏菌WS13 基因组信息,对其TCSs 进行筛选和鉴定,并对TCSs 的功能域、跨膜区、进化关系进行分析。同时,结合互作蛋白分析和KEGG 数据库对TCSs 功能进行预测,全面分析水产品腐败希瓦氏菌TCSs 的特征和功能,以期为抑制水产品中的腐败希瓦氏菌提供新的研究思路。
1 材料与方法
1.1 基因组数据的获得
通过美国国立生物技术信息中心数据库(National Center for Biotechnology Information,NCBI,https://www.ncbi.nlm.nih.gov/)检索目前已有的腐败希瓦氏菌基因组信息,下载腐败希瓦氏菌WS13(NZ_CP028435.1)、腐败希瓦氏菌CN-32(NC_009438.1)、腐败希瓦氏菌FDAARGOS_681(NZ_CP046329.1)、腐败希瓦氏菌NCTC12093(NZ_LR134303.1)、腐败希瓦氏菌 200(NC_017566.1)的蛋白组和基因组序列,其中腐败希瓦氏菌WS13(NZ_CP028435.1)是从南美白对虾中分离得到的[5],被主要用于TCSs 的挖掘和表征,其它腐败希瓦氏菌基因组序列信息用于比较TCSs 在不同腐败希瓦氏菌菌株中的分布差异。
1.2 TCSs 的全基因组鉴定
参照师舷等[18]的方法,根据双组份信号系统HK 和RR 蛋白保守结构域分析腐败希瓦氏菌WS13 基因组中可能存在的TCSs。本研究中的HK和RR 结构域来自Pfam 蛋白数据库(http://pfam.xfam.org/),分别为 HATPase_c(Pfam00512、Pfam01627、Pfam02518、Pfam02895、Pfam06580、Pfam07536、Pfam07568、Pfam07730)和Response_reg(Pfam00072)。使用欧洲生物信息学研究所(European Molecular Biology Laboratory -European Bioinformatics Institute,EMBL-EBI) 研发的HMMER 软件中的hmmbuild 命令,将获取的HK 和RR 保守域对比序列转换成隐马尔科夫模型,随后使用hmmsearch 命令对腐败希瓦氏菌WS13 蛋白组进行搜索比对,分别鉴定基因组中的HKs 和RRs。通过NCBI 的BLASTp 程序对搜到的HK 和RR 进行确认。基因间距小于300 bp 的HK 和RR成功配对为成对TCS。如果HK 和RR 结构域同时出现在一个蛋白序列位置上,那么该蛋白定义为融合HK;如果HK 和RR 找不到相连的RR 和HK,则该单个的HK 和RR 定义为孤儿HK 和孤儿RR。
1.3 结构与功能分析
利用SMART(http://smart.embl-heidelberg.de/)来分析TCS 蛋白的功能域。TMHMM Serverv.2.0(http://www.cbs.dtu.dk/services/TMHMM-2.0/)用于预测蛋白质跨膜螺旋结构域。利用MEGA7.0 软件的MUSCLE 进行多重序列比对,并采用NJ 法构建进化树。利用BLASTp(https://blast.ncbi.nlm.nih.gov)程序进行序列相似性比对分析,并进行功能注释,预测分析各蛋白的功能。利用STRING(https://string-db.org/)筛选TCSs 的互作蛋白。从KEGG 数据库中获取TCS 的功能信息,同时参考文献报道对各TCS 进行功能预测。
2 结果与讨论
2.1 腐败希瓦氏菌WS13 基因组中TCSs 的分布
利用保守结构域对腐败希瓦氏菌WS13 全基因组中的TCSs 进行搜索,结果表明,腐败希瓦氏菌WS13 菌株具有31 个HKs 和36 个RRs,其中HK 与RR 成对的TCS 有3 对,融合HK 为3 个,孤儿HKs 和RRs 分别为25 个和33 个,操纵子上排列方式均为RR-HK(表1)。不同腐败希瓦氏菌株中孤儿HK/RR 的数量相近,而腐败希瓦氏菌WS13 中成对的TCSs 数量明显少于其它4 株腐败希瓦氏菌,并且缺少HK-RR 排列方式的TCS,这可能与腐败希瓦氏菌WS13 的基因组全长小于其它4 株菌有关(表2)。此外,这种TCS 多样性差异可能与菌株的遗传背景有关。腐败希瓦氏菌CN-32、腐败希瓦氏菌FDAARGOS_681 以及腐败希瓦氏菌200 这3 株菌分离自无机物(岩石、管道)环境,其融合HK 数量均为5,多于腐败希瓦氏菌WS13 中融合HK 的数量,与脑脊髓液来源的腐败希瓦氏菌NCTC12093 相差较大。融合HK 较成对的TCSs 对信号的响应更加灵活,可更及时、准确地对细菌中各信号通路进行调控[19]。水产品中的腐败希瓦氏菌可分解动物肌肉中的肌原蛋白;无机环境中的腐败希瓦氏菌需要面对极端环境,同时还能广泛利用各种物质,其中包括具有强毒性的环境污染物、氯代有机污染物、重金属和放射性元素[20]。脑脊髓液中的腐败希瓦氏菌则要面对和适应复杂的宿主环境。环境和利用碳源的不同,可能导致这几株菌之间遗传背景存在差异,从而产生TCSs 分布的差异。
表1 腐败希瓦氏菌WS13 基因组中TCSs 的蛋白ID 和类型
Table 1 Protein ID and types of TCSs in the whole genome of S.putrefaciens WS13

?TCS 类型 蛋白质ID TCSs(RR-HK)(01)WP_011789576.1/WP_011789577.1;(02)WP_025007618.1/WP_025007619.1;(03)WP_025007933.1/WP_025007934.1融合HK(01)WP_011788995.1;(02)WP_025007562.1;(03)WP_128090270.1孤儿HK(01)WP_011787427.1;(02)WP_011787496.1;(03)WP_011790295.1;(04)WP_011790877.1;(05)WP_011918470.1;(06)WP_011918496.1;(07)WP_011919441.1;(08)WP_011919453.1;(09)WP_014609564.1;(10)WP_025007509.1;(11)WP_025007695.1;(12)WP_025007839.1;(13)WP_025007861.1;(14)WP_025008336.1;(15)WP_025008530.1;(16)WP_025008575.1;(17)WP_061782889.1;(18)WP_061782945.1;(19)WP_061783040.1;(20)WP_061783060.1;(21)WP_061783061.1;(22)WP_061783391.1;(23)WP_128090133.1;(24)WP_128090183.1;(25)WP_128090199.1孤儿RR(01)WP_006079718.1;(02)WP_007644695.1;(03)WP_011623460.1;(04)WP_011787535.1;(05)WP_011787598.1;(06)WP_011787648.1;(07)WP_011787994.1;(08)WP_011787998.1;(09)WP_011788017.1;(10)WP_011788181.1;(11)WP_011788245.1;(12)WP_011788854.1;(13)WP_011789087.1;(14)WP_011789095.1;(15)WP_011789255.1;(16)WP_011790420.1;(17)WP_011790737.1;(18)WP_011790767.1;(19)WP_011790889.1;(20)WP_011791129.1;(21)WP_011918560.1;(22)WP_014611380.1;(23)WP_025007657.1;(24)WP_025007729.1;(25)WP_025008408.1;(26)WP_025008415.1;(27)WP_025008418.1;(28)WP_025008490.1;(29)WP_025008546.1;(30)WP_061782828.1;(31)WP_128090206.1;(32)WP_128090283.1;(33)WP_128090291.1
表2 腐败希瓦氏菌WS13 与其它菌株基因组中TCSs 分布情况比较
Table 2 Comparison of TCSs distribution in genomes WS13 and other strains of S.putrefaciens
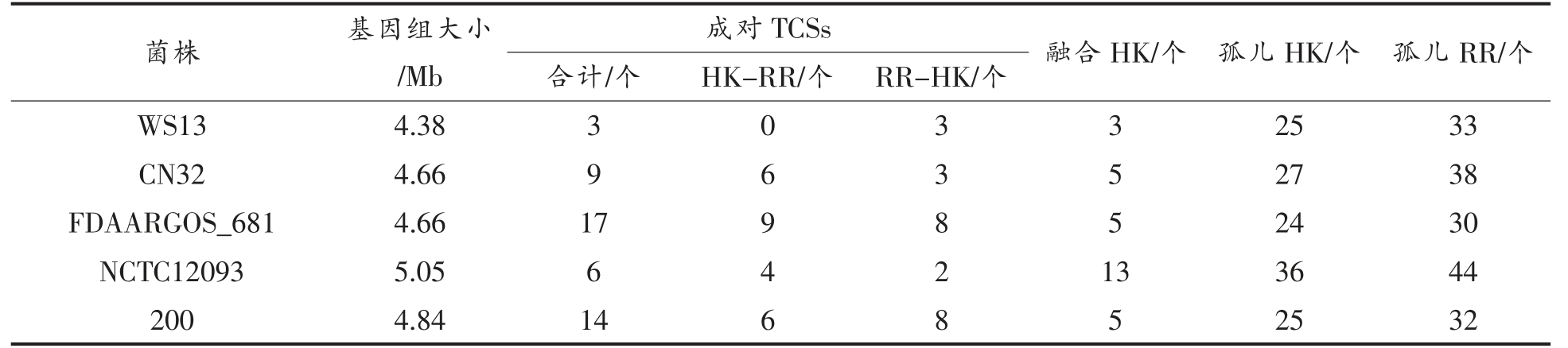
成对TCSs 融合HK/个 孤儿HK/个 孤儿RR/个合计/个 HK-RR/个 RR-HK/个WS13 4.38 3 0 3 3 25 33 CN32 4.66 9 6 3 5 27 38 FDAARGOS_681 4.66 17 9 8 5 24 30 NCTC12093 5.05 6 4 2 13 36 44 200 4.84 14 6 8 5 25 32菌株 基因组大小/Mb
2.2 HKs 的结构域组成和分类
SMART 蛋白结构域分析结果显示:腐败希瓦氏菌WS13 基因组中的31 个HKs 具有16 种结构域组成形式(图1)。11 种结构域组成形式中HKs的N 端为跨膜区,HKs 的跨膜区与其感受胞外信号有关。在成对的TCSs 中,跨膜区数量为1~3 个,而融合HKs 的跨膜区数量为1~12 个,其中融合HK01 的跨膜区为12 个。28 个HKs 的C 端具有典型的HATPase_c 激酶催化结构域;而另外3 个孤儿HK01、孤儿HK03 和孤儿HK17 通过BLASTp 分析发现具有类似HATPase_c 激酶催化结构域;少部分HKs 的C 端具有CheW 接受域、DNA 修复结构域、MutL_c 终端二聚体结构域;融合HKs 还具有REC 结构域。
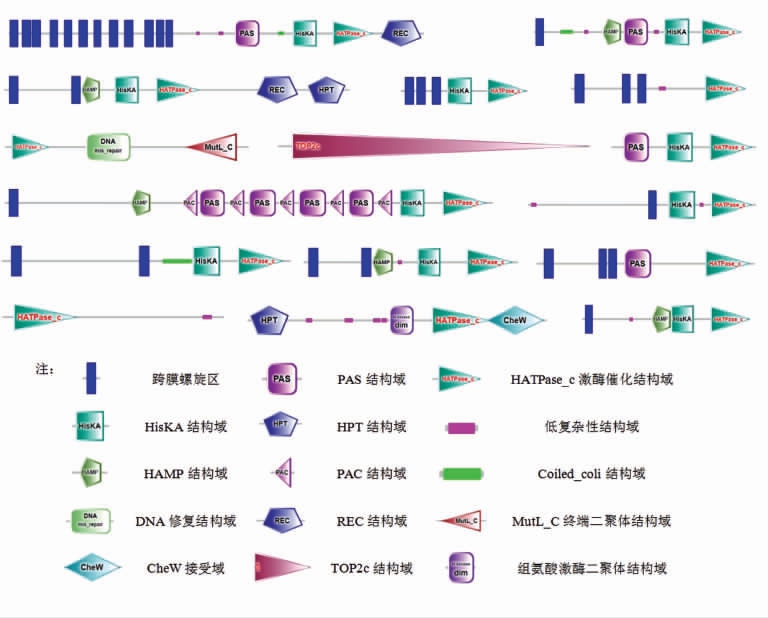
图1 腐败希瓦氏菌WS13 中HKs 的结构域组成
Fig.1 Structural domain composition of HKs in S.putrefaciens WS13
典型的HK 有3 个组成部分在发挥重要的生物学功能,第1 部分为N 末端可感应外界变化;第2 部分负责连接N 末端和C 末端区域;第3 部位为具有组氨酸残基的C 端激酶结构域。此外,还有一种杂合的HK,即除了典型的HK 结构域外,同时还含有一个天冬氨酸接受结构域[21-22]。根据结构域和跨膜区可将HKs 分为:含有PAS 和GAF 结构域的胞质感应HK;含有跨膜区和HAMP 结构域的胞外感应HK;以及仅含有跨膜区和HATPase_c 的膜感应HK[8]。根据分析结果,腐败希瓦氏菌WS13 中的HKs 绝大多数是胞外感应和膜感应类型。在结构域组成上,同时含有HisKA 和HATPase_c 结构域的HKs 有12 个,同时含有HAMP、HisKA 和HATPase_c 结构域的HKs 有8个,其余的HKs 具有多种结构域组成,如PAS、PAC、HPT、H-Kinasedim 等结构域。HisKA 是磷酸受体结构域;HAMP 域是二聚体的四螺旋束,可能调节同型二聚体受体的磷酸化或甲基化作用,可在细菌受体中转导构象信号[23]。PAS 结构域作为细胞内传感器,在包括信号转导、发色团形成、运动、孢子形成和氮固定等不同方面中发挥作用[24]。PAC 可修饰PAS,可能有助于PAS 的折叠[15]。除了常规结构域的HKs 外,孤儿HK01 具有TOP2c结构域,与DNA 拓扑异构酶Ⅱ功能相关。孤儿HK10 的C 端有一个DNA 错配修复蛋白结构域。一个MutL_C 末端二聚结构域,这是DNA 修复机制中纠正复制错误的关键组件。孤儿HK21 的N端为HPT 结构域,中部有H-Kinase dim 结构域,C 端有一个CheW 接受域,其结构与张娜等[15]发现的单増李斯特菌中HK02(RS05925) 结构相似。HPT 结构域可介导HKs 和RRs 间的信号传递。H-Kinase dim 是含有该结构域的组氨酸激酶形成二聚体的区域;CheW 参与细菌的趋化性信号传导[15]。
2.3 RRs 的结构域组成和分类
与HKs 相比,RRs 在结构域的组成上相对保守,它由N 端高度保守的含天冬氨酸残基的磷酸受体接受域(REC),以及C 端的输出结构域两部分组成[7]。TMHMM 的跨膜区分析结果显示RRs 均不具备跨膜区。腐败希瓦氏菌WS13 基因组中共有36 个RRs,然而,只有10 种结构域组成形式(图2)。每种结构域组成形式中均含有REC 结构域,表明磷酸受体结构域对RRs 是必需的。15 个RRs 属于REC 和Transreg_C 两种结构域组成形式。1 个RRs 属于2 个REC 结构域组成形式。23个RRs 属于由单个REC 结构域或REC 结构域与其它C 端的输出结构域组成形式。C 端的输出结构域种类主要包括CheW 结构域、AAA 结构域、HTH_LUXR 结构域、EAL 结构域、GGDEF 结构域和HDc 结构域。不同的C 端的输出结构域赋予RRs 的不同的功能。例如,CheW 结构域与细菌趋化性信号传导有关;AAA 结构域是与多种细胞活动相关的ATPase;HDc 结构域是具有保守结构的金属依赖性磷酸水解酶,包含环状核苷酸磷酸水解酶;HTH_LUXR 结构域,它可激活生物发光操纵子,也可能与DNA 转录作用相关;EAL 结构域,具有二鸟苷酸磷酸二酯酶活性;GGDEF 结构域具有二鸟苷酸环化酶活性[25]。
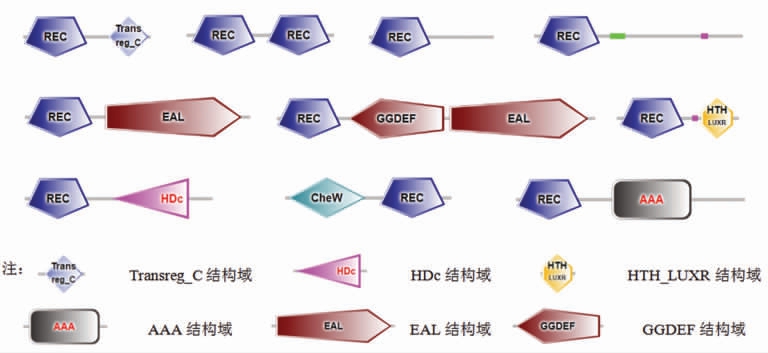
图2 腐败希瓦氏菌WS13 中RRs 的结构域组成
Fig.2 Structural domain composition of RRs in S.putrefaciens WS13
2.4 HKs 和RRs 进化分析
为了解腐败希瓦氏菌WS13 基因组中TCSs的进化关系,分别对所有HKs 和RRs 进行多序列比对和进化分析(图3a,3b)。从进化关系来看,HKs 主要分为4 个聚簇(图3a),不同结构域的HKs 具有不同的进化关系。成对TCSs 中的HK1(WP_011789577.1)和HK3(WP_025007934.1)聚在一起,具有较近的亲缘关系,而HK2(WP_025007619.1)位于另外一个聚簇。3 个融合HK,分别位于3 个聚簇,表明其亲缘关系较远,可能具有不同功能特点。从结构域组成和序列比对分析来看,RRs 的保守性比HKs 要强一些,其进化树主要划分为6 个聚簇(图3b)。成对TCSs 中的 RRs 分别位于不同聚簇,其中 RR1(WP_011788995.1)和RR3(WP_128090270.1)分别位于第5 和第6 聚簇,表明其亲缘关系更近,这与HK 的分析结果类似。位于同一聚簇的HKs 和RRs 显示较近的亲缘关系和高度的同源性,它们更可能具有相近的功能。
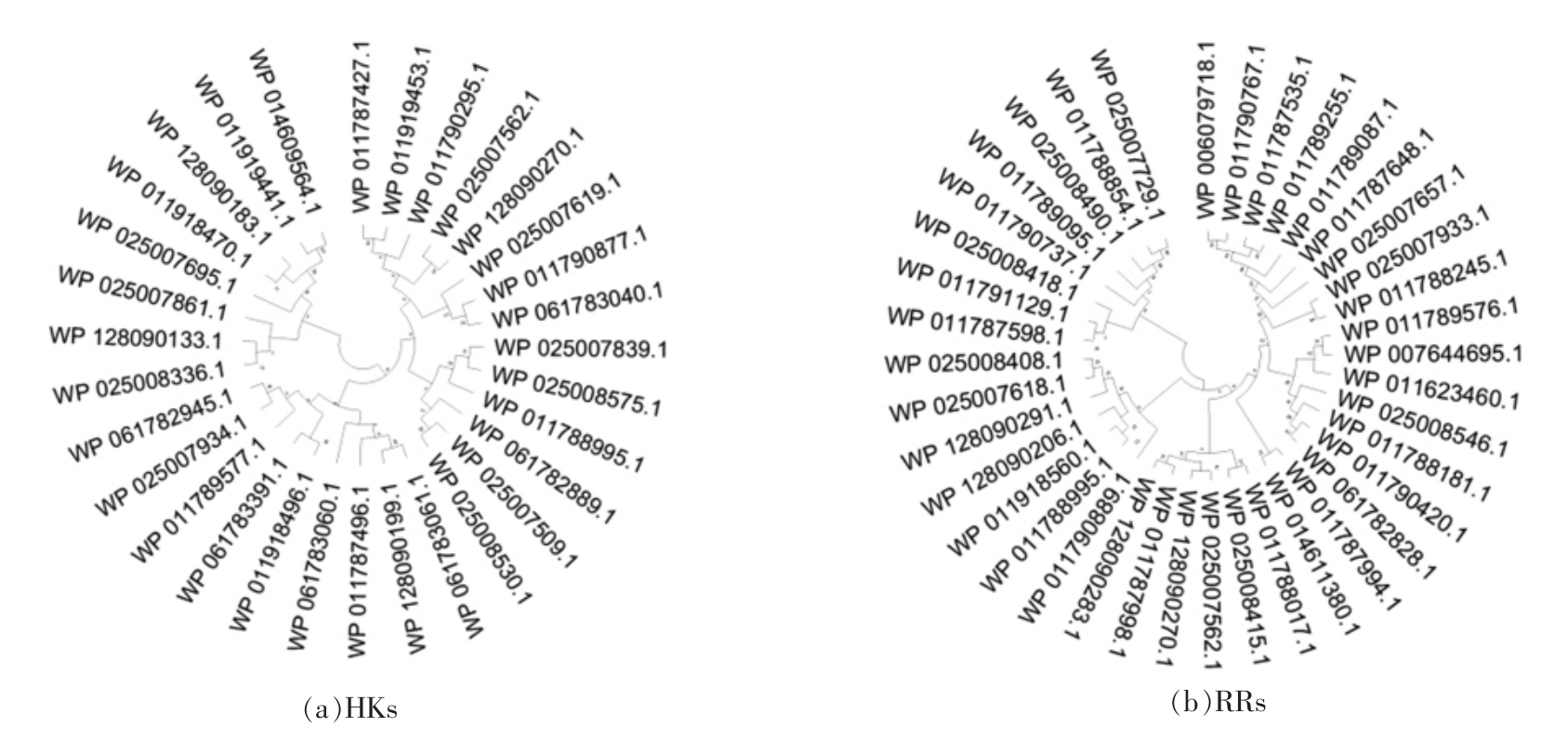
图3 腐败希瓦氏菌WS13 中HKs 和RRs 的系统进化分析
Fig.3 Phylogenetic analysis of HKs and RRs in S.putrefaciens WS13
2.5 成对TCSs 和融合HKs 的互作蛋白分析及功能预测
利用STRING 对腐败希瓦氏菌WS13 成对TCSs 和融合HKs 的互作蛋白进行分析,结果如图4所示。每种TCSs 与多种蛋白存在相互作用,通过Blast P 和KEGG 蛋白数据库注释对其功能进行预测和分析(表3)。与TCSs01 存在相互作用的蛋白主要有pdsR 蛋白酶(ABP75463.1)、组氨酸激酶(ABP76854.1、ABP75462.1、ABP76854.1、ABP77677.1)、分选酶(ABP75465.1)、分选酶靶蛋白(ABP75464.1)、次黄嘌呤磷酸核糖基转移酶(ABP76895.1)。BLASTp 结果表明:TCSs01 为PdsR/PsdS 系统,目前该TCS 系统在细菌中的功能未知,结合KEGG 数据库结果可推测TCSs01 主要含有多糖结合位点,可影响细胞壁代谢(表3)。预测TCSs02 的互作蛋白包括组氨酸激酶(ABP76854.1、ABP76486.1)、转运蛋白(ABP76707.1)、分选酶靶蛋白(ABP76704.1)、功能未知的DUF 家族蛋白(ABP76703.1)、ABC 转运蛋白蛋白(ABP76705.1)、RNA 聚合酶因子(ABP74445.1)、铁氧化还原蛋白(ABP74700.1)、二硫键交换蛋白(ABP74702.1)。BLASTp 注释分析TCSs02RR 可能属于NtrC 亚家族,推测TCSs02 的功能可能同NtrX/NtrY,该TCS 在苜蓿中华根瘤菌和变形红细菌中可影响胞外被膜的生成并影响细胞形态[26]。此外,可能影响细胞壁代谢和乙酰乙酸代谢。TCSs03 可能的互作蛋白包括转录调节因子(ABP73784.1、ABP76790.1)、钾转运 ATP 酶(kdpB、kdpC、kdpA)、整合膜传感器组氨酸激酶(ABP73785.1)、K +通道信号转导组氨酸激酶(ABP76791.1)、整合膜传感器组氨酸激酶(ABP75803.1)、组氨酸激酶(ABP76854.1、ABP75703.1)。注释分析表明TCSs03 的功能同kdpDE,包括感应K+信号刺激、调节渗透压等。与融合HK01 存在相互作用的蛋白包括组氨酸激酶(ABP76854.1)、LuxR 家族的转录调节子(ABP75968.1)、L-脯氨酸脱氢酶(ABP76812.1)、乙酰辅酶A 合成酶(acsA)、CheA 信号转导组氨酸激酶(ABP76277.1)。注释分析表明融合HK01 可能参与Na+/脯氨酸转运,并与三甲胺形成相关。与融合HK02 存在互作的蛋白包括组氨酸激酶(ABP76040.1)、应答调节剂受体调节的CheW 蛋白(ABP76214.1、ABP76517.1、ABP76320.1)、转录调节子(ABP75664.1)。融合HK02 含有WalK(NF033092)超家族的保守结构,是一种细胞壁代谢传感器组氨酸激酶,结合BLASTp 结果,推测融合HK02 可能参与渗透压调节,并与细胞壁代谢、三甲胺形成相关。融合HK03 的互作蛋白可能包括组氨酸激酶(ABP76854.1、ABP76040.1)、LuxR家族的转录调节子(ABP75364.1)、双鸟苷酸环化酶(ABP76798.1)、应答调节剂受体调节的CheW蛋白(ABP76214.1)。注释分析表明融合HK03 可能参与碳代谢的调节,并与三甲胺形成相关。3 个融合HK 均有TorS 结构域,在希瓦氏菌中,Tor 在缺氧环境下参与氧化三甲胺呼吸,表明可能与三甲胺生成相关[27]。碳源的利用影响细菌的生长繁殖,融合HK 与氧化三甲胺的还原有关,这可能有助于腐败希瓦氏菌的营养物质摄入。
表3 腐败希瓦氏菌WS13 TCSs 功能预测
Table 3 Functional prediction of TCSs in S.putrefaciens WS13

?
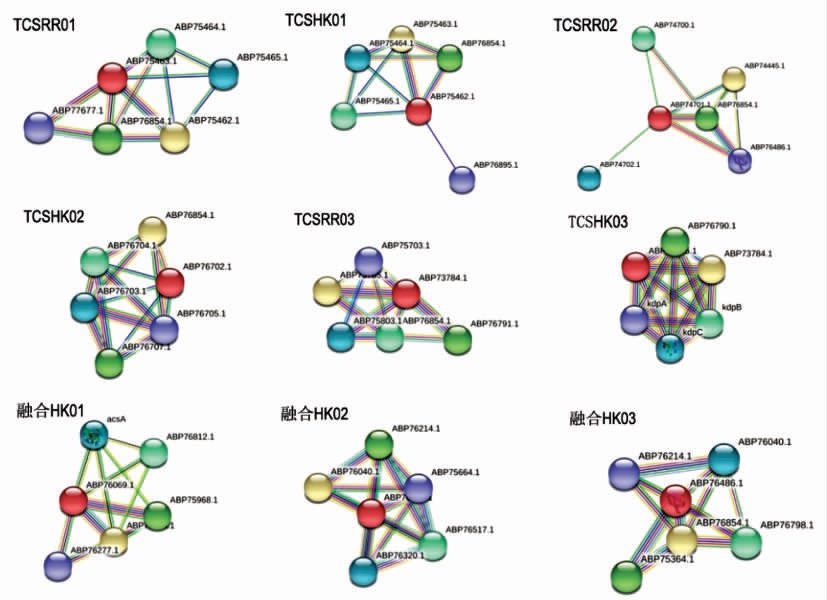
图4 腐败希瓦氏菌WS13 TCSs 的互作蛋白
Fig.4 Interaction proteins of TCSs in S.putrefaciens WS13
2.6 孤儿HKs 和RRs 的功能预测
通过Blast P 和KEGG 蛋白数据库注释对孤儿HKs 和RRs 的功能进行预测和分析。25 个孤儿HK 预测的功能主要包括:参与DNA 解旋、错配修复;响应渗透压、氮信号、金属离子和重金属信号刺激;调节三甲胺形成、C4-二羧酸盐转运、有氧呼吸过程、分子伴侣表达、磷酸盐代谢以及柠檬酸/苹果酸代谢。2 个孤儿HK 与3 个融合HKs 一样具有TorS 结构域,可能与三甲胺生成相关[27]。
33 个孤儿RR 主要属于OmpR、CheB、CheC、CheW、CheY、NarL、NtrC 等亚家族,预测其主要功能包括:控制细菌运动能力、磷酸化介导起始、调节DNA 结合、转录和乙酰乙酸代谢等。OmpR 是孤儿RR 中最多的亚家族,也是最大的RR 蛋白家族,在伤寒杆菌中可参与酸化,调节细胞内pH值,而未见其在希瓦氏菌属中的报道[28]。其它部分孤儿RR 分属于CitB、PhoB、PhoP、FixJ、KdpE、RpfG、RssB、RstA、RcpA 等亚家族,预测功能包括:参与氧化还原反应、细菌色素调节;调节细胞表面的黏附性、磷酸盐转运以及柠檬酸/苹果酸代谢;影响细菌毒力和致病性等。
3 结论
采用生物信息学方法对腐败希瓦氏菌WS13的TCSs 进行全基因组鉴定、结构域组成分析和功能预测。腐败希瓦氏菌WS13 菌株含有3 对TCSs,3 个融合HK,25 个孤儿HKs 和33 个孤儿RRs。这些HKs 和RRs 的结构域组成类型丰富。其中,HKs 具有16 种结构域组成,大多数HKs 的N 末端具有接受信号的信号输入结构域,如跨膜区、HisKA、HAMP、PAS、PAC 等结构域。RRs 有10种结构域组成,并主要属于OmpR、CheB、CheC、CheW、CheY、NarL、NtrC 等亚家族。腐败希瓦氏菌WS13 中的成对的TCSs 与融合HKs 的数量较少,同时与多种蛋白存在相互作用。研究结果呈现了腐败希瓦氏菌WS13 全基因组中TCSs 的整体情况,为后续开展TCSs 在腐败希瓦氏菌中的功能研究及水产品腐败希瓦氏菌新型抑制剂的筛选提供一定的参考。
[1]杨胜平,章缜,程颖,等.培养温度对腐败希瓦氏菌DSM6067 生长动力学及细胞膜理化特性的影响[J].中国食品学报,2020,20(2):228-236.
YANG S P,ZHANG Z,CHENG Y,et al.Effects of culture temperature on the growth parameters of Shewanella putrefaciens DSM6067 and its membrane physic-chemical properties[J].Journal of Chinese Institute of Food Science and Technology,2020,20(2):228-236.
[2]朱金帅,李秋莹,于昕睿,等.酚酸对水产品腐败希瓦氏菌的抑菌作用[J].食品与机械,2020,36(6):133-137.
ZHU J S,LI Q Y,YU X R,et al.Antibacterial effect of phenolic acid on Shewanella putrefaciens from aquatic product[J].Food and Machinery,2020,36(6):133-137.
[3]朱军莉,赵二科,孙丽霞,等.冷藏大黄鱼腐败菌群和群体感应信号分子的动态变化规律[J].中国食品学报,2015,15(4):175-182.
ZHU J L,ZHAO E K,SUN L X,et al.Dynamic variation rule of spoilage bacterial and quorum sensing signal molecules in the refrigerated large yellow croaker[J].Journal of Chinese Institute of Food Science and Technology,2015,15(4):175-182.
[4]CHEN L,YANG S,QIAN Y,et al.Sequencing and Analysis of the Shewanella putrefaciens WS13 Genome[J].Journal of Biobased Materials and Bioenergy,2019,13(2):182-187.
[5]YANG Z Q,TAO X Y,ZHANG H,et al.Isolation and characterization of virulent phages infecting Shewanella baltica and Shewanella putrefaciens,and their application for biopreservation of chilled channel catfish(Ictalurus punctatus)[J].International Journal of Food Microbiology,2019,292:107-117.
[6]SANDEEP T,JAMAL S B,HASSAN S S,et al.Two -component signal transduction systems of pathogenic bacteria as targets for antimicrobial therapy:An overview[J].Frontiers in Microbiology,2017,8:1878.
[7]JACOB-DUBUISSON F,MECHALY A,BETTON J M,et al.Structural insights into the signaling mechanisms of two-component systems [J].Nature Reviews Microbiology,2018,16(10):585-593.
[8]李秋莹,张东栋,王司雯,等.食源性细菌低温适应的分子机制研究进展[J].食品与发酵工业,2019,45(5):246-252.
LI Q Y,ZHANG D D,WANG S W,et al.Advances in molecular mechanisms of cold-adapting foodborne bacteria[J].Food and Fermentation Industries,2019,45(5):246-252.
[9]WUICHET K,CANTWELL B J,ZHULIN I B.Evolution and phyletic distribution of two-component signal transduction systems[J].Current Opinion in Microbiology,2010,13(2):219-225.
[10]LEMMER K C,ALBERGE F,MYERS K S,et al.The NtrYXtwo-Component system regulates the bacterial cell envelope[J].Molecular Biology and Physiology,2020,11(3):e00957-20.
[11]DHAR Y V,LAKHWANI D,PANDEY A,et al.Genome-wide identification and interactome analysis of members of two-component system in banana[J].BMC Genomics,2019,20(1):674.
[12]MANASI P B,MOLNAR K S,GOULIAN M,et al.Signal transduction in histidine kinases:insights from new structures[J].Structure,2015,23(6):981-994.
[13]HISCOX T J,OHTANI K,SHIMIZUT,et al.Identification of a two -component signal transduction system that regulates maltose genes in Clostridium perfringens[J].Anaerobe,2014,30:199-204.
[14]CHEUNG J K,WISNIEWSKI J A,ADAMS V M,et al.Analysis of the virulence-associated RevSR two -component signal transduction system of Clostridium perfringens[J].International Journal of Medical Microbiology,2016,306(6):429-442.
[15]张娜,欧娅,郑金水,等.单增李斯特菌食品分离株LM201 的双组分信号转导系统的生物信息学分析[J].微生物学杂志,2018,38(1):96-104.
ZHANG N,OU Y,ZHENG J S,et al.Bioinformatics analysis of double-component signal transduction systems of Listeria monocytogenes LM201 isolated strain from food stuff[J].Journal of Microbiology,2018,38(1):96-104.
[16]AGUSTINA T,VARELA M C,ROSATO R R,et al.VraSR and virulence trait modulation during daptomycin resistance in methicillin-resistant Staphylococcus aureusinfection[J].mSphere,2019,4(1):e00557-18.
[17]TIWARI N,LÓPEZ -REDONDO M,MIGUEL -ROMERO L,et al.The SrrAB two-component system regulates Staphylococcus aureus pathogenicity through redox sensitive cysteines[J].Proceedings of the National Academy of Sciences of the United States of America,2020,117(20):10989-10999.
[18]师舷,刘佳璐,董极靓,等.解淀粉芽孢杆菌Q426双组分信号转导系统的生物信息学分析[J].生物技术,2019,29(4):348-354.
SHI X,LIU J L,DONG J L,et al.Bioinformatic analysis of two-component signal transduction systems in Bacillus amyloliquefaciens Q426[J].Biotechnology,2019,29(4):348-354.
[19]杨若兰.欧美杨细菌性溃疡病菌双组分系统的鉴定、突变及KdpD-KdpE 致病性功能解析[D].北京:北京林业大学,2018.
YANG R L.Identification and mutation of two-component systems and functional analysis of KdpDKdpE of Lonsdaleaquercina subsp.popul[D].Beijing:Beijing Forestry University,2018.
[20]潘婷婷.腐败希瓦氏菌介导合成纳米材料及其催化还原硝基苯的性能研究[D].杭州:浙江大学,2019.
PAN T T.Formation of nanomaterials mediated by Shewanellaputrefaciens and catalytic properties towards nitrobenzene[D].Hangzhou:Zhejiang University,2019.
[21]ZSCHIEDRICH C P,KEIDEL V,SZURMANT H.Molecular mechanisms of two -component signal transduction[J].Journal of Molecular Biology,2016,428(19):3752-3775.
[22]陈利达,黄彬.铜绿假单胞菌的双组分系统[J].中国微生态学杂志,2011,23(10):941-945.
CHEN L D,HUANG S.The two-component regula tory systems in Pseudomonas aeruginosa[J].Chinese Journal of Microecology,2011,23(10):941-945.
[23]SUKOMON N,WIDOM J,BORBAT P P,et al.Stability and conformation of a chemoreceptor HAMP domain chimera correlates with signaling properties[J].Biophysical Journal,2017,112(7):1383-1395.
[24]SARKAR I,NORMAND P,TISA L S,et al.Characterization of PAS domains in Frankia and selectedActinobacteria and their possible interaction with other co-domains for environmental adaptation[J].Symbiosis,2016,70(1):69-78.
[25]SCHAECHTER M.Encyclopedia of Microbiology(3rd Edn)[M].Pittsburgh:Academic Press,2009:447-463.
[26]NIEVES C M,JOAQUINA N,KINIA A,et al.The NtrY/NtrX System of Sinorhizobium meliloti GR4 regulates motility,EPS I production,and nitrogen metabolism but is dispensable for symbiotic nitrogen fixation [J].Molecular Plant -Microbe Interactions,2017,30(7):566-577.
[27]LEMAIRE O N,HONORÉ F A,JOURLIN -CASTELLI C,et al.Efficient respiration on TMAO requires TorD and TorE auxiliary proteins in Shewanella oneidensis[J].Research in Microbiology,2016,167(8):630-637.
[28]CHAKRABORTY S,WINARDHI R S,MORGAN L K,et al.Non-canonical activation of OmpR drives acid and osmotic stress responses in single bacterial cells[J].Nature Communications,2017,8(1):1587.