甲壳素作为常见的生物多糖,在自然界中含量排名第二,主要分离自虾壳、蟹壳、藻类细胞壁等,可通过脱N-乙酰基得到一种阳离子聚合物——壳聚糖,该聚合物是一种无毒、无味,半透明、略有光泽的白色粉末状固体物质,主要结构由D-氨基葡萄糖通过β-1,4-糖苷键连接而成,分子质量可达数十万到百万[1-2]。壳聚糖在化工、食品及其它行业有广泛的应用价值[3]。壳聚糖的高分子质量特性,使其在自然状态下非常稳定,因分子间具有很强的氢键作用而导致其水溶性差,只能溶于醋酸等一些无机酸中,难以直接利用,故未经降解处理的壳聚糖在实际应用中会受到限制[2,4]。
壳聚糖酶(EC 3.2.1.132)是一种常用于降解壳聚糖的糖苷水解酶[5],广泛分布于细菌、真菌中[6-7]。该酶通过内切方式水解乙酰化壳聚糖的β-1,4-氨基葡萄糖苷键生成壳寡糖(或称低聚壳聚糖)[8],这种物质具有低分子质量、低黏度、水溶性好的特性[9],不仅弥补壳聚糖的不足,而且具有特殊的生理活性和功能[10]。例如可调节脂肪代谢基因,减少肝毒性脂质、非必需脂肪细胞在体内的过度积累[11]。目前已有多种生物的壳聚糖酶基因在大肠杆菌中实现了重组表达。Lin 等[12]利用大肠杆菌作为宿主实现了枯草芽孢杆菌NCHU-05 的壳聚糖酶基因重组表达,酶比活力分别为3 655 U/mg 和3 780 U/mg。Kilani-Feki 等[13]将一株耐热型枯草芽孢杆菌csnv26 的壳聚糖酶在大肠杆菌中表达,重组菌的酶产量高达6.2 g/L。段静[4]在大肠杆菌BL21 中构建并表达解淀粉芽孢杆菌壳聚糖酶与谷胱甘肽转移酶(Glutathione S- transferase,GST)的融合蛋白,由GST 亲和层析柱对重组酶完成高效纯化。
在壳聚糖的酶法降解处理过程中,会涉及高温和酸处理环节[14]。如何提高壳聚糖酶的稳定性,是该酶实现大规模应用过程中的主要问题。目前,通过微生物获得高稳定性的酶是最直接和最可靠的来源[15]。中村芽胞杆菌是一种革兰氏阳性菌,具有较强的环境耐受性,可在pH 5.5~10、环境温度17~50 ℃、高浓度盐溶液(9% NaCl)的环境中正常生长[16]。目前该菌株壳聚糖酶基因序列已被公开,然而未见有关该菌株的壳聚糖酶酶学性质的研究报道。本研究首次成功构建表达中村芽胞杆菌壳聚糖酶的重组菌,并对得到的重组酶进行酶学性质研究,以拓展壳聚糖酶酶库资源,为该酶的后续研究和应用提供理论依据。
1 材料与方法
1.1 材料与试剂
大肠杆菌BL21(DE3)、pGEX-4T-3 原核表达载体由广西科技大学发酵工程研究室提供;壳聚糖(脱乙酰度大于90%)、氨苄青霉素、低分子质量蛋白marker、异丙基-β-D-硫代半乳糖苷(Isopropyl-beta-D-thiogalactopyranoside,IPTG),北京索莱宝科技有限公司;质粒提取试剂盒、DNA 琼脂糖凝胶回收试剂盒,北京天根生化科技有限公司;DNA marker DL 2000、DNA T4 连接酶、限制性内切酶,BamHI 和sall TAKARA 公司;GSTSepharose FF 层析柱,北京韦氏博慧色谱科技有限公司。LB(Luria-Bertani)培养基:1%蛋白胨、0.5%酵母膏、1%氯化钠,pH 7.2~7.4;SOB(Super Optima1 Broth) 培养基:0.05%氯化钠,0.5%酵母浸粉,2%蛋白胨,无菌水定容100 mL,pH 7.0;1%壳聚糖溶液:壳聚糖0.1 g,乙酸-乙酸钠(pH 5.5)缓冲液10 mL;氨苄青霉素溶液:氨苄青霉素0.1 g,无菌水定容2 mL,过滤除菌;IPTG 溶液:IPTG 0.119 g,无菌水定容5 mL。柱层析纯化试剂:缓冲液1:Tris 碱0.6 g、NaCl 0.9 g、无菌水定容至100 mL,HCl 调pH 值至8.3;缓冲液2:Tris 碱 0.61 g、还原型谷胱甘肽0.641 g,无菌水定容至100 mL,HCl 调pH 值至8.3,4 ℃保存。
1.2 仪器与设备
电子天平,赛多利斯科学仪器;UV-8000S 紫外可见分光光度计,上海元析仪器有限公司;H2100R 医用离心机,湖南湘仪实验室仪器开发有限公司;BLON-1000Y,超声波信号发生器,上海比郎仪器制造有限公司。
1.3 试验方法
1.3.1 中村芽胞杆菌壳聚糖酶基因克隆与表达载体构建 根据GeneBank 数据库中中村芽胞杆菌壳聚糖酶的氨基酸序列(GeneBank:WP_061527864.1) 重新设计基因。在http://www.jcat.de/Start.jsp 网站针对大肠杆菌密码子偏好性进行优化,同时分别在5’和3’端添加酶切位点BamHI 和salI,重新设计得到的基因命名为ChiA,委托杭州弘赛生物科技有限公司使用pUC18 载体进行基因克隆载体的构建并进行测序,构建得到的载体命名为pUC18-ChiA。结果使用Vector NTI 软件对比验证。通过BamHI 和salI 双酶切处理pUC18-ChiA 基因克隆载体和pGEX-4T-3 表达载体得到线性载体与ChiA 基因,1%琼脂糖凝胶电泳观察结果并回收,回收片段用T4 连接酶连接,最终得到的重组表达载体命名为pGEX-4TChiA。
1.3.2 重组表达菌株构建和验证 将-80 ℃保存的感受态大肠杆菌细胞BL21(DE3)冰浴解冻,之后加入20 μL 重组载体pGEX-4T-ChiA,冰浴30 min 后转移到42 ℃恒温水浴锅中孵育120 s。将孵育后的细胞悬液置于冰水浴2 min,转入SOB 培养基并放置在37 ℃恒温水浴中静置30 min,37℃培养1 h。将上述成功转化的感受态细胞4 ℃离心(10 000 r/min,10 min),弃上清,加入100 μL PBS 缓冲液吹打混匀沉淀。吸取50 μL 细胞悬液涂布于LB 固体培养基 (Amp 质量浓度80 μg/mL),37 ℃倒置培养12 h,挑选阳性克隆,经PCR、双酶切以及测序确认,构建成功的重组表达菌株命名为大肠杆菌(E.coli BL21-pGEX-4T-ChiA)。
1.3.3 重组酶诱导表达和纯化 将重组表达载体转接到LB 液体培养基中 (Amp 质量浓度50 μg/mL),37 ℃培养至OD600nm 为0.4~0.6 时,加入诱导剂IPTG(终浓度0.07 mmoL/L),16 ℃诱导10 h。12 000 r/min,4 ℃离心10 min 收集菌体。加入30 mL PBS,吹打混匀,随后冰浴条件下超声细胞破碎,条件设置为功率100 W,工作2 s、停止2 s,时间10 min。细胞破碎液12 000 r/min,4 ℃离心15 min,取上清得重组酶粗酶液。重组酶已融合GST标签蛋白,因此使用亲和层析法对重组酶进行纯化,层析柱为GST-Sepharose FF。具体步骤如下:粗酶液经0.45 μm 滤膜过滤。加入7 个柱体积缓冲液1 平衡层析柱。将过滤好的上清液与缓冲液1 混匀,0.45 μm 滤膜过滤,上样。加入20 个柱体积的缓冲液1 清洗层析柱。加入5 mL 缓冲液2 进行洗脱,收集含有纯化的重组酶洗脱液。SDSPAGE 电泳检测纯化结果。
1.3.4 重组酶酶活和蛋白浓度测定 酶活采用3,5-二硝基水杨酸 (3,5-dinitrosalicylic acid,DNS)法进行测定[17],步骤如下:首先将100 μL 重组酶酶液、700 μL 乙酸-乙酸钠缓冲液 (pH 5.5)和200 μL 1%壳聚糖溶液(pH 5.5) 混匀后置于37 ℃水浴20 min,再加入2 mL DNS 溶液沸水浴7 min,最后用无菌水稀释至5 mL 体积并在470 nm 处测定OD 值。使用经高温灭活的酶液作为对照。酶活定义:在上述反应条件下1 min 内生成1 μmol 还原糖所需的酶量。蛋白浓度测定参照考马斯亮蓝法[18],以牛血清蛋白作标准蛋白。在6 组试管内分别加入不同体积的牛血清蛋白样品溶液(0,0.2,0.4,0.6,0.8,1 mL)、无菌水(1,0.8,0.6,0.4,0.2,0 mL)、相同体积的考马斯亮蓝溶液(5 mL),充分混匀后放置5 min,在595 nm 处测定各管吸光度值,以吸光度值为纵坐标,牛血清蛋白样品浓度为横坐标,绘制标准曲线,之后在比色管内将50 μL 纯化后的重组酶洗脱液与5 mL 考马斯亮蓝溶液混匀,室温静置5 min,将测得的595 nm 处的吸光度值代入标准曲线求得重组酶浓度。
1.3.5 酶学性质测定 最适温度: 将重组酶在25,30,34,37,41,46,51,55 ℃温度下进行酶促反应,按照DNS 法测定酶活,以测得的酶活最大值为100%,计算各温度下的相对酶活。
温度稳定性: 将重组酶在不同温度下保温15,30,45,60,80 min 之后在最适温度下进行酶促反应,按照DNS 法测定酶活,以各温度下未进行保温处理的酶活为100%,计算各温度下不同保温时间的相对酶活。
最适pH 值:将重组酶分别加入到乙酸-乙酸钠(pH 2.5,3.0,3.5,4.0,4.5,5.0,5.5,6.0,6.5)缓冲液及磷酸氢二钠-柠檬酸(pH 7.0,7.5,8.0)缓冲液中,按照DNS 法测定酶活。以测得的酶活最大值为100%,计算各pH 值下的相对酶活。
pH 稳定性: 将重组酶在不同pH 值下保温30,60 min,在最适温度下按照DNS 法测定酶活,以各pH 值下未进行保温处理的酶活为100%,计算各pH 值下不同保温时间的相对酶活。
不同金属离子的影响: 在最适酶促反应体系中加入浓度为5 mmol/L 的不同金属离子溶液(Fe3+、Cu2+、Zn2+、K+、Ca2+、Mn2+、Mg2+、Na2+、Ag+、Ni+、Hg+及EDTA),在37 ℃反应体系下温浴20 min,470 nm 处测吸光度值,以未处理的体系测得的酶活为对照,定义对照组酶活为100%。
1.3.6 数据处理 每组试验独立重复3 次,使用Excel 和Orign 软件(2020 版)进行数据处理和绘图,结果以平均值±标准差形式进行表示。
2 结果与分析
2.1 重组酶酶活及蛋白纯化浓度测定
重组酶诱导表达及纯化结果如图1所示,经纯化后得到一条相对分子质量大约为57 ku 的融合蛋白,其中含GST 标签蛋白26 ku,目的蛋白31 ku。由考马斯亮蓝法得蛋白质浓度线性回归方程y=4.36143x+0.0004,可得质量浓度为10.932 μg/mL。DNS 法测得酶活为6.135 U/mL,酶比活力561.19 U/mg。
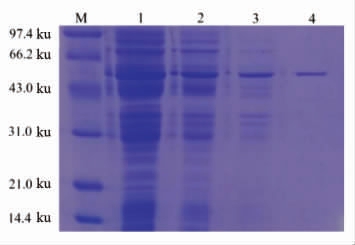
图1 SDS-PAGE 电泳分析
Fig.1 SDS-PAGE electrophoresis analysis
注:M:Marker;1:诱导后全蛋白;2:上清;3:沉淀;4:纯化后蛋白。
2.2 酶学性质测定
2.2.1 最适反应温度与温度稳定性 由图2a 可知不同温度下(25~55 ℃)重组酶的酶活力,其酶活具有先增加后降低的趋势,在37 ℃时酶活力最高,55 ℃时完全丧失活性。温度稳定性结果如图2b,重组酶在37 和42 ℃下具有极其良好的稳定性,在30 ℃时稳定性略有下降,但仍能保持较高水平酶活,而在51 ℃以上的环境中稳定性急剧下降,30 min 内即可导致酶活性丧失。
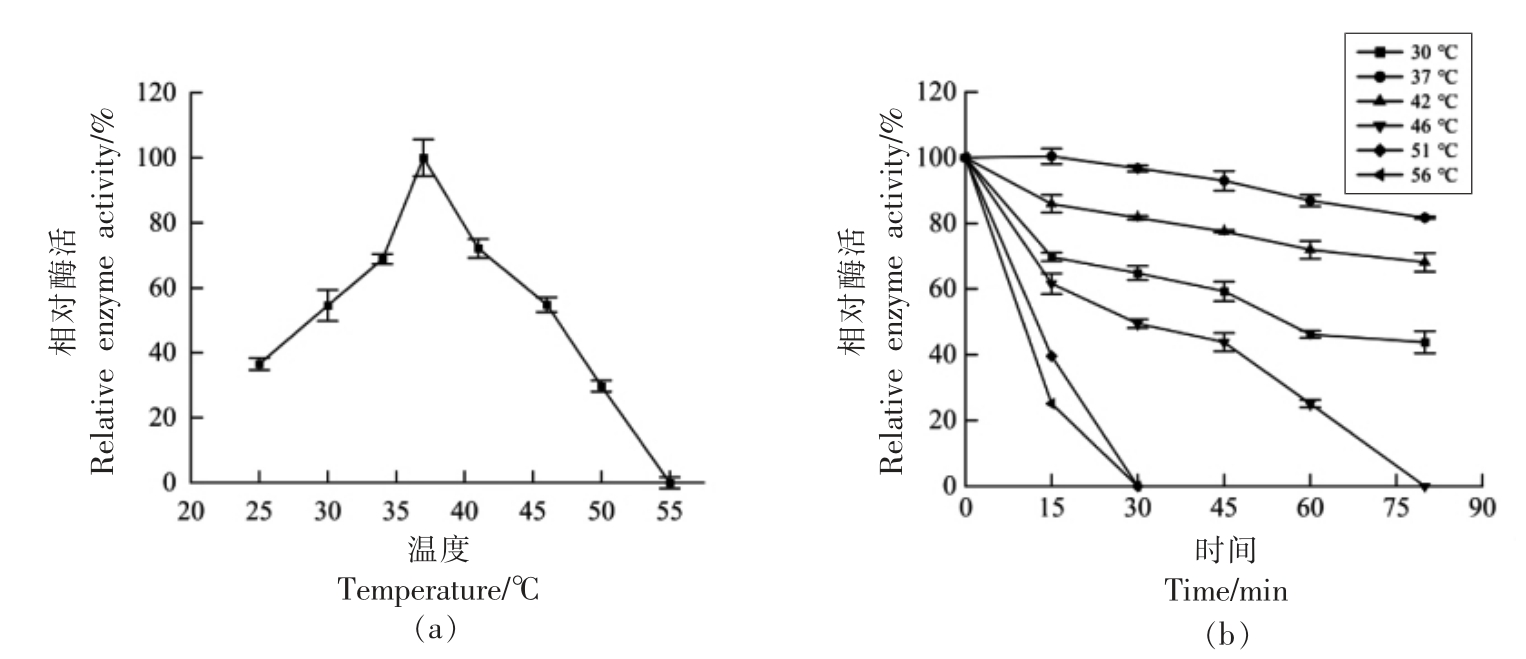
图2 壳聚糖酶最适反应温度(a)及温度稳定性(b)
Fig.2 Optimum reaction temperature of chitosanase (a),temperature stability of chitosanase (b)
2.2.2 最适pH 值和pH 值稳定性 由图3a 可知,重组酶在pH 4.5 时具有最高酶活性。pH 4.5~5 范围内具有80%以上的酶活性,随着pH 值继续升高,重组酶酶活力逐渐下降,pH 8.0 时酶活性基本丧失。由图3b 可知,在pH 2.5~6.5 的条件下处理30 min,酶活有一定程度的下降,但整体剩余的酶活仍在70%以上。该酶在酸性环境下有较好的活性和稳定性。
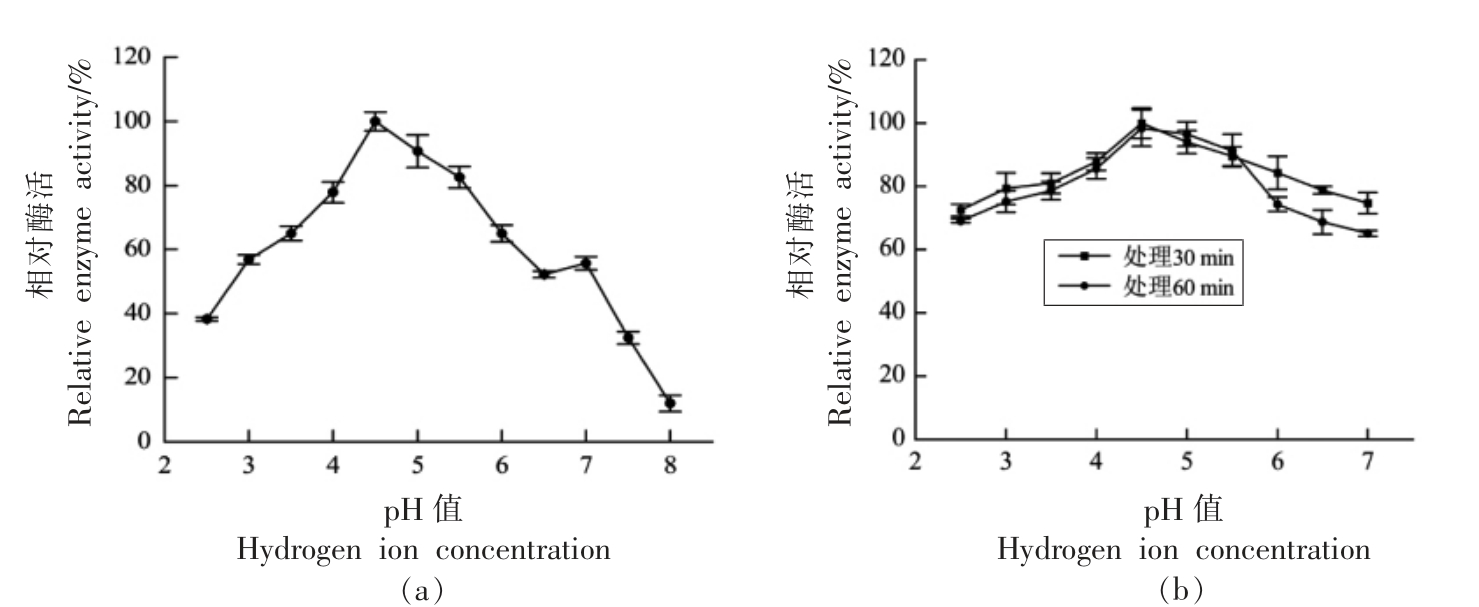
图3 壳聚糖酶最适pH 值(a)和pH 值稳定性(b)
Fig.3 Optimal reaction pH of chitosanase (a),pH stability of chitosanase (b)
2.2.3 金属离子及表面活性剂对壳聚糖酶的影响 0.5 mmol/L 金属离子及表面活性剂对重组酶酶活性影响如图4所示,金属离子Mn2+、Ca2+、Mg+对酶活性具有一定程度的激活作用,其中Mn2+激活效果最明显,可将酶活性提高至130%,Ca2+和Mg2+次之,可分别提升约10%,15%的酶活性。Cu2+、Zn2+、Ni+、Fe3+这些离子对重组酶的酶活性有比较明显的抑制作用,Cu2+、Fe3+离子可削弱酶活至60%的水平,而Zn2+、Ni+离子可减少约50%的酶活。Ag+、Hg+会导致酶活性完全丧失。K+、Na+、EDTA 对重组酶的活性几乎没有影响,其中EDTA 的作用结果表明该蛋白的活性位点并无金属离子配体。
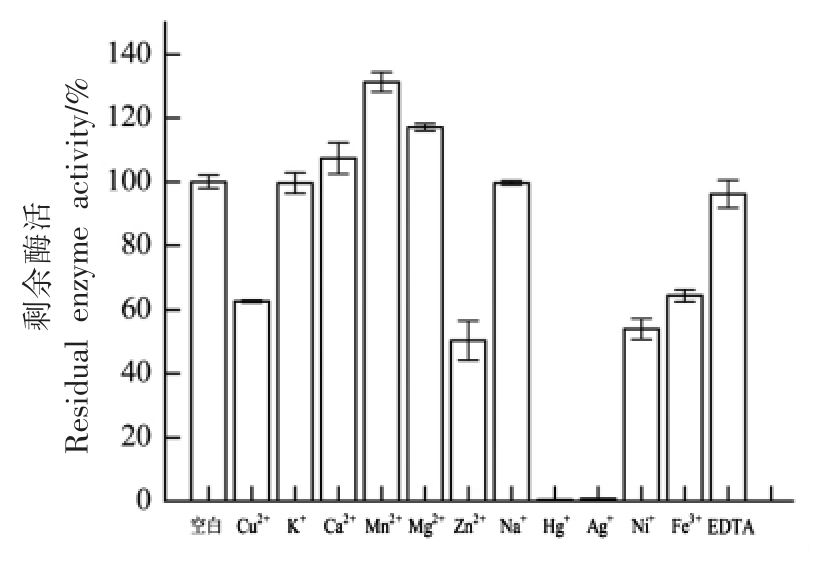
图4 金属离子及表面活性剂对壳聚糖酶的影响
Fig.4 The influence of metal ions and surfactants on chitosanase
3 讨论
大肠杆菌作为一种常见的原核表达系统具有操作简单、成本低、高生产率、表达速度快等优点[19]。本试验首次将中村芽胞杆菌该新物种体内的壳聚糖酶基因在大肠杆菌中实现异源表达并进行酶学性质分析。结果表明纯化后的中村芽胞杆菌壳聚糖酶酶比活力为561.19 U/mg,远高于Rivas 等[20]研究的芽孢杆菌属壳聚糖酶(56.9 U/mg)以及段衫[5]重组表达的沙雷氏菌壳聚糖酶(69.7 U/mg),但是大大低于Lin 等[12]在大肠杆菌中表达的枯草芽孢杆菌壳聚糖酶(3 655 U/mg)。因此,认为中村芽胞杆菌壳聚糖酶相较于其它一些菌种在壳聚糖水解方面具有一定的优势。
重组表达后的中村芽胞杆菌壳聚糖酶最适温度为37 ℃,42 ℃处理60 min 以上可以保持75%酶活,46 ℃下处理30 min 最低酶活仍然可以维持在50%,具有良好的温度稳定性。同其它壳聚糖酶相比最适温度低10~15 ℃,但是温度稳定性要高5 ℃[21-22],这与中村芽胞杆菌自身强环境耐受性特点相一致。在壳寡糖生产过程中通常会维持在较高的反应温度,目的是为了加快反应速率,减少微生物的污染[23],因此温度稳定性高的壳聚糖酶在工业生产中有缩小成本、降低风险等优势。有研究表明壳聚糖酶通过真核表达后最适温度会提高10 ℃,温度稳定性提高5 ℃[24-25],这可能是真核表达系统对酶进行糖基化修饰的结果[26]。后续可将中村芽胞杆菌壳聚糖酶在真核表达系统进一步优化修饰表达增强其热稳定性。
中村芽胞杆菌壳聚糖酶的最适pH 值为4.5,在pH 4.0~5.5 处理60 min 后最低酶活仍可维持在70%以上这一较高水平,与枯草芽孢杆菌壳聚糖酶(最适pH 5.5)和芽孢杆菌属TS(最适pH 5.0)相比在酸性环境下具有更强的耐受力[21-22]。壳聚糖降解过程随着时间增加其溶液黏度会不断上升以及流动性降低,最终会导致酶促反应被逐渐抑制,工业上常用的解决方法是加入盐酸或醋酸等酸溶液来改变壳聚糖的构象从而降低黏度带来的负面作用[14],该步骤会导致溶液pH 值降低影响壳聚糖酶的活性,而中村芽胞杆菌壳聚糖酶较强的酸耐受力有更好的应用前景。
不同金属离子和表面活性剂对中村芽胞杆菌壳聚糖酶的酶活力影响存在差异。K+和Na+对中村芽胞杆菌壳聚糖酶酶活性没有影响的原因可能是因为这些离子在细胞内外分布最广最常见,是细胞正常生存的必需离子,同时也是胞内进行渗透压调节的最主要离子。EDTA 作为一种金属离子络合剂,并未影响中村芽胞杆菌壳聚糖酶酶活性,推测这是因为该酶活性中心不存在金属离子所导致的[15]。Ag+、Hg+完全抑制中村芽胞杆菌壳聚糖酶的酶活性,这可能与Ag+、Hg+是重金属离子相关,重金属通常情况下会造成蛋白质变性,例如Hg+可与半胱氨酸残基侧链的巯基这一活性基团发生作用改变其空间折叠结构,使得底物配对几率降低从而抑制酶的活性[27]。5 mmol/L 的Cu2+、Zn2+、Ni+、Fe3+对酶活性有40%~50%的抑制作用。在其它研究中,枯草芽孢杆菌壳聚糖酶的活性会被约6 mmol/L 的Cu2+、Fe3+完全抑制[20]。解淀粉芽孢杆菌壳聚糖酶酶活力会被0.1 mmol/L Cu2+和Fe3+减少40%,2 mmol/L Zn2+减少55%,1 mmol/L Ni+减少13%[4-5],这与本文的研究结果相一致。此外,Fe3+是一种具有较强电负性的金属阳离子,可能会改变蛋白质分子的电荷分布,使得酶与底物的结合能力被降低[4]。Mn2+、Ca2+、Mg2+离子对中村芽胞杆菌壳聚糖酶有较明显的激活作用,有研究表明枯草芽孢杆菌壳聚糖酶氨基酸序列的第186~198 位对应的天冬氨酸和天冬酰胺侧链上的氧原子是Ca2+的激活结合位点,可以引起壳聚糖酶的空间结构发生变化,促进酶活性[28]。Mg2+可以作用在蓝藻Ch1H蛋白H 亚基内第468 位缬氨酸,624 位亮氨酸,660 位谷氨酸等活性氨基酸构成的活性结合位点,促进底物激活作用,并且这些氨基酸序列在各种生物中是高度保守的[29]。在Tottey 等[30]的研究中,3 个组氨酸和1 个谷氨酸所形成的结构域会导致Cu2+和Zn2+与蛋白质结合的紧密程度往往比Mn2+更加稳定,这与作者所研究的对酶活性效果影响最高的3 种离子Zn2+(降低50%),Cu2+(降低40%),Mn2+(促进30%)一致。因此推测在中村芽胞杆菌壳聚糖酶中也存在相似的结构位点,有必要进一步分析。随着壳寡糖应用面不断扩大,探索新型稳定高效的壳聚糖酶来满足日益增长的工业需求非常必要,本研究结果拓展了壳聚糖酶资源,为中村芽胞杆菌壳聚糖酶后续基础研究和实际应用提供理论依据。
[1]胡荣康,吴林秀,陈明军,等.不同分子量壳聚糖的制备及其应用研究进展[J].食品工业科技,2017,38(15): 324-328,334.
HU R K,WU L X,CHEN M J,et al.Research progress in preparation and application of chitosan with different molecular weight[J]Science and Technology of Food Industry,2017,38(15): 324-328,334.
[2]鲁晶娣,韦盘秋,易弋,等.壳聚糖酶的研究进展[J].中国调味品,2018,43(12): 168-173.
LU J D,WEI P Q,YI Y,et al.Research progress of chitosanase[J].China Condiment,2018,43(12):168-173.
[3]赵东科.壳聚糖衍生物的制备及生物活性研究[D].杭州: 浙江大学,2012: 6-10.
ZHAO D K.Preparation and biological activities of chitosan derivatives[D].Hangzhou: Zhejiang University,2012: 6-10.
[4]段静.解淀粉芽孢杆菌HZ-1510 壳聚糖酶基因的克隆、重组表达及其活性研究[D].武汉: 华中农业大学,2017: 2-3,47-49.
DUAN J.Gene cloning,expression and activity analysis of chitosanase from Bacillus amyloliquefaciens HZ-1510[D].Wuhan: Huazhong Agricultural University,2017: 2-3,47-49.
[5]段杉.无花果沙雷氏菌壳聚糖酶的生化性质及产酶条件的研究[D].广州: 华南理工大学,2003: 8-10,83.
DUAN S.Characterization and fermentation of chitosanase from Serratia ficaria CH0203[D].Guangzhou:South China University of Technology,2003: 8-10,83.
[6]GAO X A.JU W T,JUNG W J,et al.Purification and characterization of chitosanase from Bacillus cereus D-11[J].Carbohydrate Polymers,2007,72(3): 513-520.
[7]SILVA L C A,HONORATO T L,FRANCO T T,et al.Optimization of chitosanase production by Trichoderma koningii sp.under solid-state fermentation[J].Food and Bioprocess Technology,2012,5(5):1564-1572.
[8]熊妍妍,薛宇航,陈小娥,等.壳聚糖酶高产菌株的冷源等离子体诱变及发酵条件的优化[J].食品与发酵工业,2018,44(3): 101-106,113.
XIONG Y Y,XUE Y H,CHEN X E,et al.Cold source plasma mutation and optimization of fermentation conditions for chitosanase high-yield strain[J].Food and Fermentation Industries,2018,44 (3):101-106,113.
[9]KIM S K,RAJAPAKSE N.Enzymatic production and biological activities of chitosan oligosaccharides(COS): A review[J].Carbohydrate Polymers,2005,62(4): 357-368.
[10]LIAQAT F,ELTEM R.Chitooligosaccharides and their biological activities: A comprehensive review[J].Carbohydrate Polymers,2018,184: 243-259.
[11]QIAN M Y,LYU Q Q,LIU Y J,et al.Chitosan oligosaccharide ameliorates nonalcoholic fatty liver disease (NAFLD) in diet-induced obese mice[J].Marine Drugs,2019,17(7): 391-391.
[12]LIN J S,CHEN C C H,HU H Y,et al.Homologous and heterologous expression of chitosanase gene from Bacillus subtilis NCHU-05[J].Journal of Biotechnology,2010,150(supp-S): 354-354.
[13]KILANI-FEKI O,FRIKHA F,ZOUARI I,et al.Heterologous expression and secretion of an antifungal Bacillus subtilis chitosanase (CSNV26) in Escherichia coli[J].Bioprocess and Biosystems Engineering,2013,36(7): 1-8.
[14]牛培阳.高浓度壳聚糖溶解行为研究及其酶水解过程的改进[D].广州: 华南理工大学,2017: 2-3.
NIU P Y.The research on dissolution behavior and promotion of hydrolysis process of chitosan solution with high concentration[D].Guangzhou: South China University of Technology,2017: 2-3.
[15]石征宇,鲁晶娣,韦盘秋,等.Geobacillus thermodenitrificans ɑ-半乳糖苷酶原核表达及其酶学性质[J].食品工业科技,2019,40(10): 179-183.
SHI Z Y,LU J D,WEI P Q,et al.Prokaryotic expression and enzymatic properties of ɑ-galactosidase from Geobacillus thermodenitrificans[J].Science and Technology of Food Industry,2019,40(10):179-183.
[16]DUNLAP C A,SAUNDERS L P,SCHISLER D A,et al.Bacillus nakamurai sp.nov,a black-pigmentproducing strain[J].International Journal of Systematic and Evolutionary Microbiology,2016,66 (8):2987-2991.
[17]涂绍勇,杨爱华,梅双喜,等.3,5-二硝基水杨酸法 (DNS) 测定壳聚糖酶活力的探讨[J].食品科技,2012,37(1): 240-242,245.
TU S Y,YANG A H,MEI S X,et al.Determination of chitosanase activity by 3,5-dinitrosalicylic acid[J].Food Science and Technology,2012,37(1): 240-242,245.
[18]LOWRY O H,ROSEBROUGH N J,FARR A L,et al.Protein measurement with the folin phenol reagent[J].Journal of Biological Chemistry,1951,193(1): 265-275.
[19]LIU H W,BAO X M.Overexpression of the chitosanase gene in fusarium solani via agrobacterium tumefaciens-mediated transformation[J].Current Microbiology,2009,58(3): 279-282.
[20]RIVAS L A,PARRO V C,MORENO-PAZ M,et al.The Bacillus subtilis 168 csn gene encodes a chitosanase with similar properties to a streptomyces enzyme[J].Microbiology,2000,146(11): 2929-2936.
[21]PECHSRICHUANG P,YOOHAT K,YAMABHAI M.Production of recombinant Bacillus subtilis chitosanase,suitable for biosynthesis of chitosan -oligosaccharides[J].Bioresource Technology,2013,127: 407-414.
[22]ZHOU Z P,ZHAO S Z,WANG S Q,et al.Extracellular overexpression of chitosanase from Bacillus sp.TS in Escherichia coli[J].Applied Biochemistry and Biotechnology,2015,175(7): 3271-3286.
[23]JOHNSEN M G,HANSEN O C,STOUGAARD P.Isolation,characterization and heterologous expression of a novel chitosanase from Janthinobacterium sp.strain 4239[J].Microbial Cell Factories,2010,9(1): 1-9.
[24]马帅,杨绍青,刘翊昊,等.枯草芽孢杆菌壳聚糖酶在毕赤酵母中的高效表达及其酶解特性[J].食品科学,2019,40(14): 99-106.
MA S,YANG S Q,LIU Y H,et al.High-level expression of Bacillus subtilis chitosanase in Pichia pastoris and its hydrolytic properties[J].Food Science,2019,40(14): 99-106.
[25]张舒平,周鹏,苏春元,等.重组枯草芽孢杆菌壳聚糖酶的纯化和性质研究[J].中国农业大学学报,2012,17(1): 125-131.
ZHANG S P,ZHOU P,SU C Y,et al.Purification and characterization of a recombinant chitosanase from Bacillus subtilis[J].Journal of China Agricultural University,2012,17(1): 125-131.
[26]SEVO M,DEGRASSI G,SKOKO N,et al.Production of glycosylated thermostable Providencia rettgeri penicillin G amidase in Pichia pastoris[J].FEMS Yeast Research,2002,1(4): 271-277.
[27]FRIDJONSSON O,WATZLAWICK H,GEHWEILER A,et al.Cloning of the gene encoding a novel thermostable ɑ -GalActosidase from Thermusbrockianus ITI360[J].AppLied and Environmental Microbiology,1999,65(9): 3955-3963.
[28]COLOMER -PALLAS A,PEREIRA Y,PETIT -GLATRON M F,et al.Calcium triggers the refolding of Bacillus subtilis chitosanase[J].Biochemical Journal,2003,369(Pt3): 731-738.
[29]张丹.大豆镁离子螯合酶分子机理的研究[D].西安:西北大学,2020: 9-11.
ZHANG D.Molecular mechanism of Mg-chelatase in Soybean (Glycine max (L.) Merr.)[D].Xi'an: Northwest University,2020: 9-11.
[30]TOTTEY S,WALDRON K J,FIRBANK S J,et al.Protein-folding location can regulate manganesebinding versus copper or zinc-binding[J].Nature,2008,455(7216): 1138-1142.