粮食安全始终是全社会共同关注的重大问题之一,粮食安全的一大影响因素是微生物大量繁殖而导致的粮食腐败变质,如霉变[1]。粮食自身含有丰富的营养物质,即使在收获后也是一个活的生命体,会不断地进行新陈代谢,加之粮食籽粒表面携带大量的微生物,一旦储藏不当极易引起微生物的大量快速繁殖,从而造成粮食发热、霉变,甚至代谢产生大量生物毒素,不但使粮食的食用品质下降,还直接威胁人们的身体健康[2-3]。据联合国粮农组织统计,全球每年有2%的粮食因霉变而不能食用。我国粮食霉变的情况也十分严重,尤其是在一些高温阴雨时节,粮食存放不当极易引起吸湿、霉变。从数量上来看粮食表面的微生物细菌最多,霉菌次之;从危害程度上来看,虽然霉菌数量相对较少,但是其对生存环境的要求不高,在相对恶劣的环境下也能快速生长繁殖,是引起粮食腐败变质的主要微生物类群,同时代谢生成的真菌毒素也严重威胁人们的身体健康,对粮食储藏安全的影响较大[4-5]。
玉米是我国的主要粮食作物之一,产量约占全国粮食总产量的1/4,是主要的商品粮和战略储备粮种之一,也是我国重要的饲料原料和食品工业原料。玉米籽粒的胚部较大,营养丰富,吸湿性强,表面带菌量大,加上玉米收获时水分较高等多种因素,使其成为最易受到微生物侵害的粮种,导致其储藏过程中极易出现发热、霉变、真菌毒素超标[6-7]。玉米储藏过程中常见的霉菌有青霉属、曲霉属、根霉属、镰刀霉属等[8],它们可代谢产生如黄曲霉毒素、脱氧雪腐镰刀菌烯醇、玉米赤霉烯酮等真菌毒素,直接威胁人们的生命健康[9]。
在保障玉米安全储藏过程中,较多采用的是烘干、充氮气调、低温储藏等手段来降低环境中的水分、氧气含量、环境温度等以抑制微生物的生长和霉变[10-11]。然而,这些手段不仅耗能高、费用高,还会出现局部储藏环境异常,导致玉米表面微生物的快速生长繁殖,出现发热、霉变现象,并不可避免地导致真菌毒素超标。这其中有一大因素是玉米表面的微生物种群和生长、繁殖以及代谢规律不明确,不能提前感知、预测微生物的发生、发展及代谢规律[12-13]。
明确玉米中微生物种群特征及其多样性,是阐明其储藏过程中微生物发生、发展规律的首要条件。本研究采用经典的微生物学手段对玉米表面可培养微生物进行分离、纯化,然后针对所得菌种进行测序,构建系统发育树(Phylogenetic trees),进行分子生物学鉴定。环境中可培养微生物种类只占微生物总量的10%以下[14]。高通量测序技术是目前微生物多样性研究中使用最广泛的测序技术,具有高通量、低成本、准确率高等优点,不仅能够快速分析复杂微生物群落多样性,还能检测到较低丰度及不可培养的微生物[15-17]。本文采用高通量测序技术分析玉米表面的细菌和真菌群落,揭示其储藏过程中细菌和真菌的演替规律。
1 材料与方法
1.1 材料与试剂
玉米:2021 年购于郑州市高新区古荥村。
营养琼脂培养基(LB)、马铃薯葡萄糖琼脂培养基(PDA),北京奥博星生物技术有限责任公司;琼脂粉、丙三醇、Ezup 柱式细菌、真菌基因组DNA抽提试剂盒、RnaseA 酶、6×Loding dye、琼脂糖BBI、50×TAE、无毒核酸染料、DNA 分子量标准Marker、引物、ddH2O,1×TE、Big 2×super pcr mix,生工生物工程(上海)股份有限公司。
1.2 仪器与设备
2720 thermal cycler PCR 仪,赛默飞世尔科技(中国)有限公司;DYCP-31DNDNA 电泳槽、DYY-5 稳压电泳仪、紫外分析仪,北京六一仪器厂;DW-86L630 低温保存箱,澳柯玛股份有限公司;凝胶成像系统,BIO-RAD 公司。
1.3 试验方法
1.3.1 玉米中微生物的分离与纯化 称取玉米样品25 g,在无菌环境中加入装有225 mL 无菌水的三角瓶中,摇床振荡30 min,得到菌悬液。采用稀释平板法[18],取1 mL 菌悬液于9 mL 无菌蒸馏水中,依次稀释成10-2,10-3,10-4,10-5 浓度梯度的菌悬液。取不同稀释梯度的菌悬液100 μL,加入标记好的LB 和PDA 培养基平板中,用无菌涂布棒涂布均匀后,将LB 和PDA 平板分别置于37 ℃和28 ℃恒温培养箱中培养。菌落形态观察包括:菌落大小、颜色、高度,菌丝长短,气生菌丝质地(绒毛状、棉絮状、粉粒状、毡状或毯状等),菌落表面纹饰(皱纹、辐射沟纹或同心纹),菌落背面颜色等特征。挑取单个菌落在相应的培养基多次纯化,然后将菌落置30%甘油中,于-80 ℃冰箱保藏,备用。
1.3.2 细菌菌株的分子生物学鉴定 菌株活化:取1 管冰箱中保藏的菌株,室温解冻,在平板上活化两代。
引物序列:27F(5'-AGAGTTTGATCCTGGCT CAG-3')、1492R(5'-CTACGGCTACCTTGTTACGA-3')。
PCR 扩增:PCR 扩增体系:25 μL 2×高保真PCR 预混反应液(Big 2×super pcr mix),浓度为10 μmol/L 的引物各1 μL,模板DNA 2 μL,加dd H2O 至总体积50 μL。扩增条件:94 ℃预变性5 min,94 ℃变性30 s,55 ℃退火45 s,72 ℃延 伸1 min,30 次循环,最后72 ℃延伸10 min。PCR 产物经1%的琼脂糖凝胶电泳检测,凝胶成像分析仪观察电泳结果,将亮度清晰且条带长度1 500 bp 的PCR 产物送华大基因科技有限公司测序。测序结果输入美国国家生物信息技术中心(National Center of Biotechnology Information,NCBI)数据库中进行BLAST 比对,比对和查找亲源关系较近菌株的基因序列并下载。使用MEGA7 软件构建系统发育树,确定菌株的分类地位。
1.3.3 真菌菌株的分子生物学鉴定 DNA 模板的制备:将纯化的菌株在PDA 培养基上培养48 h后,刮取约0.2 g 菌丝体,置于1.5 mL 灭菌离心管中,然后将菌体放入研钵(-80 ℃预冷)中,反复液氮研磨至细粉状,迅速平均分装至2 个1.5 mL 离心管中。用Ezup 柱式真菌基因组DNA 抽提试剂盒,按其操作步骤提取真菌基因组DNA。使用紫外分光光度计检测DNA 的浓度。检测合格后将DNA 样品保存在-20 ℃,备用。
ITS 序列扩增引物:ITS1:5'-TCCGTAGGTGA ACCTGCGG-3' 和ITS4:5'-TCCTCCGCTTATTGA TATGC-3'。
PCR 扩增:PCR 扩增体系:25 μL Big 2×super pcr mix,浓度为10 μmol/L 的引物各1 μL,模板DNA 2 μL,加dd H2O 至总体积50 μL。扩增条件:94 ℃预变性5 min,94 ℃变性30 s,55 ℃退火45 s,72 ℃延伸1 min,30 次循环,最后72 ℃延伸10 min。PCR 产物经1%的琼脂糖凝胶电脉检测,用凝胶成像分析仪观察电泳结果。将亮度清晰且条带长度600 bp 的PCR 产物送华大基因科技有限公司测序。
1.3.4 微生物的多样性分析 样品准备:称取玉米50 g,置于450 mL 无菌水中,密封后在恒温摇床中振荡30 min。用0.22 μm 滤膜抽滤菌悬液,获得的菌体吸附在滤膜上。将该滤膜在4 ℃下送武汉菲沙基因有限公司进行高通量测序。
数据分析:根据细菌和真菌的不同,采用PCR方法对其高度保守序列如16S/18S/ITS 进行扩增,用PacBio 测序平台测序。PacBio SMRT 测序得到的原始数据,通过比对自我纠错,得到高质量的环状一致性序列(CCS),然后经去嵌合体序列等步骤,得到有效数据(Clean Data)。将最终有效数据用QIIME[19]软件中的UCLUS 在97%的相似度水平下进行可操作分类单元(Operational taxonomic units,OTU)聚类,并基于细菌NCBI 和真菌UNITE分类学数据库对OTU 进行分类学注释,再对OTU进行物种组成分析、多样性指数等分析[11,20]。在OTU 水平上计算覆盖度指数(Coverage)、丰富度指数(Chao1 和Ace)和多样性指数(Shannon-wienner 和Simpson),并在门和属分类水平上统计群落组成。
2 结果与分析
2.1 优势菌株的分离纯化与保守序列的提取
经分离与纯化,共得到11 株不同的细菌优势菌株,分别编号为B-1、B-2、B-3、B-4、B-5、B-6、B-7、B-8、B-9、B-10、B-11,其菌落都具有细菌的典型菌落特征(图1)。分别提取每株细菌的DNA,进行PCR 扩增,获得1 500 bp 左右的清晰条带,电泳条带清晰,符合测序要求,其特征如图2 所示。将相应的PCR 产物序列送往华大基因科技有限公司测序。
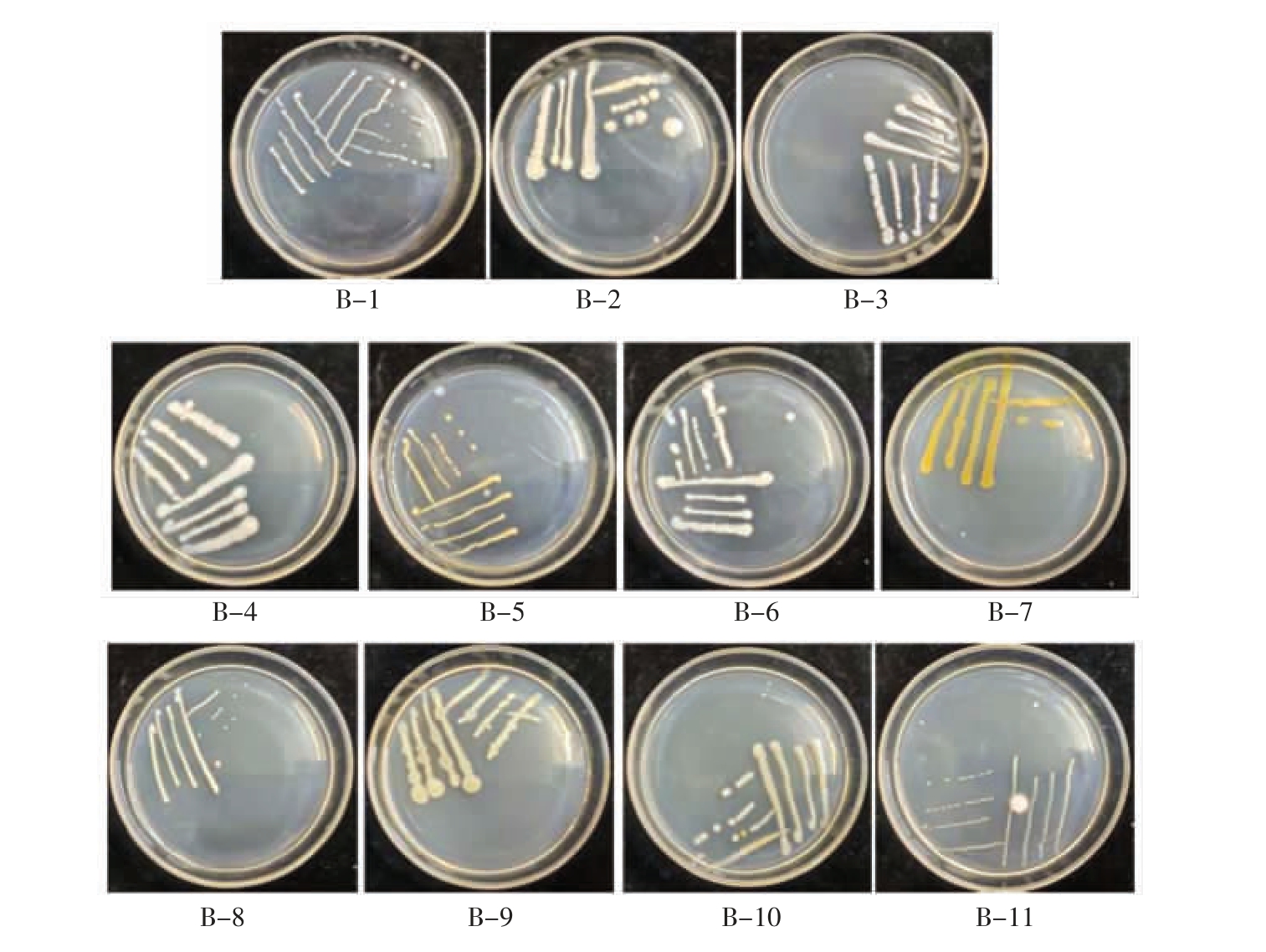
图1 分离纯化的优势细菌菌落特征
Fig.1 Colony characteristics of the isolated and purified dominant bacteria
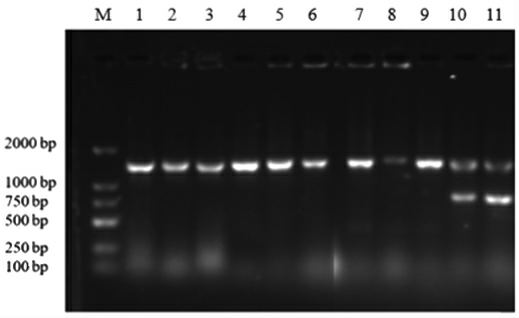
图2 细菌PCR 扩增产物电泳结果
Fig.2 Electrophoretic results of bacterial PCR amplification products
经分离与纯化,共得到12 株不同霉菌的优势菌株,分别编号为M-2、M-3、M-4、M-5、M-7、M-10、M-18、M-21、M-24、M-30、M-31、M-33,其菌落都具有真菌的典型群落特征(图3)。采用ITS 通用引物进行PCR 扩增,获得600 bp 左右的清晰条带,符合测序要求,其特征如图4 所示。将相应的PCR 产物序列送往华大基因科技有限公司测序。

图3 分离纯化的优势霉菌菌落特征
Fig.3 Colony characteristics of the isolated and purified dominant mould
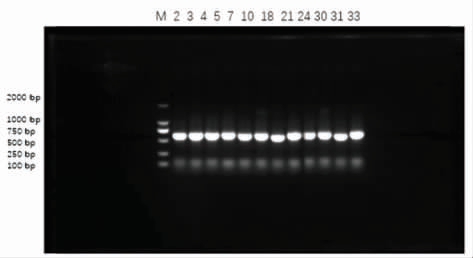
图4 真菌PCR 扩增产物电泳结果
Fig.4 Electrophoretic results of fungal PCR amplification products
2.2 系统发育树的构建与比对
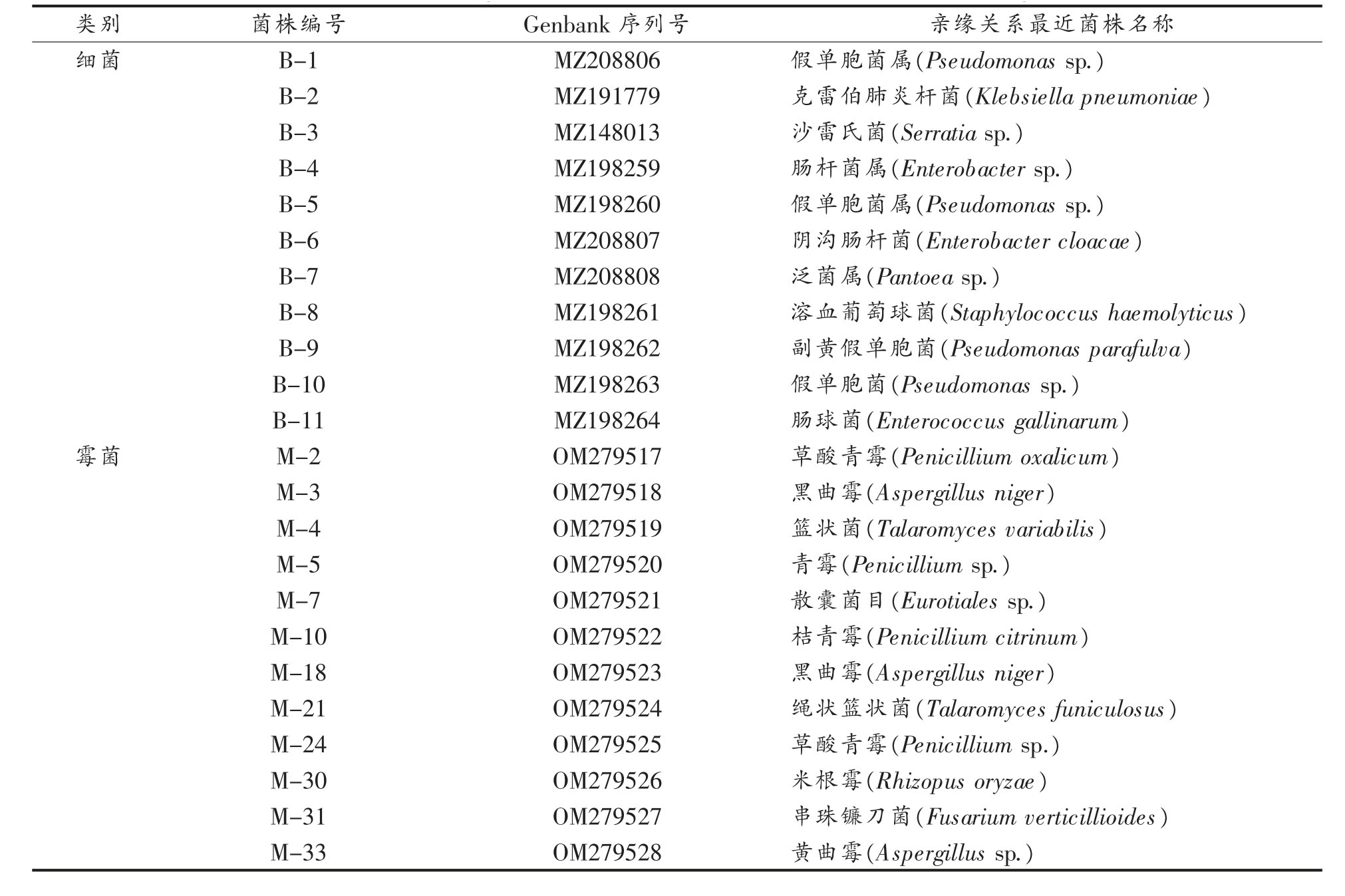
将华大基因科技有限公司反馈的每个菌株的基因序列上传至Genbank,获得相应的序列号(Accession number)。同时采用MEGA7 软件构建系统发育树,获得亲缘关系最近的菌株名称。该序列对应的名称即该菌株种属,所分离的优势细菌、霉菌菌株的Genbank 序列号和菌株名称见表1。
表1 玉米中部分细菌和霉菌的分子生物学鉴定结果
Table 1 Molecular biological identification results of some bacteria and fungi in maize
2.3 玉米中细菌群落多样性分析
2.3.1 细菌α-多样性分析
1)稀释曲线 采用随机抽取序列的方法,利用qiime 构建稀释曲线图(Rarefaction curve)。从样品中细菌的稀释曲线(图5)可以看出,随着测序数量的增加,它们所能代表构建的OTUs 数量也增加,曲线的斜率先上升后变得平坦,说明该样本的测序量充足,测序结果可靠,能够反映样品中微生物群落的种类和结构。
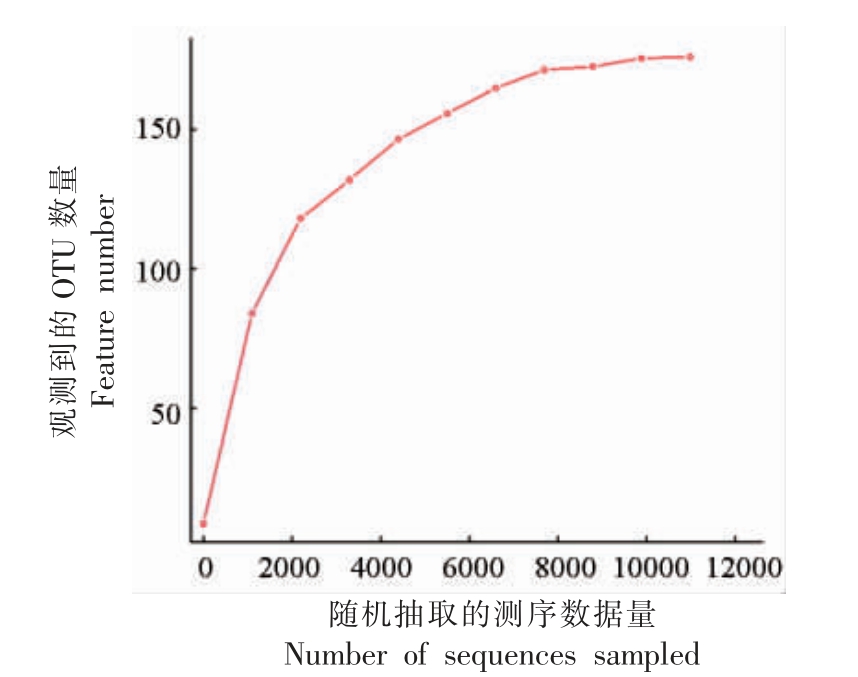
图5 细菌样品的序列稀释曲线图
Fig.5 Dilution curve of bacterial sample
2)秩-丰度曲线 秩-丰度曲线可直观反映样品中包含的物种分类丰富度和均匀度,该样品的秩-丰度曲线(Rank abundance)较宽且平坦,表明储藏玉米中的细菌群落具有良好的丰富度和均匀度(图6)。
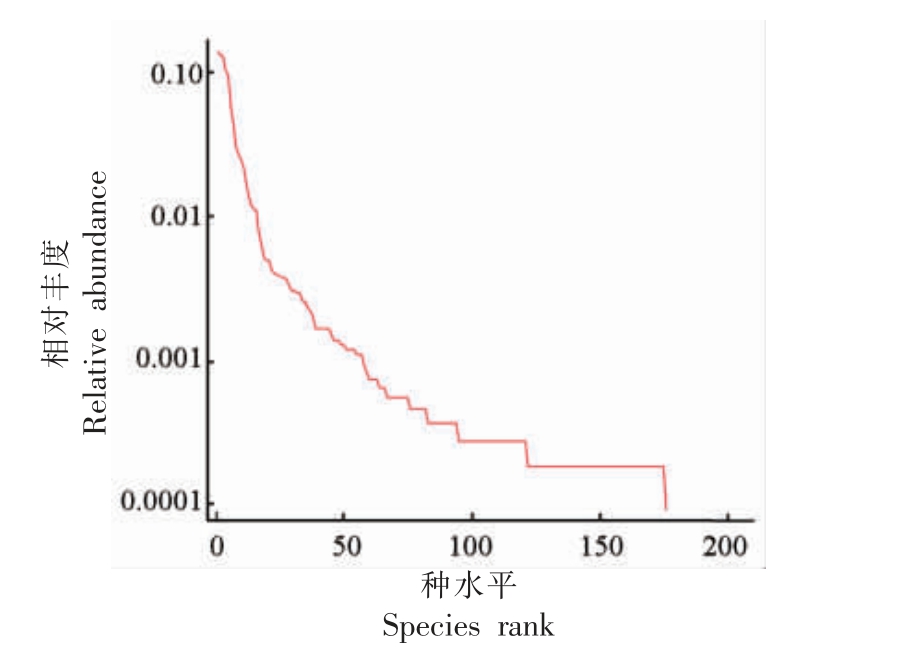
图6 细菌样品的秩-丰度曲线
Fig.6 Rank-abundance curve of bacteria sample
3)α-多样性指数 细菌群落多样性指数统计见表2。α-多样性指数反映样品内部物种的丰富度和多样性。根据97%相似性水平下的OTU 信息,采用α-多样性指标的覆盖率、Chao1、ACE,Shannon 指 数、Simpson、PD-whole-tree 指数对 样品微生物物种的丰富度和多样性进行评估。
表2 细菌群落的多样性指数
Table 2 Diversity index of bacterial community

每个样 品的覆盖率、Chao1、ACE,指数和Simpson、Shannon 指数分别用于评估物种的测序深度、丰度和多样性。覆盖率反映样本的真实情况,该指数越高,样本中序列未被测出的概率越低。Chao1、ACE 指数是群落丰度指数,Chao1、ACE指数越大,样品微生物群落丰度越高。Shannon、Simpson 指数是群落分布多样性指数,Simpson 指数越大,群落多样性越低,而Shannon 指数越大,样品微生物多样性越高。由表2 可知样品中的序列基本被测出,即样本测序结果可以反映样品的真实情况,并具有较高的丰富度和均匀性。
2.3.2 属分类水平上的物种组成分析 采用软件SMRT Link v8.0 对PacBio SMRT 测序得到的原始数据进行预处理和过滤,经测序分析得到14 734 条有效序列数据,序列长度在1 300~1 600 bp 之间,平均长度1 452 bp。用qiime 软件对有效序列进行OTU 聚类,结果显示14 734 条有效序列数据分归于176 个OTU 单元。
根据OTU 物种注释结果,可以了解每个样品的物种组成及其相对丰度。将检测合格的序列在97%的相似度下被划分至176 个OTU 中,从每个OTU 中挑选1 条代表性序列进行比对,然后统计其界、门、纲、目、科和属的种类和数量。在属分类水平上丰度排名前20 的群落中(图7),丰度最高的细菌属是泛菌属(Pantoea),所占比例为14.8%,肠球菌属(Enterococcus)、鞘氨醇杆菌属(Sphingobacterium)、金黄杆菌属(Chryseobacterium)所占比例均占10%以上,这与Liu 等[21]和陈泽斌等[22]研究结果基本一致。肠球菌属(Enterococcus)、鞘氨醇杆菌属(Sphingobacterium)差异较小,不足0.5%。其它组分如黄杆菌属(Flavobacterium)、寡养单胞菌属(Stenotrophomonas)、无色杆菌属(Achromobacter)、丛毛单胞菌属(Comamonas)、新鞘氨醇杆菌属(Novosphingobium)、短波单胞菌属(Brevundimonas)、葡糖杆菌属(Gluconobacter)、鞘脂菌属(Sphingobium)、乳球菌属(Lactococcus)等含量较低,均不足1%。Others 表示图中这30 个物种之外的其它所有物种的相对丰度之和,占微生物总量的27.1%。较多的研究认为玉米表面细菌的变化不会影响其储藏稳定性,而作为玉米粒内部菌群的主要来源,其表面细菌的变化直接影响玉米内生细菌的多样性和群落结构,从而影响玉米籽粒的生理和病理特性。该研究中丰度较高的泛菌属、肠球菌属、鞘氨醇杆菌属、伯克霍尔德菌属、假单胞菌属、无色杆菌属、短波单胞菌属都是玉米内生菌的主要来源[23]。也有研究表明,在储藏过程中不控制玉米中细菌的生长繁殖,其生长过程对周围环境的影响,比如增加底物的水解,造成环境温度升高等,也会影响真菌的生长和群落结构[24]。同时,玉米中细菌的生长繁殖会直接影响玉米中微生物含量,从而影响玉米粉的质量与安全[25]。
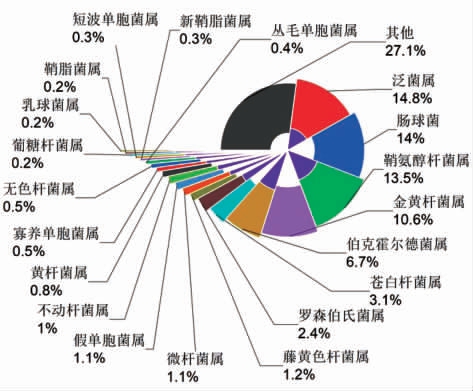
图7 玉米中细菌群落在属分类水平上的组成分布
Fig.7 The composition distribution of bacteria in maize on genus classification level
2.4 玉米中真菌群落多样性分析
2.4.1 生物信息学分析 采用Illumina MiSeq 平台对群落DNA 片段进行双端(Paired-end)测序,共得到120 580 条序列。通过qiime 软件,采用DADA2 方法对原始序列去噪。去除低质量序列后得到112 912 条序列,去噪后得到112 772 条序列,拼接后得到112 288 条序列,去除嵌合体后得到104 589 条序列(表3)。
表3 样本测序量统计表
Table 3 Statistical table of sample sequencing amount

对去除嵌合体后的高质量片段进行序列长度统计,结果发现高质量序列长度大都在200~250 bp 间,如图8 所示。
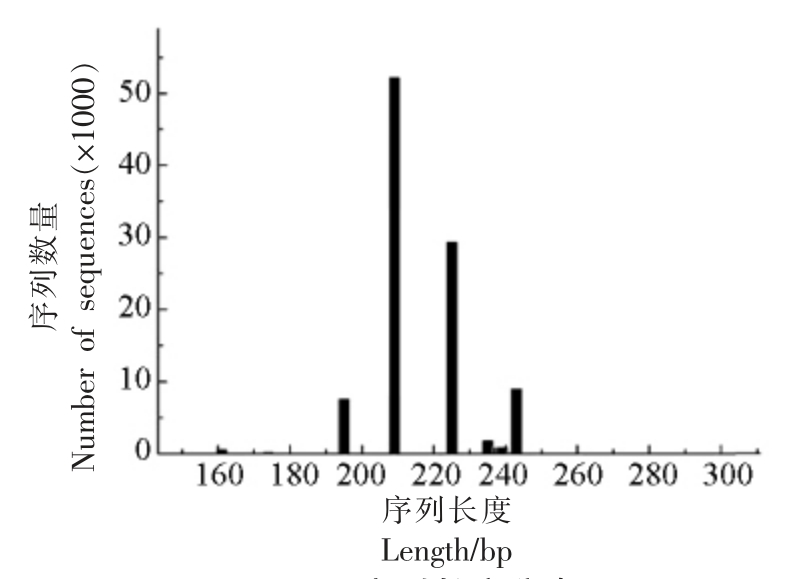
图8 序列长度分布图
Fig.8 Sequence length distribution
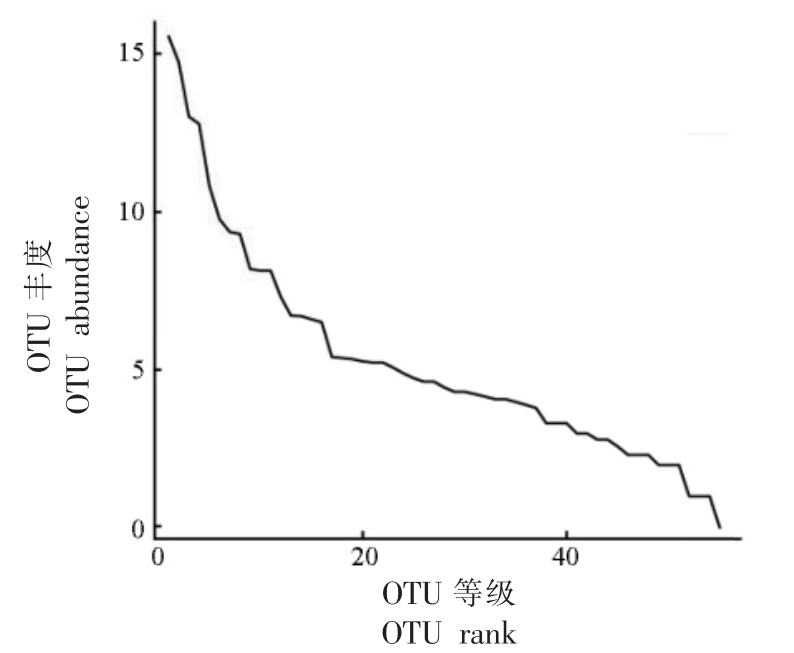
图9 真菌丰度等级曲线图
Fig.9 Fungal abundance grade curve
2.4.2 α-多样性分析
2.4.2.1 α-多样性指数 真菌群落多样性指数统计见表4。样本的α-多样性指数中Chao1 和Observed specie 与群落的丰富度有关,OTU 数量亦可以代表样品物种的丰度。Shannon 指数和Simpson 指数与群落的多样性有关。Pielou's evenness 指数和Good's coverage 指数(测序深度指数)分别表征群落的均匀度和检出的物种所占比例。由表4 可知,Good's coverage 指数大于0.9,说明样品文库中序列基本被测出。由其它指数可以看出所测样本中真菌菌群有较好的丰富度、多样性和均匀性。
表4 真菌群落的多样性指数
Table 4 Diversity index of fungal community

2.4.2.2 丰度等级曲线 样品的群落丰度等级曲线(Rank abundance curve)的折线总体较为平缓(图10),说明样本中各真菌群落OTU 间的丰度差异小,群落组成的均匀度较高,丰度分布较为均匀。
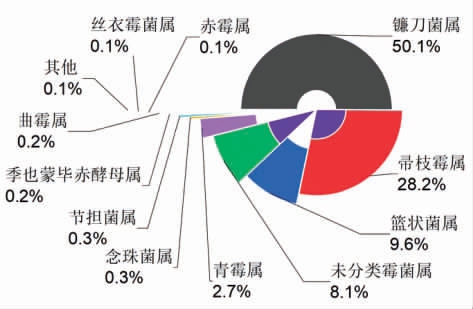
图10 玉米中真菌群落在属分类水平上的组成分布
Fig.10 The composition distribution of fungi in maize on genus classification level
2.4.3 属分类水平上的物种组成分析 针对真菌的ITS 序列,采用UNITE 数据库进行注释,各分类水平的统计结果见表5。经分析比对OTUs,共鉴定到2 门、9 纲、10 目、14 科、21 属、22 种。
表5 各水平微生物分类单元数统计表
Table 5 Statistical table of microbial taxa number at each level

统计在属(Genus)分类水平上丰度大于0.1%的群落,得到储藏玉米中真菌物种组成饼图(图9)。真菌中丰度最高的菌群为镰刀菌属(Fusarium),所占比例为50.1%,其次为帚枝霉属(Sarocladium),所占比例为28.2%,篮状菌属(Talaromyces)占9.6%,青霉属(Penicillium)占2.7%,曲霉属0.2%等。这与文献[26]和[27]研究结果一致。其它组分如念珠菌属(Candida)、节担菌属(Wallemia)、季也蒙毕赤酵母(Meyerozyma)、赤霉菌属(Gibberella)等含量较低,所占比例均不足1%,未分类霉菌属(unclassified_Fungi)占总量的8%。在玉米中的优势菌属中,帚枝霉属(Sarocladium)主要有玉米类帚枝霉(S.zeae)、纠缠帚枝霉(S.implicatum)和未分类种共3 个种,篮状菌属(Talaromyces)主要有沃特曼篮状菌(T.wortmannii)、产紫篮状菌(T.purpureogenus)、白双轮篮状菌(T.albobiverticillius)和2 个未分类种,共5 个种。青霉属(Penicillium)主要有P.bissettii、草酸青霉(P.oxalicum)和类阿达青霉(P.adametzioides)3 个种,曲霉属(Aspergillus)主要有黄曲霉(A.flavus)、黑曲霉(A.niger)、赤曲霉(A.ruber)3 个种。霉菌在生长过程中,一方面代谢产生一些淀粉酶、蛋白酶、普鲁兰酶、木聚糖酶等酶类水解底物供自身利用,另一方面产生一些诸如酮类、肽类、毒素类等代谢产物来抑制其它微生物,尤其是细菌的生长[28]。玉米中富含的曲霉属、镰刀菌属、青霉属等霉菌属,是黄曲霉毒素、呕吐毒素、玉米赤霉烯酮、伏马毒素、展青霉素、T-2 毒素等真菌毒素污染的主要代谢产物。如果能够有效抑制玉米表面的优势霉菌的生长代谢,则有助于控制玉米中的真菌毒素污染问题[29]。
3 结论
对玉米表面的优势细菌和真菌进行分离纯化,共获得11 株细菌菌株和12 株霉菌菌株。通过其保守序列的PCR 测序及构建系统发育树,初步鉴定其菌株名称。通过高通量测序技术分析玉米中细菌和真菌的多样性。结果表明,玉米中主要优势细菌为泛菌属、肠球菌属、鞘氨醇杆、金黄杆菌属,主要的优势真菌为镰刀菌属、帚枝霉、篮状菌属、青霉属、曲霉属。高通量测序分析所得结果中高丰度细菌和真菌种类与分离纯化得到的菌株结果基本一致。表明镰刀菌、青霉、曲霉等是玉米中主要的污染真菌,这些菌株也是代谢产生真菌毒素的重要菌群,对储粮安全有重要影响[30-31]。
[1] LANE B,SHARMA S,NIU C X,et al.Changes in the fungal microbiome of maize during hermetic storage in the united states and kenya[J].Frontiers in Microbiology,2018,9:13.
[2] SUN X D,SU P,SHAN H.Mycotoxin contamination of maize in China[J].Comprehensive Reviews in Food Science and Food Safety,2017,16(5):835-849.
[3] ZHANG D D,HU S,WU Q,et al.Construction of ZnO@mSiO2 antibacterial nanocomposite for inhibition of microorganisms during Zea mays storage and improving the germination[J].LWT-Food Science and Technology,2022,168:113907.
[4] 刘新,任凤山,徐晓辉,等.山东省玉米青枯病主要致病菌的分离鉴定及优势致病菌的致病性测定[J].山东农业科学,2021,53(9):100-104.LIU X,REN F S,XU X H,et al.Isolation and identification of main pathogens of corn stalk rot and pathogenicity determination of dominant pathogens in Shandong province[J].Shandong Agricultural Sciences,2021,53(9):100-104.
[5] QUIJADA N M,HERNANDEZ M,RODRIGUEZLAZARO D.High-throughput sequencing and food microbiology[J].Advances in Food and Nutrition Research,2020,91:275-300.
[6] MANNAA M,KIM K D.Influence of temperature and water activity on deleterious fungi and mycotoxin production during grain storage[J].Mycobiology,2017,45(4):240-254.
[7] BOLANOS-CARRIEL C,WEGULO S N,HALLENADAMS H,et al.Effects of field-applied fungicides,grain moisture,and time on deoxynivalenol during postharvest storage of winter wheat grain[J].Canadian Journal of Plant Science,2020,100(3):304-313.
[8] QIU J B,LU Y N,HE D,et al.Fusarium fujikuroi species complex associated with rice,maize,and soybean from jiangsu province,China:Phylogenetic,pathogenic,and toxigenic analysis[J].Plant Disease,2020,104(8):2193-2201.
[9] WAWRZYNIAK J,WASKIEWICZ A,RYNIECKI A.Evaluation of critical points of mould growth and mycotoxin production in the stored barley ecosystem with a hazardous initial microbiological state of grain[J].Journal of Stored Products Research,2018,77:166-176.
[10] ELMHOLT S,KRISTENSEN E F,THRANE U.Comparing the effect of continuous drying and drum drying on fungal contamination of bread grain(rye)[J].Biosystems Engineering,2007,97(3):425-428.
[11] EL-KHOLY M M,KAMEL R M.Performance analysis and quality evaluation of wheat storage in horizontal silo bags[J].International Journal of Food Science,2021,2021:1248391.
[12] FELSOCIOVA S,KOWALCZEWSKI P L,KRAJCOVIC T,et al.Quantitative and qualitative composition of bacterial communities of malting barley grain and malt during long-term storage[J].Agronomy-Basel,2020,10(9):1301.
[13] SCHMIDT M,ZANNINI E,ARENDT E K.Recent advances in physical post -harvest treatments for shelf-life extension of cereal crops[J].Foods,2018,7(4):45.
[14] SOLANKI M K,ABDELFATTAH A,BRITZI M,et al.Shifts in the composition of the microbiota of stored wheat grains in response to fumigation [J].Frontiers in Microbiology,2019,10:1098.
[15] 岳晓禹,张华,陈威风,等.应用高通量测序技术检测小麦储藏过程中真菌群落的变化[J].食品科学,2020,41(14):109-115.YUE X Y,ZHANG H,CHEN W F,et al.Using high-throughput sequencing to study the development of fungal community in stored wheat[J].Food Science,2020,41(14):109-115.
[16] XING H Q,MA J C,XU B L,et al.Mycobiota of maize seeds revealed by rDNA -ITS sequence analysis of samples with varying storage times[J].Microbiologyopen,2018,7(6):e00609.
[17] NILSSON R H,ANSLAN S,BAHRAM M,et al.Mycobiome diversity:High -throughput sequencing and identification of fungi[J].Nature Reviews Microbiology,2019,17(2):95-109.
[18] ZHANG D D,LIU J M,SONG N,et al.Fabrication of mesoporous La3Ga5GeO14:Cr3+,Zn2+ persistent luminescence nanocarriers with super-long afterglow for bioimaging-guided in vivo drug delivery to the gut[J].Journal of Materials Chemistry B,2018,6(10):1479-1488.
[19] CAPORASO J G,KUCZYNSKI J,STOMBAUGH J,et al.QIIME allows analysis of high -throughput community sequencing data[J].Nature Methods,2010,7(5):335-336.
[20] EDGAR R C.UPARSE:highly accurate OTU sequences from microbial amplicon reads[J].Nature Methods,2013,10(10):996-998.
[21] LIU Y,YAN H,ZHANG X X,et al.Investigating the endophytic bacterial diversity and community structures in seeds of genetically related maize(Zea mays L.)genotypes[J].3 Biotech,2020,10(1):10.
[22] 陈泽斌,李冰,王定康,等.应用Illumina MiSeq高通量测序技术分析玉米内生细菌多样性[J].现代食品科技,2016,32(2):113-120.CHEN Z B,LI B,WANG D K,et al,Study on the diversity of endophytic bacteria in maize using Illumina MiSeq High-throughput sequencing system[J].Modern Food Science and Technology,2016,32(2):113-120.
[23] LIU Y,YAN H,ZHANG X,et al.Investigating the endophytic bacterial diversity and community structures in seeds of genetically related maize(Zea mays L.)genotypes[J].3 Biotech,2020,10(1):27.
[24] GUO J H,SUN L H,GUO H W,et al.Effect of Cellulase -producing bacteria on fungi community structure and ester generation in Chinese liquor fermenting grains[J].Journal of the American Society of Brewing Chemists,2018,76(2):130-140.
[25] LOPEZ A M M,SIMSEK S.Pathogens control on wheat and wheat flour:A review[J].Cereal Chemistry,2021,98(1):17-30.
[26] TWARUZEK M,SKRZYDLEWSKI P,KOSICKI R,et al.Mycotoxins survey in feed materials and feedingstuffs in years 2015-2020[J].Toxicon,2021,202:27-39.
[27] HABSCHIED K,KRSTANOVIC V,ZDUNIC Z,et al.Mycotoxins biocontrol methods for healthier crops and stored products[J].Journal of Fungi,2021,7(5):25.
[28] CADELIS M M,NIPPER N S L,GREY A,et al.Antimicrobial polyketide metabolites from Penicillium bissettii and P.glabrum[J].Molecules,2022,27(1):240.
[29] 张咚咚,胡思,HAYAT Z,等.氧化锌纳米颗粒对玉米储藏过程中微生物的抑制作用[J].食品科学,2021,42(21):137-144.ZHANG D D,HU S,HAYAT Z,et al.Inhibition of ZnO nanoparticles on microorganisms during maize storage[J].Food Science,2021,42(21):137-144.
[30] VANDICKE J,DE VISSCHERE K,AMEYE M,et al.Multi-mycotoxin contamination of maize silages in flanders,belgium:Monitoring mycotoxin levels from seed to feed[J].Toxins,2021,13(3):202.
[31] LIU Z,ZHANG G,ZHANG Y,et al.Factors controlling mycotoxin contamination in maize and food in the Hebei province,China[J].Agronomy for Sustainable Development,2016,36(2):39.