鼠李糖乳杆菌(Lactobacillus rhamnosus)是厚壁菌门乳杆菌科的一种革兰氏阳性杆状细菌。鼠李糖乳杆菌广泛存在于婴儿粪便样本以及人类母乳中[1],这些细菌通常对消化道中的条件具有耐受性(即低pH 值、胆盐、厌氧条件)[2]。国内外大量研究发现,鼠李糖乳杆菌具有调节肠道菌群[3],调节肠道免疫[4-7],抗氧化[8]、抑制生物毒素[9],抑制致病菌[10-11]和调控脂肪细胞分化[12]等功能。据报道,在新生儿中使用某些鼠李糖乳杆菌菌株,特别是鼠李糖乳杆菌GG,可促进早产和低出生体重新生儿的生长[13-14]。
前期研究[15]从40 份人初乳样品中分离出有活性的益生菌菌株——鼠李糖乳杆菌Probio-M9,该菌株在人工胃肠液中消化11 h 后的存活率为78.33%,在中国具有自主知识产权,推测更适合中国人和其他亚洲人,因此本文对该菌株做后续安全性评估、动物实验和全基因组测序等研究具有参考价值。
比较基因组学是从基因水平比较不同物种的基因组序列,从而了解其基因的功能、表达机制及物种进化的学科[16]。Sun 等[17]通过比较基因组学对213 株乳杆菌属进行分析,系统解析了乳杆菌属不同谱系的遗传进化历程,发现所有的乳杆菌都是由一个共同的祖先进化而来的。Yu 等[18]通过核心基因比对分析乳酸乳球菌、植物乳球菌等若干乳球菌属的核心基因,解析乳球菌属内的遗传进化关系,发现乳球菌属在基因组大小,基因含量和碳水化合物代谢方面具有高度多样性。孙靓[19]通过比较基因组学分析两株鼠李糖乳杆菌,结果发现鼠李糖乳杆菌基因组层面的断裂基因和转录组层面的丙酮酸代谢途径上表达的差异,可能是鼠李糖乳杆菌高产L-乳酸的原因。比较基因组学能有效分析、挖掘和利用微生物基因组各个层面信息,能够准确且快速地研究菌种的同源基因以及特有功能。
目前对来源于母乳的菌株研究较少。特别是在中国,关于从人乳中分离得到的鼠李糖乳杆菌的研究报道更少,而关于母乳分离株鼠李糖乳杆菌的比较基因组研究更是少之又少。本研究将以母乳分离株鼠李糖乳杆菌Probio-M9 结合NCBI Refseq 数据库下载的214 株鼠李糖乳杆菌基因组序列为研究对象,通过比较基因组学解析不同菌株基因特征及种内基因差异,了解其遗传进化机制,为鼠李糖乳杆菌Probio-M9 开发利用提供参考。
1 材料与方法
1.1 试验菌株
鼠李糖乳杆菌Probio-M9 由内蒙古农业大学乳酸菌菌种资源库(Lactic acid bacteria collection center,LABCC)提供。
1.2 鼠李糖乳杆菌基因组序列
截止2021年4月17日,已将NCBI(National Coalition Building Institute,https://www.ncbi.nlm.nih.gov/)Refseq 数据库中214 株鼠李糖乳杆菌基因序列以及一株暂未公开的鼠李糖乳杆菌Probio-M9 全部下载完成。
1.3 菌株培养与基因组DNA 的提取、测序
菌株鼠李糖乳杆菌Probio-M9 的培养具体操作步骤如下:将该菌接种到37 ℃的MRS 液体培养基中,无氧条件培养24 h 后进行传代,最后使用PBS 缓冲液将3 代菌泥中废弃培养基去除,然后进行全基因组DNA 的提取。全基因组DNA 提取方法参考钟智等[20]的方法。将提取的DNA 通过1%琼脂糖凝胶电泳进行完整度和纯度检测,将符合要求的高质量DNA 按照PacBio SMRT 全基因组DNA 建库流程建立10 kb 文库,建库后根据PacBio SMRT RS II 测序平台上机流程进行全基因组测序。
1.4 基因组测序和组装
将测序所得原始数据进行质量评估[21],去掉低质量测序序列和接头,使用RS_HGAP_Assembly.3 软件对鼠李糖乳杆菌Probio-M9 序列进行质控和基因组组装,并使用Circlator(V1.5.5)[22]软件对3 代数据进行环化,最后获得Probio-M9 的全基因序列。
1.5 比较基因组分析
1.5.1 泛-核心基因集构建 利用Prokka[23]软件对菌株基因组进行基因预测后,采用Roary[24]软件对核心基因集、泛基因集进行识别。
1.5.2 系统发育树构建 使用经Roary 软件分析得到的核心基因序列,通过TreeBest 软件(http://www.mybiosoftware.com/treebest)构建邻接系统发育树(Neighbor-Joining,NJ)。使用iTol 在线软件对系统发育树进行可视化(http:// https://itol.embl.de/)[25]。
1.5.3 平均核苷酸一致性(Average nucleotide identity,ANI)计算 本研究参考Jain 等[26]报道的fastANI(https://github.com/ParBLiSS/FastANI)计算215 株鼠李糖乳杆菌的菌株间ANI 值。利用TBtools[7]软件绘制ANI 聚类热图。
1.5.4 全基因组圈图和功能基因组分析 将215株鼠李糖乳杆菌核酸序列文件分别上传至RAST(Repaid Annotion using Subsystem Technology,http://rast.nmpdr.org/rast.cgi)进行注释,并下载对应的基因功能注释文件。将鼠李糖乳杆菌Probio-M9 的功能基因注释文件上传至CGView Server(http://stothard.afns.ualberta.ca/cgview_server/)进行圈图绘制。
1.5.5 碳水化合物活性酶 将组装完成的鼠李糖乳杆菌Probio-M9 全基因组序列上传至在线注释dbCAN 平台中(http://bcb.unl.edu/dbCAN2/)进行注释,下载其注释文件。统计鼠李糖乳杆菌Probio-M9 基因组信息。
2 结果和分析
2.1 鼠李糖乳杆菌基因组基本信息
对样品鼠李糖乳杆菌Probio-M9 测序数据进行评估组装后,拼接成完整的基因组圈图(图1),并统计鼠李糖乳杆菌所有菌株的基因组信息。鼠李糖乳杆菌Probio-M9 组装后,基因组大小为2 987 632 bp,GC 含量为46.76%,包含2 934 个蛋白质编码区(Coding sequence,CDS)、60 个tRNA、75 个RNA。215 株鼠李糖乳杆菌基因组大小(2.97±0.11)Mbp,GC 含量为(46.68±0.016)%,CDS为(2 671±130)个。
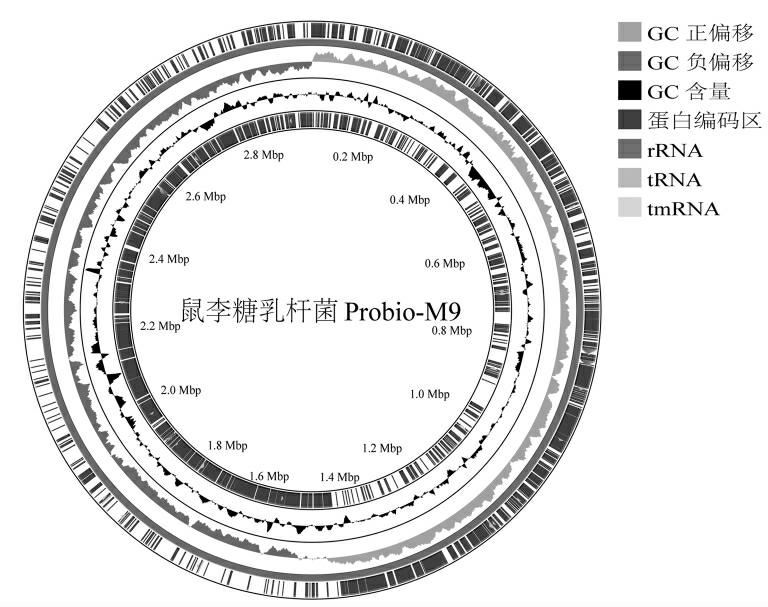
图1 鼠李糖乳杆菌Probio-M9 全基因组圈图
Fig.1 The whole genome circle-map of L.rhamnosus Probio-M9
2.2 泛-核心基因集构建
一个物种泛基因组主要由核心基因组、非必须基因组以及特有基因组3 部分组成[28]。215 株鼠李糖乳杆菌共识别到16 915 个泛基因,247 个核心基因,并且215 株鼠李糖乳杆菌核心基因占平均CDS 为9.2%,揭示鼠李糖乳杆菌存在一个较大的泛基因组,但是拥有一个较小的核心基因组。鼠李糖乳杆菌的泛-核心基因统计结果如图2所示,核心基因随着基因组的增加整体呈现一个先下降后趋于稳定的状态而泛基因组大小整体呈现上升趋势,表明鼠李糖乳杆菌的泛基因组尚且处于一个相对开放的状态,与Kant 等[29]的研究结果基本一致。
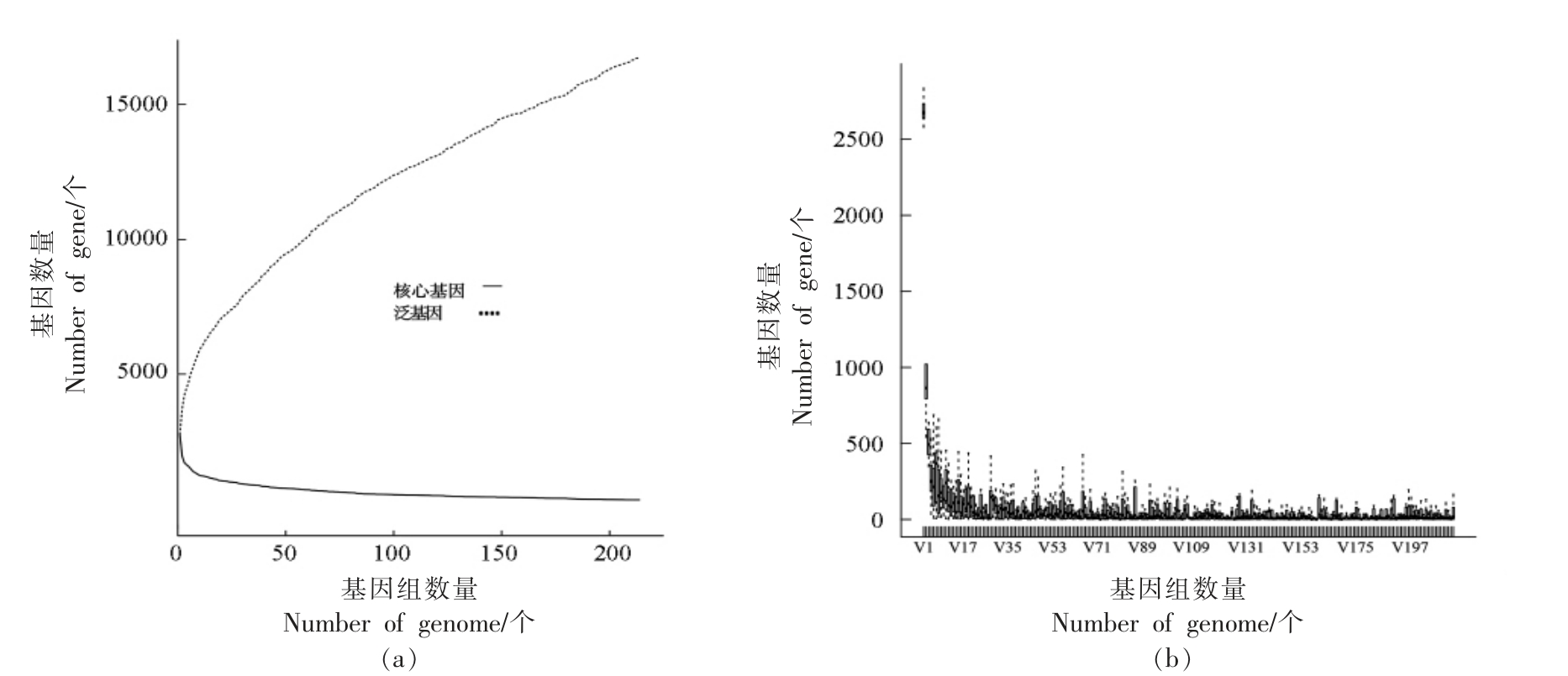
图2 泛-核心基因集变化(a)和新基因集变化(b)趋势图
Fig.2 The trend chart of the size of pan-core genes(a)and new genes(b)
2.3 核心基因构建系统发育树
系统发育树可直观反映菌株间群体结构和遗传进化关系。为了研究鼠李糖乳杆菌种内遗传进化关系,本研究基于215 株鼠李糖乳杆菌(包含模式菌株鼠李糖乳杆菌DSM20021T=鼠李糖乳杆菌NBRC3425T=鼠李糖乳杆菌JCM1136T、鼠李糖乳杆菌NRRL B-442T),采用邻接法,基于247 个核心基因通过1 000 次的引导迭代,构建系统发育树。
由图3 可知,215 株乳酸菌主要分为7 大分支,分别命名为分支Ⅰ、分支Ⅱ、分支Ⅲ、分支Ⅳ、分支Ⅴ、分支Ⅵ和分支Ⅶ。由图可知,分支Ⅱ是该树最大的分支且鼠李糖乳杆菌Probio-M9 和模式菌株均在该分支中,并且该分支各菌株之间的遗传关系近、差异很小、区分难度大。同时根据系统发育树外圈注释发现,鼠李糖乳杆菌的遗传关系与分离源和分离地相关性不明显。
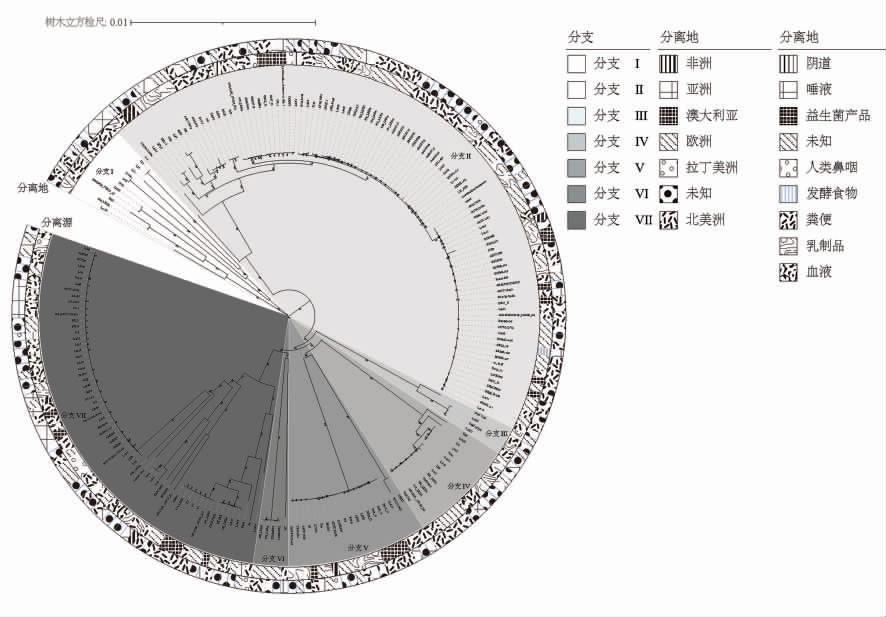
图3 基于核心基因的系统发育树
Fig.3 Phylogenetic tree based on core genes
2.4 ANI 分析
ANI 是通过比对基因组的同源序列以鉴定菌株亲缘关系[30]。在比较基因组学分析过程中,ANI可用于评估基因组间多态性的程度,也可基于基因组序列进行物种鉴别,一般认为ANI 值大于95%即为同一物种[31]。2018年Ciufo 等[32]将95%的阈值改为96%,并以此作为物种边界。
为解析鼠李糖乳杆菌遗传多样性,本研究对215 株鼠李糖乳杆菌进行两两之间ANI 计算,并构建聚类热图,结果如图4所示,全部菌株之间ANI 值均大于96.38%,鼠李糖乳杆菌菌株之间具有较高的总体序列同一性(>96%),这与Nissila等[33]和Arnold 等[34]的研究结果一致,提示鼠李糖乳杆菌种内的相似度较高。
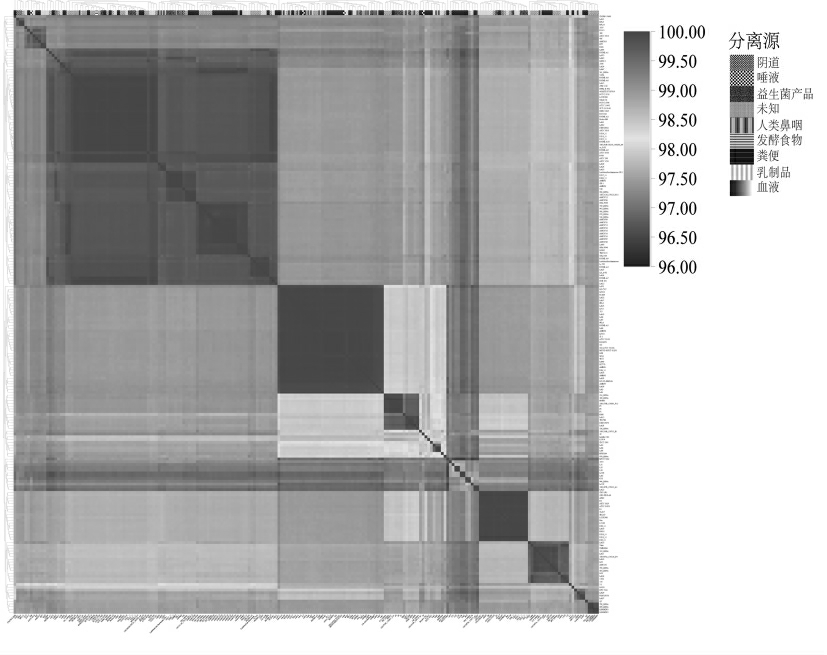
图4 平均核酸一致性
Fig.4 Average nucleotide identity
2.5 基因组预测与注释
由于分支Ⅱ是系统发育树最大分支且鼠李糖乳杆菌Probio-M9 和模式菌株均在该分支中,因此利用RAST 在线工具对分支Ⅱ中98 株鼠李糖乳杆菌菌株进行RAST 注释,结果如图5所示。在鼠李糖乳杆菌基因组中注释到碳水化合物代谢(Carbohydrates)、蛋 白 质 代 谢(Protein metabolism)、氨基酸及其衍生物(Amino acids and derivatives)、细胞壁和被膜(Cell wall and capsule)、DNA 代谢(DNA metabolism)、RNA 代谢(RNA metabolism)和应激反应(Stress response)等,共计26 个功能类别。其中占比最大的是“碳水化合物代谢”,其次是“蛋白质代谢”和“氨基酸及其衍生物”。根据RAST 注释发现菌株Lrh44、CRL1505、Lactobacillus rhamnosus、KF7、BIOML -A4、Lrh26、Lrh25、AMC010、1019 在硫代谢方面要显著高于鼠李糖乳杆菌Probio-M9,而其它菌株与鼠李糖乳杆菌Probio-M9 呈现一种高度相似,从RAST 注释无法找到差异。

图5 RAST 注释
Fig.5 RAST annotation
2.6 基因组圈图和功能基因组分析
本文以RAST 注释中与鼠李糖乳杆菌Probio-M9 差异明显的3 株菌和一株模式菌株鼠李糖乳杆菌DSM20021T 为研究对象,通过BRIG 软件进行分析,结果如图6所示,参考菌株鼠李糖乳杆菌Probio-M9 与模式菌株鼠李糖乳杆菌DSM20021T 匹配度最高为100%,最低为63.31%。约有344 bp 和293 bp 基因组片段匹配度大于等于90%,占总片段长度的22.21%;分析发现477 bp 和613 bp 基因片段匹配度小于70%,约占总片段的3.08%。鼠李糖乳杆菌Probio-M9 注释到12个特有基因(见表1),主要负责自身代谢、转录、运输等方面的调控。图中白色部分是核苷酸同一性小于50%的区域,主要包括编码应激蛋白、生物合成相关蛋白、假定蛋白和噬菌体相关蛋白等多种蛋白质的基因,而这些蛋白质小范围的插入与缺失可以使菌株基因组结构多样化[35-37]。
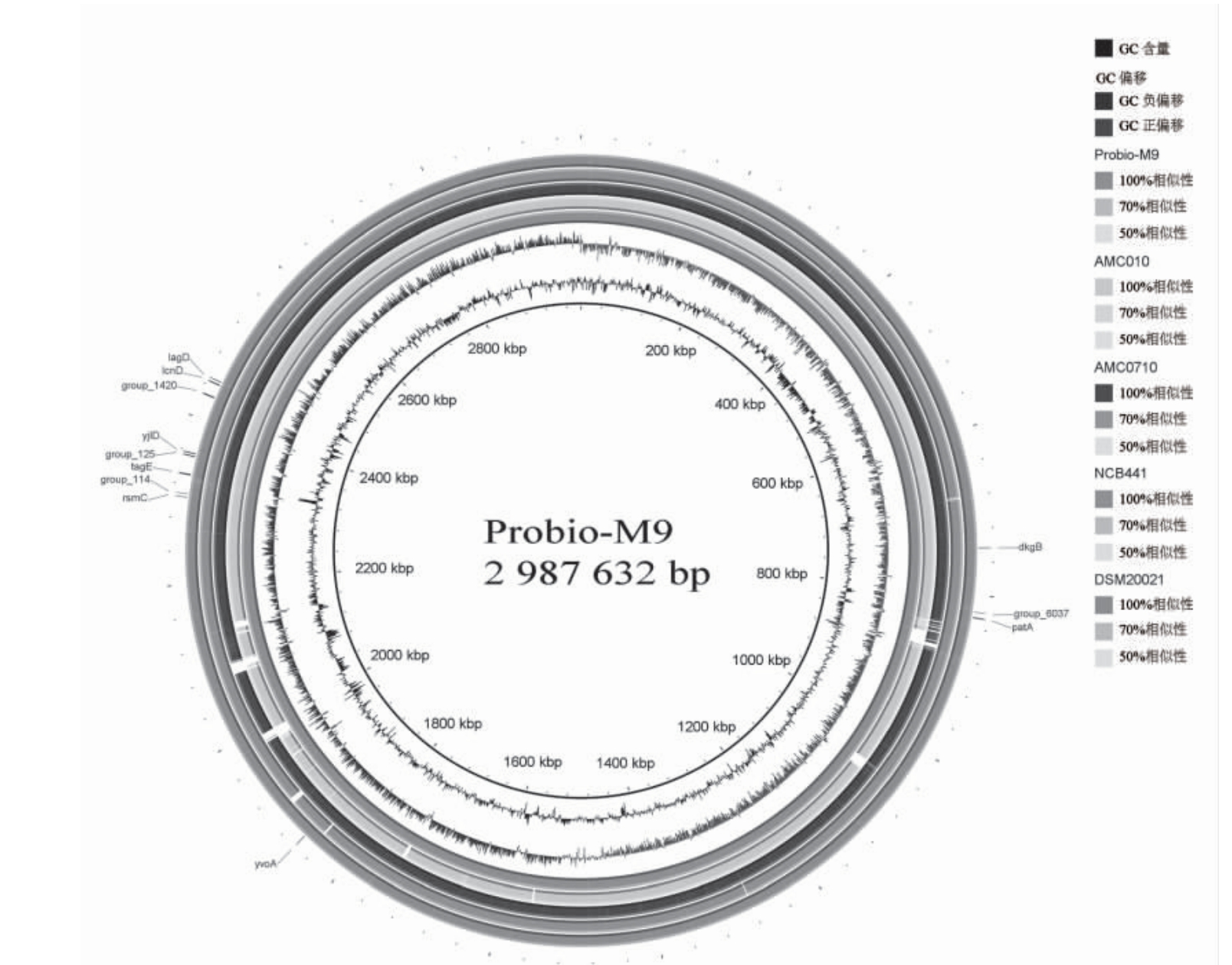
图6 以鼠李糖乳杆菌Probio-M9 为参考基因组的Brig 分析
Fig.6 Brig analysis based on L.rhamnosus Probio-M9 as reference genome
表1 鼠李糖乳杆菌Probio-M9 特有基因
Table 1 L.rhamnosus Probio-M9 specific gene

基因名称功能注释yvoA转录抑制因子rsmC核糖体RNA 小亚基甲基转移酶yjlDNADH 脱氢酶样蛋白dkgB2,5-二酮-d -葡萄糖酸还原酶patA假定n -乙酰- ll -二氨基丙二酸转氨酶tagE假定的聚(甘油-磷酸)-葡萄糖转移酶lagD乳酸球菌- g 加工和运输ATP 结合蛋白lcnD乳球菌蛋白A 分泌蛋白group_125假定的转座酶group_1420b 型结构域蛋白group_6037RNA 聚合酶因子西格玛70 group_114螺旋-turn-螺旋结构域蛋白
同时又选取了2 株益生菌明星菌株鼠李糖乳杆菌GG、鼠李糖乳杆菌HN001 和一株模式菌株鼠李糖乳杆菌 DSM20021 与鼠李糖乳杆菌Probio-M9 进行BRIG 分析(如图7),结果如表2所示,鼠李糖乳杆菌Probio-M9 相较于市面上的益生菌明星菌株编码更多的关于水解、还原相关酶类,同时还发现鼠李糖乳杆菌Probio-M9 编码一个特别的基因(xly),与黄原胶裂解酶相关[38],黄原胶裂解酶是一种黄原胶修饰酶,对黄原胶的改性及新型黄原胶寡糖的制备具有十分重要的意义。
表2 以鼠李糖乳杆菌Probio-M9 为参考基因组的功能基因组分析
Table 2 Functional genome analysis using L.rhamnosus Probio-M9 as a reference genome

基因名称功能注释malL nfrA1寡核苷酸-1-6-葡萄糖苷酶FMN 还原酶xly黄原胶裂解酶前体bglFPTS 系统β -葡萄糖苷特异性EIIBCA 组分kdgK2 脱氢3 脱氧葡萄糖酸激酶group_3970α / β 水解酶家族蛋白dkgB2,5-二酮-d -葡萄糖酸还原酶ecsAABC 型转运蛋白ATP 结合蛋白ytpP硫氧还蛋白样蛋白group_1823细菌ABC 转运蛋白nhaS3高亲和Na(+)/H(+)反转运体rafAα-半乳糖苷酶group_1420B 型结构域蛋白sftADNA 转位酶pepF1寡肽酶,质粒ctpF假定的阳离子转运ATP 酶bglH假定的聚(甘油-磷酸)-葡萄糖转移酶tagE螺旋-turn-螺旋结构域蛋白group_114螺旋转螺旋结构域蛋白wbbIΒ-1-6-半乳糖呋喃糖基转移酶group_3797糖基水解酶家族25 lcnD乳球菌蛋白A 分泌蛋白prsA折叠酶蛋白前体group_119假定的转座酶DNA 结合结构域蛋白lagD乳酸球菌- g 加工和运输ATP 结合蛋白
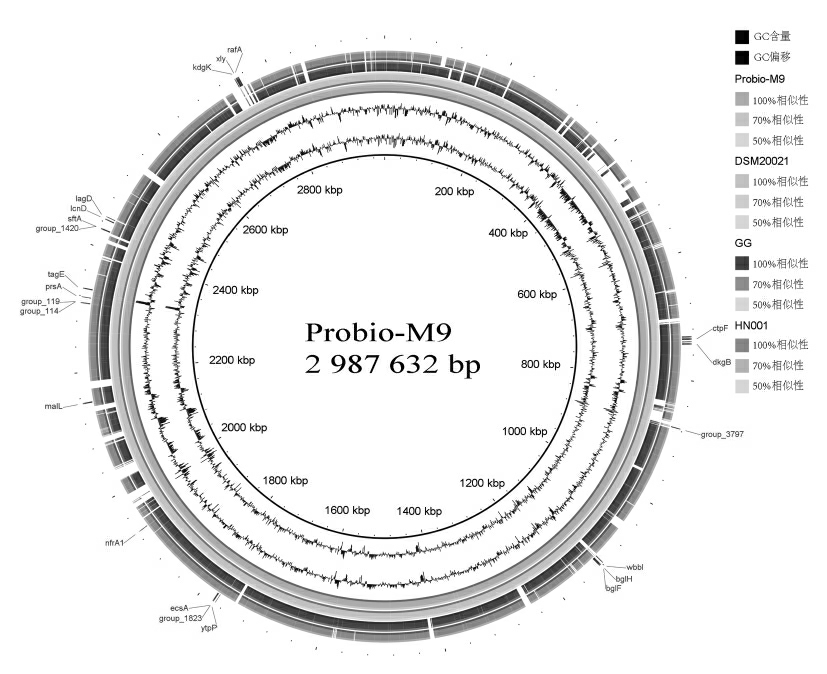
图7 以鼠李糖乳杆菌Probio-M9 为参考基因组的全基因组圈图
Fig.7 Genome-wide cycle map with the reference genome of L.rhamnosus Probio-M9
2.7 益生特性相关基因分析
本研究基于Roary 软件注释到鼠李糖乳杆菌Probio-M9 含有与益生特性相关的基因,具体信息见表3。鼠李糖乳杆菌Probio-M9 含有谷胱甘肽合成(gshAB)、分泌胞外多糖(rmlA~rmlD、epsH)及核黄素合成(ribF)相关基因,同时含有耐酸基因(clpP)。研究表明[39],谷胱甘肽是细胞内调节代谢的重要物质,并且常参与肠黏膜的抗氧化机制,保护肠道免受组织损伤,提高有益菌株在胃肠道中的存活率。鼠李糖乳杆菌EPS 具有益生元、调节机体免疫功能、抗氧化效应、抑制动物脂肪生成和吸收重金属等生理功能[40-43]。本研究还发现,鼠李糖乳杆菌Probio-M9 存在提高宿主代谢能力(tagE)和产乳酸(ldh)相关的基因,可提高菌株益生特性。本研究利用比较基因组学从基因水平揭示了鼠李糖乳杆菌Probio-M9 具有多个益生特性相关基因,认为其是一株具有潜在益生功能的菌株。
表3 鼠李糖乳杆菌Probio-M9 具有的益生特性相关基因
Table 3 Genes related to probiotic properties of L.rhamnosus Probio-M9

?基因名称功能注释益生特性ldhL-乳酸脱氢酶糖酵解过程atpCATP 合酶链黏附分子的产生tagE假定的聚(甘油-磷酸)-葡萄糖转移酶改善宿主代谢gshAB谷胱甘肽生物合成双功能蛋白谷胱甘肽的生物合成ribF核黄素生物合成蛋白核黄素合酶clpPATP 依赖性Clp 蛋白酶的蛋白水解亚基应激反应rmlDDTDP 4 脱氢鼠李糖还原酶胞外多糖生物合成工艺rmlBDTDP 葡萄糖42C6 脱水酶胞外多糖生物合成工艺rmlCDTDP 4 脱氢鼠李糖32C5 差向异构酶碳水化合物代谢,脂多糖生物合成rmlA葡萄糖-1-磷酸胸腺苷转移酶脂多糖生物合成epsH假定的糖基转移酶转移酶的活动
3 结论
本研究以鼠李糖乳杆菌Probio-M9 为例,结合NCBI 数据库214 株鼠李糖乳杆菌基因组序列进行比较基因组学研究。215 株鼠李糖乳杆菌通过247 个核心基因构建系统发育树发现分离源和分离地不存在明显聚类趋势。鼠李糖乳杆菌Probio-M9 处在分支最大的分支Ⅱ中,该分支各菌株之间的遗传关系近、差异很小、区分难度大。然后对分支Ⅱ中98 株鼠李糖乳杆菌进行RAST 注释分析发现,鼠李糖乳杆菌虽在功能方面整体存在高度的相似性,但其中部分菌株与鼠李糖乳杆菌Probio-M9 依然存在着一定差异,鼠李糖乳杆菌Probio-M9 相较于其它鼠李糖乳杆菌有着关于自身代谢、转录、运输等方面更强的调控能力并且含有与益生功能相关的基因,如谷胱甘肽(gshAB)、胞外多糖(rmlA~rmlD、epsH)及提高宿主代谢能力(tagE)相关基因。本文通过比较基因组学分析发现鼠李糖乳杆菌的序列与功能有着高度的相似性,并且鼠李糖乳杆菌Probio-M9 存在益生功能相关的基因,为后续鼠李糖乳杆菌Probio-M9 基因组研究及其益生功能开发奠定遗传学基础。
[1]NAM S O,JAE J Y,LEE Y,et al.Probiotic and anti-inflammatory potential of Lactobacillus rhamnosus 4B15 and Lactobacillus gasseri 4M13 isolated from infant feces[J].PLoS One,2018,13(2):1-15.
[2]MUHAMMAD S R R,HAFIZA M M,MUHAMMAD S,et al.Identification,characterization,and probiotic potential of Lactobacillus rhamnosus isolated from human milk[J].LWT,2017,84(12):271-280.
[3]ALANDER M,SATOKARI R,KORPELA R,et al.Persistence of colonization of human colonic mucosa by a probiotic strain, Lactobacillus rhamnosus GG,after oral consumption[J].Applied and Environmental Microbiology,1999,65(1):351-354.
[4]BERNI C R,NOCERINO R,TERRIN G,et al.Effect of Lactobacillus GG on tolerance acquisition in infants with cow's milk allergy:A randomized trial[J].Journal of Allergy and Clinical Immunology,2012,129(2):580-582.
[5]INTURRI R,STIVALA A,BLANDINO G.Microbiological characteristics of the probiotic strains B.longum BB536 and L.rhamnosus HN001 used in combination[J].Minerva Gastroenterol Dietol,2015,61(4):191-197.
[6]NERMES M,KANTELE J M,ATOSUO T J,et al.Interaction of orally administered Lactobacillus rhamnosus GG with skin and gut microbiota and humoral immunity in infants with atopic dermatitis[J].Clinical& Experimental Allergy,2011,41(3):370-377.
[7]HOLOWACZ S,BLONDEAU C,GUINOBERT I,et al. Lactobacillus salivarius LA307 and Lactobacillus rhamnosus LA305 attenuate skin inflammation in mice[J].Beneficial Microbes,2018,9(2):299-309.
[8]ESCAMILLA J,LANE M A,MAITIN V.Cell-free supernatants from probiotic Lactobacillus casei and Lactobacillus rhamnosus GG decrease colon cancer cell invasion in vitro[J].Nutrition and Cancer,2012,64(6):871-878.
[9]KANKAANPÄÄP T E,EL-NEZAMI H.Binding of aflatoxin B1 alters the adhesion properties of Lactobacillus rhamnosus strain GG in a Caco-2 model[J].Journal of Food Protection,2000,63(3):412-414.
[10]NÄSE L,HATAKKA K,SAVILAHTI E,et al.Effect of long-term consumption of a probiotic bacterium, Lactobacillus rhamnosus GG,in milk on dental caries and caries risk in children[J].Caries Research,2001,35(6):412-420.
[11]HE X L,ZENG Q,PUTHIYAKUNNON S,et al.Lactobacillus rhamnosus GG supernatant enhance neonatal resistance to systemic Escherichia coli K1 infection by accelerating development of intestinal defense[J].Scientific Reports,2017,7(1):43305-43318.
[12]ZHANG Z,ZHOU Z G,LI Y,et al.Isolated exopolysaccharides from Lactobacillus rhamnosus GG alleviated adipogenesis mediated by TLR2 in mice[J].Scientific Reports,2016,6(1):368-374 .
[13]MANZONI P,LISTA G,GALLO E,et al.Routinary probiotic Lactobacillus rhamnosus GG administration in preterm very-low-birth-weight neonates:A retrospective,6-year cohort study from two large tertiary NICUs in Italy[J].Early Human Development,2009,85(10):594-565.
[14]SKÓRKA A,PIESCIK-LECH M,KOŁODZIEJ M,et al.To add or not to add probiotics to infant formulae? An updated systematic review[J].Beneficial Microbes,2017,8(5):717-725.
[15]Liu W J,CHEN M X,DUO L.Characterization of potentially probiotic lactic acid bacteria and bifidobacteria isolated from human colostrum[J].Journal of Dairy Science,2020,103(9):17602-17615.
[16]KALINOWSKI J,BATHE B,BARTELS D,et al.The complete Corynebacterium glutamicum ATCC 13032 genome sequence and its impact on the production of L-aspartate-derived amino acids and vitamins[J].Journal of Biotechnology,2003,104(1/3):5-25.
[17]SUN Z H,HARRIS H M B,MCCANN A,et al.Expanding the biotechnology potential of Lactobacilli through comparative genomics of 213 strains and associated genera[J].Nature Communications,2015,29(6):8322-8331.
[18]YU J,SONG Y Q,REN Y,et al.Genome-level comparisons provide insight into the phylogeny and metabolic diversity of species within the genus Lactococcus[J].BMC Microbiology,2017,17(3):213-248.
[19]孙靓.鼠李糖乳杆菌利用木薯淀粉发酵产乳酸工艺及组学研究[D].南宁:广西大学,2018.SUN L.Study on L-lactic acid production from cassavausing Lactobacillus rhamnosus with genomice and transcriptomics analysis[D].Nanning:Guangxi University,2018.
[20]钟智,孙天松,陈永福.基因组分析揭示Streptococcus thermophilus ND-07 富产胞外多糖分子机制[J].中国乳品工业,2018,46(4):9-11,21.ZHONG Z,SUN T S,CHEN Y F.Genomic insight into the high exopolysaccharides-producing bacterium Streptococcus thermophilus ND-07[J].Dairy Industry,2018,46(4):9-11,21.
[21]刘文君,吕瑞瑞,李伟程,等.基于比较基因组学揭示不同植物乳杆菌的遗传特征及菌株差异——以Lactobacillus plantarum P9 和Lp-6 研究为例[J].微生物学报,2021,61(8):2370-2381.LIU W J,LÜ R R,LI W C,et al.Comparative genomics revealed genetic characteristics of different Lactobacillus plantarum strains:Using P9 and Lp-6 strains as examples[J].Acta Microbiologica Sinica,2021,61(8):2730-2381.
[22]MARTIN H,NISHADI D S,THOMAS D O,et al.Circlator:Automated circularization of genome assemblies using long sequencing reads[J].Genome Biology,2015,16(1):294-304.
[23]SEEMANN T.Prokka:Rapid prokaryotic genome annotation[J].Bioinformatics,2014,30(14):2068-2069.
[24]PAGE A J,CUMMINSET C A,HUNT M,et al.Roary:Rapid large-scale prokaryote pan genome analysis[J].Bioinformatics,2015,31(22):3691-3693.
[25]吴琼,李伟程,李敏,等. Limosilactobacillus fermentum F-6 的遗传背景和功能基因组[J].微生物学报,2021,62(4):1438-1451.WU Q,LI W C,LI M,et al.Genetic background and functional genome of Limosilactobacillus fermentum F-6[J].Acta Microbiologica Sinica,2021,62(4):1438-1451.
[26]JAIN C,RODRIGUEZ-R L M,PHILLIPPY A M,et al.High throughput ANI analysis of 90K prokaryotic genomes reveals clear speciesboundaries[J].Nature Communications,2018,9(1):5114-5121.
[27]CHEN C J,CHEN H,ZHANG Y,et al.TBtools:An integrative Toolkit developed for interactive analyses of big biological data [J].Molecular Plant,2020,13(8):1-8.
[28]ARBOLEYA S,BOTTACINI F,O'CONNELL -MOTHERWAY M,et al.Gene-trait matching across the Bifidobacterium longum pan -genome reveals considerable diversity in carbohydrate catabolism a mong human infant strains[J].BMC Genomics,2018,19(1):33-49.
[29]KANT R,RINTAHAKA J,YU X,et al.A comparative pan-genome perspective of niche-adaptable cell -surface protein phenotypes in Lactobacillus rhamnosus[J].PLoS One,2014,9(7):102762-102777.
[30]宋宇琴.德氏乳杆菌保加利亚亚种的群体遗传学和功能基因组学研究[D].呼和浩特:内蒙古农业大学,2018.SONG Y Q.Study on population genetics and functional genomics of Lactobacillus delbrueckii subsp.bulgaricus [D].Hohhot:Inner Mongolia Agricultural University,2018.
[31]ARAHAL D R.Whole-genome analyses:Average nucleotide identity [J].Methods in Microbiology,2014,12(41):103-122.
[32]CIUFO S,KANNAN S,SHARMA S,et al.Using average nucleotide identity to improve taxonomic assignments in prokaryotic genomes at the NCBI[J].International Journal of Systematic and Evolutionary Microbiology,2018,68(7):2386-2392.
[33]NISSILA E,DOUILLARD F P,RITARI J,et al.Genotypic and phenotypic diversity of Lactobacillus rhamnosus clinical isolates,their comparison with strain GG and their recognition by complement sys tem[J].PLoS One,2017,12(5):0176739-0176742.
[34]ARNOLD J W,SIMPSON J B,ROACH J,et al.Prebiotics for lactose intolerance:Variability in galacto-oligosaccharide utilization by intestinal Lactobacillus rhamnosus[J].Nutrients,2018,10(10):1-17.
[35]剧柠.西藏,新疆和云南地区传统发酵乳制品中乳杆菌的生物多样性研究[D].呼和浩特:内蒙古农业大学,2009.JU N.Biodiversity of Lactobacilli from traditional fermented milk in Tibet,Xinjiang and Yunnan of China[D].Hohhot:Inner Mongolia Agricultural University,2009.
[36]RUIZ L,GUEIMONDE M,COUTE Y,et al.Margolles,evaluation of the ability of Bifidobacterium longum to metabolize human intestinal mucus [J].FEMS Microbiol Lett,2011,314(2):125-130.
[37]PENG C,WANG Y H,LIU F,et al. FLOURY ENDOSPERM6 encodes a CBM48 domain‐containing protein involved in compound granule formation and starch synthesis in rice endosperm[J].The Plant Journal,2014,77(6):917-930.
[38]杜元元,郭小宇,李鹤,等.一种新型黄原胶裂解酶的异源表达及酶学性质表征[J].食品工业科技,2017,38(2):175-181.DU Y Y,GUO X Y,LI H,et al.Heterologous expression and characterization of a novel xanthan lyase[J].Science and Technology of Food Industry,2017,38(2):175-181.
[39]NISHIO Y,NAKAMURA Y,USUDA Y,et al.Evolutionary process of amino acid biosynthesis in corynebacterium at the whole genome level[J].Narnia,2004,21(9):719-732.
[40]PERAN L,CAMUESCO D,COMALAND M,et al.Lactobacillus fermentum,a probiotic capable to release glutathione,prevents colonic inflammation in the TNBS model of rat colitis[J].International Journal of Colorectal Disease,2006,21(8):737-746.
[41]KONIECZNA C,SŁODZINSKI M,SCHMIDT M T.Exopolysaccharides produced by Lactobacillus rhamnosus KL 53A and Lactobacillus casei Fyos affect their adhesion to enterocytes[J].Polish Journal of Microbiology,2018,67(3):273-281.
[42]NOWAK B,SRÓTTEK M,CISZEK-LENDA M,et al.Exopolysaccharide from Lactobacillus rhamnosus KL37 inhibits T cell-dependent immune response in mice[J].Archivum Immunologiae et Therapiae Experimentalis,2020,68(3):1-11.
[43]CISZEK-LENDA M,NOWAK B,SRÓTTEK M,et al.Immunore_gulatory potential of exopolysaccharide from Lactobacillus rhamnosus KL37.Effects on the production of inflammatory mediators by mouse macrophages[J].International Journal of Experimental Pathology,2011,92(6):382-391.