副干酪乳酪杆菌作为乳杆菌科中具应用价值的菌种,是干酪乳酪杆菌和鼠李糖乳酪杆菌的近缘物种。副干酪乳酪杆菌是国家卫生健康委员会发布的可用于食品的菌种之一,其常用来制备益生菌和发酵乳制品制剂[1]。然而,副干酪乳酪杆菌的系统命名和分类一直存在争议,特别是随着分类学技术的进步,该物种的模式株、系统分类和命名几经变迁[2]。1989 年Collins 等[3]通过DNA-DNA杂交分析发现干酪乳杆菌(Lactobacillus casei)中存在大量与模式菌株ATCC 393T 同源性较低而菌株间同源性较高的菌株,因此建议干酪乳杆菌阿拉伯糖亚种(Lactobacillus casei subsp.alactosus)、干酪乳杆菌假植物亚种(Lactobacillus casei subsp.pseudoplantarum)、干酪乳杆菌坚韧亚种(Lactobacillus casei subsp.tolerans)以及大部分的干酪乳杆菌干酪亚种(Lactobacillus casei subsp.casei)划分为一个新的独立菌种即副干酪乳杆菌(Lactobacillus paracasei)。2020 年副干酪乳杆菌更名为副干酪乳酪杆菌(Lacticaseibacillus paracasei),其模式菌株为ATCC 25302T,并保留了副干酪乳酪杆菌副干酪亚种(L.paracasei subsp.paracasei)和副干酪乳酪杆菌坚韧亚种(L.paracasei subsp.tolerans)2 个亚种[4-5]。
干酪乳杆菌Zhang(Zhang)是本研究团队于2001 年从内蒙古锡林郭勒盟正蓝旗大草原传统发酵酸马奶中分离得到的1 株益生菌[6-7],其具有较强耐人工胃液[8]、胆盐消化[9]、免疫调节[10]、抗氧化[11]等益生功能。2008 年,本研究团队经生理生化鉴定和16S rDNA 序列分析将Zhang 的分类学地位鉴定为干酪乳杆菌[12-13]。干酪乳杆菌与其近缘物种间的亲缘关系错综复杂,基于传统的分类学方法和16S rDNA 序列很难从种水平上进行区分[14]。本研究团队于2010 年完成了Zhang 全基因组测序,其是我国第1 株完成全基因组测序的乳酸菌菌株,经进一步比较基因组学分析确认其的分类学地位为副干酪乳酪杆菌[15]。由于副干酪乳酪杆菌分类学地位的变迁和更新,Zhang 的分类学地位需进一步确认。此外,目前尚无关于该菌株耐药基因、致病性基因和环境抗性基因的研究,限制了其进一步开发和应用。
《益生菌的科学共识》(2020 版)中建议针对益生菌菌株的安全性评估,应基于全基因组测序生物信息分析,阐述待评价益生菌菌株所携带的耐药基因、致病性基因和环境抗性基因的特征[16]。基于此,本研究利用比较基因组学方法解析Zhang的分类学地位和遗传背景,评估Zhang 的耐药基因、致病性基因和环境抗性基因特征,揭示其基因组上携带的细菌素基因簇及潜在益生基因,旨在为菌株进一步开发及产业化提供数据参考。
1 材料与方法
1.1 基因组序列下载
截至2023 年3 月9 日,采用ncbi-genomedownload 软件将NCBI(National Coalition Building Institute,https:// www.ncbi.nlm.nih.gov/)Refseq 数据库中318 株副干酪乳酪杆菌基因组完成图全部下载完成。
本研究团队前期已完成Zhang 连续传代过程遗传稳定性的研究(已接收,未见刊),对第0,25,50,75,100 代(每代3 组平行)共15 株菌进行了基因组测序和组装工作。
1.2 比较基因组分析
1.2.1 基因组信息统计 采用李伟程[17]的方法,统计所有副干酪乳酪杆菌的基因组大小、GC 含量以及蛋白质编码区(Coding sequence,CDS)。
1.2.2 分类学地位分析和平均核苷酸一致性计算 本研究使用blastall 软件[18]将Zhang 与GTDB 数据库(http://gtdbecogenomic.org/)进行比 对,e 值小于e×10-10。使用fastANI 软件[19]计算335 株副干酪乳酪杆菌的平均核苷酸一致性(Average nucleotide identity,ANI)值,比对片段长度设置为1 000。
1.2.3 核心基因系统发育树 使用prokka 软件对菌株进行注释,通过Roary[20]软件分析得到的核心基因序 列,采 用TreeBeST[21]软 件(http://www.mybiosoftware.com/treebest/)基于邻接法(Neighbor-joining,NJ)进行系统发育树构建,自举值为1 000。通过iTol 在线软件(https://itol.embl.de/)进行系统发育树可视化。
1.2.4 单核苷酸多态性(SNP)系统发育树的构建 本研究以副干酪乳酪杆菌ATCC 334 的全基因组完成图序列(GCF_000014525.1)作为参考基因组,使用Snippy 软件[22](V4.6.0)中snippy-multi 组件计算副干酪乳酪杆菌群体的核心SNP(Core-SNP)。
采 用TreeBeST[21]软 件(http://www.mybiosoftware.com/treebest/)中的邻接法进行系统发育树构建,自举值为200。在iTol 在线软件(https://itol.embl.de/)中进行系统发育树可视化。
1.2.5 耐药基因、致病性基因和环境抗性基因注释 将Zhang 基因组序列分别上传至在线注释CARD 数据库(http://card.mcmaster.ca/,采 用RGI5.1.0 软件中的Perfect 和Strict 算法为筛选标准)、ResFinder 数据库(https://cge.food.dtu.dk/services/ResFinder-4.1/),序列相似性>90%,序列覆盖度>60%、VFDB 数据库(Virulence Factors of Pathogenic Bacteria,http://www.mgc.ac.cn/VFs/main.htm/),序列相似性>90%,序列覆盖度>60%和VirulenceFinder 数据库(https://cge.food.dtu.dk/services/Virulence Finder/),序列相似性>90%,序列覆盖度>60%进行注释耐药基因、致病性基因和环境抗性基因。
1.2.6 共线性分析 以副干酪乳酪杆菌ATCC 334 为参考序列,使用Mauve 软件[23]分析ATCC 334T、Zhang 和PC-01 之间的共线性关系。
1.2.7 细菌素基因簇分析 利用BAGEL4 在线数据 库(http://bagel4.molgenrug.nl/index.php),对Zhang 中核糖体合成、翻译后修饰肽和细菌素产生基因簇进行注释[24]。
1.2.8 潜在益生基因分析 参照刘旭等[25]和吴琼等[26]的方法利用Prokka 软件对菌株基因组进行基因预测后,挖掘菌株潜在益生基因。
2 结果与讨论
2.1 基因组特征
本研究团队前期已完成Zhang 的基因组测序和组装,发现Zhang 包含1 条染色体和1 个质粒,其基因组大小为2.90 Mb,GC 含量为46.43%,其中染色体大小为2.73 Mb,GC 含量为46.51%;质粒大小为36.08 kb,GC 含量为39.65%,共包含了2 694 个蛋白质编码区[15]。
本研究团队前期完成的副干酪乳酪杆菌PC-01 的基因组测及序组装[27],发现基因组仅有1 条染色体组成。其基因组大小为2.82 Mb,GC 含量为46.64%,包含了2 738 个蛋白质编码区。王静等[28]研究发现副干酪乳酪杆菌基因组大小为2.38~3.29 Mb,平均为2.98 Mb;GC 含量为46.00%~46.60%,平均为46.33%;平均包含了2 777 个蛋白质编码区。本研究发现副干酪乳酪杆菌基因组大小为(2.87±0.15)Mb;GC 含量为(46.31±0.25)%;包含了2 875.8±164.8 个蛋白质编码区。
2.2 分类学地位确定
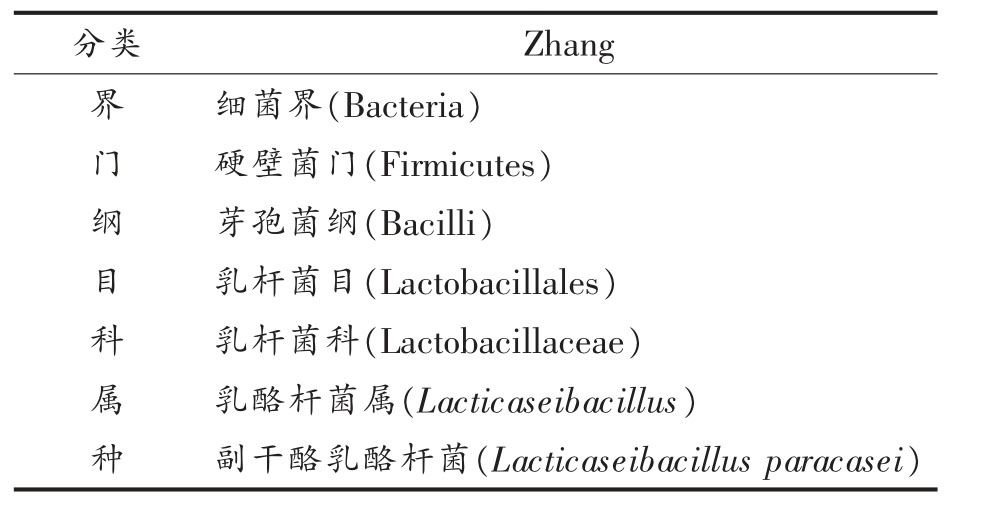
GTDB 数据库使用相对进化差异来描述高等级的分类单元并划分物种集群、限定分类单元,因此该数据库可以实现基因组在物种水平的分类[29]。本研究使用GTDB 数据库对Zhang 的分类学地位进行确定,结果显示其为副干酪乳酪杆菌。
ANI 通过在全基因组学水平进行基因组的同源序列比对从而鉴定菌株的亲缘关系[30]。比较基因组学中,一般将ANI 值大于95%视为同一物种[31]。结果显示近缘物种干酪乳酪杆菌ATCC 393T、鼠李糖乳酪杆菌DSM 20021T 与334 株副干酪乳酪杆菌的ANI 值小于95%,333 株副干酪乳酪杆菌之间ANI 值均大于95%,表明其为同一物种。Zhang 与模式菌株ATCC 25302T 的ANI 值是98.49%,大于95%,说明Zhang 与副干酪乳酪杆菌ATCC 25302T 为同一物种。上述结果表明,Zhang的分类学地位应为副干酪乳酪杆菌(Lacticaseibacillus paracasei)。
2.3 系统发育分析
系统发育树可直观反映菌株间群体结构和遗传进化关系[32]。为了分析Zhang 与公共数据库中318 株副干酪乳酪杆菌的系统发育关系,本研究以近缘物种干酪乳酪杆菌ATCC 393T 和鼠李糖乳酪杆菌DSM 20021T 作为外群,基于59 个核心基因的核酸序列,通过邻接法构建系统发育树。由图2a 可知,核心基因系统发育树分为3 个大分支,分别为作为外群的干酪乳酪杆菌ATCC 393T、鼠李糖乳酪杆菌DSM 20021T 以及333 株副干酪乳酪杆菌,表明不同物种在核心基因系统发育树上具有较大差异。此外,Zhang 与模式菌株副干酪乳酪杆菌ATCC 25302T 聚集在同一大分支中,该结果支持Zhang 为副干酪乳酪杆菌。Zhang 原始菌株与其第0,25,50,75,100 代菌株聚集在同一小分支,与副干酪乳酪杆菌SCB0563 的遗传距离较近。对干酪乳酪杆菌ATCC 393T、鼠李糖乳酪杆菌DSM 20021T、副干酪乳酪杆菌ATCC 25302T 和副干酪乳酪杆菌坚韧亚种DSM 20258T 以及Zhang的335 个核心基因的核酸序列,使用最大似然法(Maximum likelihood,ML)构建系统发育树。由图2b 可知,副干酪乳酪杆菌ATCC 25302T、副干酪乳酪杆菌坚韧亚种DSM 20258T 和Zhang 聚集在同一进化枝上,再次验证了Zhang 的分类学地位为副干酪乳酪杆菌。然而,由于不同物种之间差异较大,因此无法判断Zhang 亚种水平的分类学地位。本研究团队前期通过比较16S rRNA、看家基因和核心基因的系统发育树,发现副干酪乳酪杆菌与亚种干酪乳酪杆菌ATCC 393T、鼠李糖乳酪杆菌DSM 20021T 遗传差异较大,基于核心基因构建的系统发育树可以较好的区分干酪乳酪杆菌的近缘种[14]。

图1 333 株副干酪乳酪杆菌基因组序列的ANI 值
Fig.1 ANI value of the genome sequences of 333 strains of L.paracasei
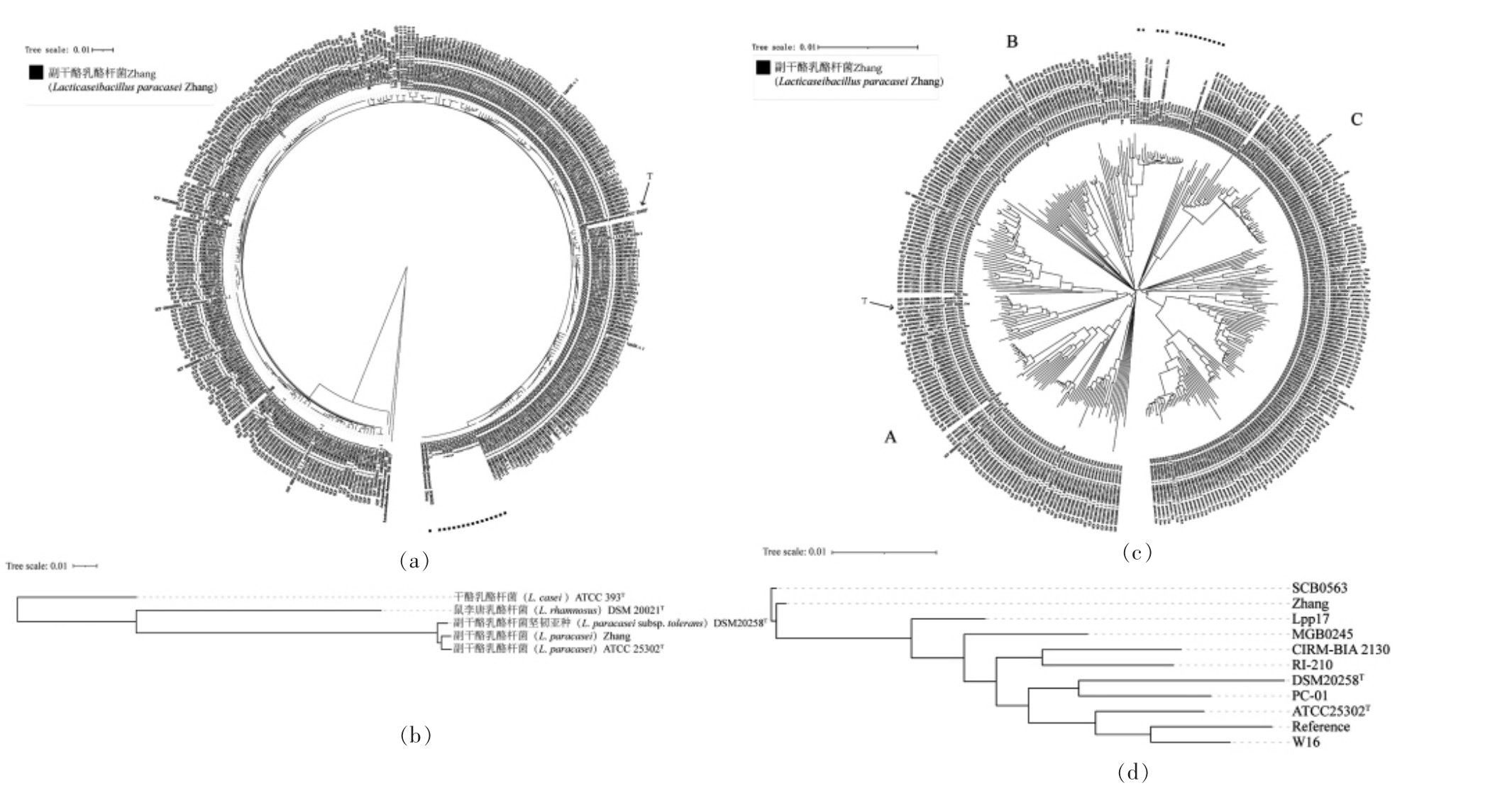
图2 副干酪乳酪杆菌系统发育树
Fig.2 Phylogenetic tree of Lactobacillus paracasei
注:a 图和b 图为基于核心基因构建的系统发育树,c 图和d 图为基于SNP 构建的系统发育树。

图3 副干酪乳杆菌基因组多序列比对
Fig.3 Multiple sequence alignment of the L.paracasei genomic
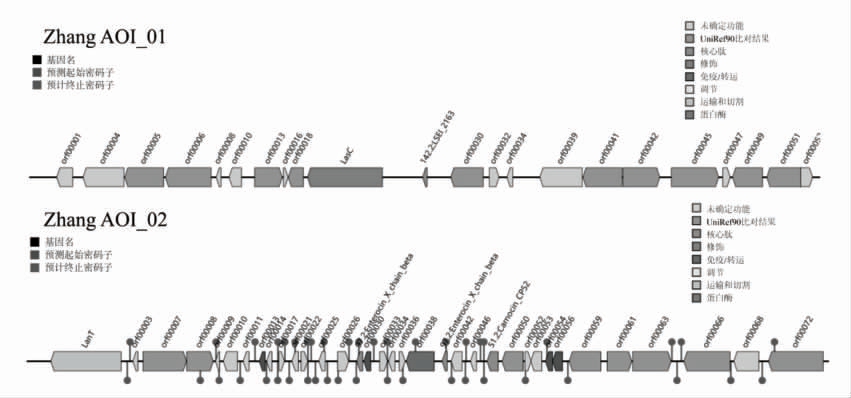
图4 Zhang 细菌素基因簇
Fig.4 Bacteriocin gene cluster of Zhang
表1 Zhang 的GTDB 数据库比对结果
Table 1 The alignment results of the GTDB database of Zhang
由于外群菌株与333 株副干酪乳酪杆菌之间的遗传距离较远,故进一步基于以副干酪乳酪杆菌ATCC 334 的全基因组完成图序列作为参考基因组进行Core-SNP 分析,并使用NJ 法构建系统发育树。结果如图2c 所示,SNP 系统发育树分为3个大分支,Zhang 原始菌株与其 第0,25,50,75,100 代菌株聚集在分支B 的同一小分支,其相互之间略有差异,且与副干酪乳酪杆菌SCB0563 的遗传距离较近。以副干酪乳酪杆菌ATCC 334 的全基因组完成图序列作为参考基因组,选取各分支选取代表菌株进行Core-SNP 分析,并使用ML法构建系统发育树,结果如图2d 所示。Zhang 的分类学地位为副干酪乳酪杆菌,然而基于核心基因和Core-SNP 都无法在副干酪乳酪杆菌的亚种水平上对其进行区分。本研究团队前期发现不同代数15 株菌所识别到的SNP 位点均小于21 个,表明干酪乳杆菌Zhang 在MRS 液体培养基中连续传代过程中有较好的遗传稳定性(已接收,未见刊)。Fontana 等[33]研究发现副干酪乳酪杆菌群体在系统发育树中大致可划分为4 个分支,与本研究系统发育树聚类结果一致。综上所述,通过ANI、GTDB 数据库注释、基于核心基因的系统发育分析和基于Core-SNP 的系统发育分析结果一致,表明Zhang 的分类学地位应为副干酪乳酪杆菌。
2.4 共线性分析
共线性可通过对同一物种不同个体基因组间同源序列及其排列顺序一致性的检测,来探究基因组间的相关性[34]。共线性分析结果显示,Zhang与ATCC 334 基因组共线性良好。然而,Zhang 在其基因组328.49 kb 位置处存在其49.37 kb 处的特有片段,在Zhang 基因组392.52 kb 处存在PC-01 没有的片段,而ATCC 334 存在此片段。Zhang在其基因组1.81 Mb 位置处存在19.55 Kb 的特有片段,包含酪氨酸蛋白激酶(yveL)和荚膜多糖生物合成蛋白质(ywqC)等基因。Zhang 基因组在511.30 kb 处长度为311.29 kb 的片段在PC-01的基因组中发生了基因组易位。以上结果表明,Zhang 和PC-01 存在较大遗传差异。有研究表明当一个基因刚好位于易位区域边缘时基因会被打断,促使基因内区、非翻译区域以及编码区域改变,导致DNA 序列有较大的改变[35]。
2.5 耐药基因、致病性基因和环境抗性基因的特征
比较基因组学分析可对乳酸菌基因组所含有的耐药、致病性、环境抗性等相关基因的结构和功能信息进行分析,对乳酸菌在基因层面的安全性进行评估,一定程度上能有效地反映供试乳酸菌的生物安全性[36-37]。通过生物信息学分析发现,Zhang 及其第0,25,50,75,100 代菌株的基因组上均检测到了1 种抗生素外排泵基因qacj,相似度为38.24%,小于40%,故认为不具有该耐药基因。与此同时,并未在Zhang 及其第0,25,50,75,100代菌株的基因组上检测到致病性基因和环境抗性基因。综上,Zhang 及其第0,25,50,75,100 代菌株在基因组上不具有耐药基因、致病性基因和环境抗性基因。
2.6 细菌素基因簇分析
细菌素是指细菌在代谢过程中合成并分泌的具有抗菌活性的物质,对其自身没有抑菌活性,而对亲缘关系较近的菌株具有抑制作用[38]。相关研究表明,可以通过基因挖掘工具发现新的细菌素基因簇[39-40]。通过细菌素基因簇注释,发现Zhang有2 个潜在的热点区域(Areas of interest,AOI),以操纵子的结构排列。AOI_01 由36 个开放阅读框(Open reading frames,ORFs)组成,包含Ⅱc 细菌素相关核心肽LSEI_2386。在基因簇的上游有天冬酰胺合成酶(LasC)及orf00005 编码细菌素生成相关的乳酸脱氢酶。下游位置的orf00045 编码细菌素生成相关的乙酸激酶。AOI_02 由37 个ORFs 组成,核心肽包括Enterocin_X_chain_beta、Carnocin_CP52。在基因簇的上游有ABC 转运蛋白(LanT)及orf0007 编码细菌素生成相关的组氨酸激酶,中游位置的orf00038 编码免疫蛋白PlnI。PlnI 属于CAAX 氨基蛋白酶家族,在其它细菌素基因簇中这种类CAAX 基因十分罕见,目前其在细菌素合成过程中的作用尚不明确[41]。下游位置orf0007 编码细菌素生成相关的氧化还原酶YdhF。瞿茜楠等[42]发现副干酪乳酪杆菌PC-724 的细菌素基因簇含有细菌素Carnocin 基因、专用免疫蛋白基因、ABC 转运蛋白基因及其辅助蛋白基因,推测其可产生细菌素。综上所述,推测Zhang 可能具有产生细菌素Carnocin 的潜力。
2.7 潜在益生基因
Zhang 发现有15 个潜在益生特性相关基因(如表2 所示)。吕瑞瑞等[27]在Zhang 中注释到与糖基转移、转录调节等代谢功能相关基因(epsE、yybR、mshA)。本研究还注释到了谷胱甘肽合成(gshAB)、核黄素合成(ribF、ribU)、黏附因 子(atpC)、分泌胞外多糖(rmlA、rmlB、rmlD、epsE、epsJ、epsH)、耐酸基因(clpP)、提高宿主代谢能力(tagE)、乳酸和转运乳酸(pyk)产生相关的基因。rmlA 作为葡萄糖-1-磷酸胸苷酰基转移酶,能催化D-葡萄糖-1 转化为dTDP-葡萄糖。已有研究证明rmlA 基因与细菌形态及耐受性相关[43]。据报道谷胱甘肽对于维持正常的免疫系统、整合解毒、抗氧化等方面都具有积极的影响[44]。此外谷胱甘肽也有助于菌株在胃肠道存活率的增加[45],利于菌株益生功能的发挥。
表2 副干酪乳酪杆菌Zhang 具有的潜在益生基因
Table 2 Potential probiotic genes of L.paracasei Zhang

3 结论
本研究对Zhang 进行比较基因组学分析发现,Zhang 的分类学地位为副干酪乳酪杆菌。Zhang在基因组上不含有耐药基因、致病性基因和环境抗性基因。此外,Zhang 携带核心肽、LanT 转运和引导切割及免疫或运输功能的Carnocin 相关细菌素基因簇,推测其可能具有产生细菌素Carnocin的潜力。Zhang 还携带谷胱甘肽合成(gshAB)、核黄素合成(ribF)、黏附因子(atpC)、耐酸基因(clpP)和分泌胞外多糖(rmlA、epsH)等潜在益生基因。本研究明确了Zhang 的分类学地位为副干酪乳酪杆菌,评估发现其在基因组上不具有耐药基因、致病性基因和环境抗性基因,并注释发现完整的细菌素基因簇及谷胱甘肽生物合成等潜在益生基因,为其进一步开发研究以及产业化提供了遗传学基础。
[1] 国家卫生健康委员会.国家卫生健康委员会公告(2022 年第4 号)[EB/OL].(2022-08-18)[2023-05-03]https://www.nhc.gov.cn/sps/s7892/202208/1d6c229d6f 744b35827e98161c146afb.shtml.National Health Commission.Announcement of the National Health Commission(No.4 of 2022)[EB/OL].(2022-08-18)[2023-05-03].https://www.nhc.gov.cn/sps/s7892/202208/1d6c229d6f744b35827e98161c146 afb.shtml.
[2] 张哲,于学健,曹艳花,等.干酪乳杆菌Lactobacillus casei 及相近菌种的分类变迁[J].食品与发酵工业,2020,46(16):243-248.ZHANG Z,YU X J,CAO Y H,et al.Taxonomic changes of Lactobacillus casei and its closely raleted species[J].Food and Fermentation Industries,2020,46(16):243-248.
[3] COLLINS M D,PHILLIPS B A,ZANONI P,Deoxyribonucleic acid homology studies of Lactobacillus casei,L.paracasei sp.nov.Subsp.paracasei and subsp.tolerans,and Lactobacillus rhamnosus sp.Nov.,comb.nov[J].Int J Syst Bacteriol,1989,39(2):105-108.
[4] WARD L,TIMMINS M.Differentiation of Lactobacillus casei,Lactobacillus paracasei and Lactobacillus rhamnosus by polymerase chain reaction[J].Letters in Applied Microbiology,1999,29(2):90-92.
[5] 刘光全,刘勇,李辉,等.pheS 基因序列分析在干酪乳杆菌群种水平鉴定中的应用[J].食品与发酵工业,2011,37(9):94-97.LIU G Q,LIU Y,LI H,et al.Species identification of the Lactobacillus casei group by pheS gene sequences analysis[J].Food and Fermentation Industries,2011,37(9):94-97.
[6] WU R N,WANG L P,WANG J C,et al.Isolation and preliminary probiotic selection of Lactobacilli from koumiss in Inner Mongolia[J].Journal of Basic Microbiology,2009,49(3):318-326.
[7] GUO Z,WANG J C,YAN L Y,et al.In vitro comparison of probiotic properties of Lactobacillus casei Zhang,a potential new probiotic,with selected probiotic strains [J].LWT -Food Science and Technology,2009,42(10):1640-1646.
[8] WU R N,WANG W W,YU D L,et al.Proteomics analysis of Lactobacillus casei Zhang,a new probiotic bacterium isolated from traditional homemade koumiss in Inner Mongolia of China[J].Molecular & Cellular Proteomics,2009,8(10):2321-2338.
[9] WU R,SUN Z,WU J,et al.Effect of bile salts stress on protein synthesis of Lactobacillus casei Zhang revealed by 2-dimensional gel electrophoresis[J].Journal of Dairy Science,2010,93(8):3858-3868.
[10] YA T,ZHANG Q J,CHU F L,et al.Immunological evaluation of Lactobacillus casei Zhang:A newly isolated strain from koumiss in Inner Mongolia,China[J].BMC Immunology,2008,9:1-9.
[11] ZHANG Y,DU R T,WANG L F,et al.The antioxidative effects of probiotic Lactobacillus casei Zhang on the hyperlipidemic rats[J].European Food Research and Technology,2010,231:151-158.
[12] WANG J G,CHEN X,LIU W,J et al.Identification of Lactobacillus from koumiss by conventional and molecular methods[J].European Food Research and Technology,2008,227:1555-1561.
[13] 刘文俊,乌日娜,张和平.益生菌L.casei Zhang 的多项分类鉴定[J].中国乳 品工业,2009,37(2):14-18.LIU W J,WU R N,ZHANG H P.Classification of the probiotics L.casei Zhang with polyphasic taxonomy[J].China Dairy Industry,2009,37(2):14-18.
[14] 多拉娜,宋宇琴,李伟程,等.干酪乳杆菌的近缘种及亚种部分看家基因的系统发育分析[J].微生物学通报,2018,45(12):2751-2761.DUO L N,SONG Y Q,LI W C,et al.Phylogenetic analysis of Lactobacillus casei and closely related species/subspecies based on sequences of housekeeping genes[J].Microbiology China,2018,45(12):2751-2761.
[15] ZHANG W Y,YU D L,SUN Z H,et al.Complete genome sequence of Lactobacillus casei Zhang,a new probiotic strain isolated from traditional homemade koumiss in Inner Mongolia,China [J].Journal of Bacteriology,2010,192(19):5268-5269.
[16] AUTHORITY E F S.EFSA statement on the requirements for whole genome sequence analysis of microorganisms intentionally used in the food chain[J].EFSA Journal,2021,19(7):e06506.
[17] 李伟程.自然发酵乳中乳酸乳球菌乳酸亚种群体遗传学和功能基因组学研究[D].呼和浩特:内蒙古农业大学,2019.LI W C.Population genetics and functional genomics of lactic acid subspecies of Lactococcus lactis in naturally fermented milk[D].Hohhot:Inner Mongolia Agricultural University,2019.
[18] PARKS D H,CHUVOCHINA M,WAITE D W,et al.A standardized bacterial taxonomy based on genome phylogeny substantially revises the tree of life[J].Nature Biotechnology,2018,36(10):996-1004.
[19] JAIN C,RODRIGUEZ-R L M,PHILLIPPY A M,et al.High throughput ANI analysis of 90K prokaryotic genomes reveals clear species boundaries[J].Nature Communications,2018,9(1):5114.
[20] PAGE A J,CUMMINS C A,HUNT M,et al.Roary:Rapid large-scale prokaryote pan genome analysis[J].Bioinformatics,2015,31(22):3691-3693.
[21] VILELLA A J,SEVERIN J,URETA-VIDAL A,et al.EnsemblCompara GeneTrees:Complete,duplica tion-aware phylogenetic trees in vertebrates[J].Genome Research,2009,19(2):327-335.
[22] SEEMANN T.Snippy:Rapid haploid variant calling and core genome alignment[EB/OL].(2020-08-04)[2023-05-01].GitHub https://github.com/tseemann/snippy.
[23] DARLING A C,MAU B,BLATTNER F R,et al.Mauve:Multiple alignment of conserved genomic sequence with rearrangements[J].Genome Research,2004,14(7):1394-1403.
[24] VAN HEEL A J,DE JONG A,SONG C,et al.BAGEL4:A user-friendly web server to thoroughly mine RiPPs and bacteriocins[J].Nucleic Acids Research,2018,46(W1):W278-W281.
[25] 刘旭,李伟 程,王可欣,等.Lactobacillus paragasseri IMAU FB017 的遗传背景和益生相关基因分析[J/OL].微生物学报,(2022-11-16)[2023-05-03].https://kns.cnki.net/kcms2/article/abstract?v=3uoqIhG 8C45S0n9fL2suRadTyEVl2pW9UrhTDCdPD65MJ_TzIdondkyQuHaWNhAUqnC7z8WpqeaA9QgEiW8YHAQ1 qtksvsr1&uniplatform=NZKPT.LIU X,LI W C,WANG K X,et al.Genetic background and probiotic-related gene analysis of Lactobacillus paragasseri IMAU FB017[J/OL].Acta Microbiologica Sinica,(2022-11-16)[2023-05-03].https://kns.cnki.net/kcms2/article/abstract?v=3uoqIhG 8C45S0n9fL2suRadTyEVl2pW9UrhTDCdPD65MJ_TzIdondkyQuHaWNhAUqnC7z8WpqeaA9QgEiW8YHAQ1 qtksvsr1&uniplatform=NZKPT.
[26] 吴琼,李伟程,李敏,等.Limosilactobacillus fermentum F-6 的遗传背景和功能基因组[J].微生物学报,2022,62(4):1438-1451.WU Q,LI W C,LI M,et al.Genetic background and functional genome of Limosilactobacillus fermentum F-6[J].Acta Microbiologica Sinica,2022,62(4):1438-1451.
[27] 吕瑞瑞,李伟程,康小红,等.副干酪乳杆菌PC-01 全基因组测序及不同副干酪乳杆菌菌株比较基因组学分析[J].微生物学通报,2021,48(9):3025-3038.LÜ R R,LI W C,KANG X H,et al.Whole genome sequencing of Lactobacillus paracasei PC-01 and comparative genomics analysis about Lactobacillus paracasei strains[J].Microbiology China,2021,48(9):3025-3038.
[28] 王静,张文羿.副干酪乳杆菌系统发育组学分析[J].基因组学与应用生物学,2022,41(7):1461-1474.WANG J,ZHANG W Y.Phylogenomics analysis of Lactobacillus paracasei[J].Genomics and Applied Biology,2022,41(7):1461-1474.
[29] PARKS D H,CHUVOCHINA M,RINKE C,et al.GTDB:An ongoing census of bacterial and archaeal diversity through a phylogenetically consistent,rank normalized and complete genome-based taxonomy[J].Nucleic Acids Research,2022,50(D1):D785-D794.
[30] ILLEGHEMS K,DE VUYST L,WECKX S.Comparative genome analysis of the candidate functional starter culture strains Lactobacillus fermentum 222 and Lactobacillus plantarum 80 for controlled cocoa bean fermentation processes [J].BMC Genomics,2015,16:1-13.
[31] SUN Z,HARRIS H M,MCCANN A,et al.Expanding the biotechnology potential of Lactobacilli through comparative genomics of 213 strains and associated genera[J].Nature Communications,2015,6(1):8322.
[32] MORITA H,TOH H,FUKUDA S,et al.Comparative genome analysis of Lactobacillus reuteri and Lactobacillus fermentum reveal a genomic island for reuterin and cobalamin production [J].DNA Research,2008,15(3):151-161.
[33] FONTANA A,ZACCONI C,MORELLI L.Genetic signatures of dairy Lactobacillus casei group [J].Frontiers in Microbiology,2018,9:2611.
[34] TANG H,BOWERS J E,WANG X,et al.Synteny and collinearity in plant genomes[J].Science,2008,320(5875):486-488.
[35] 邢锋.籼稻珍汕97 和明恢63 基因组的注释和比较分析[D].武汉:华中农业大学,2016.XING F.Annotation and comparative analysis of the genomes of indica rice Zhenshan 97 and Mingcui 63[D].Wuhan:Huazhong Agricultural University,2016.
[36] PAPIZADEH M,ROHANI M,NAHREVANIAN H,et al.Probiotic characters of Bifidobacterium and Lactobacillus are a result of the ongoing gene acquisition and genome minimization evolutionary trends[J].Microbial Pathogenesis,2017,111:118-131.
[37] PAPADIMITRIOU K,ANASTASIOU R,MAISTROU E,et al.Acquisition through horizontal gene transfer of plasmid pSMA198 by Streptococcus macedonicus ACA-DC 198 points towards the dairy origin of the species[J].PLoS One,2015,10(1):e0116337.
[38] 刘超楠,蔡熙姮,张新宇,等.植物乳杆菌基因组学研究进展[J].食品科技,2020,45(11):1-7.LIU C N,CAI X H,ZHANG X Y,et al.Advances in Lactobacillus plantarum genomics research[J].Food Science and Technology,2020,45(11):1-7.
[39] HAMMAMI R,ZOUHIR A,LAY C L,et al.Database mining for bacteriocin discovery[M].Wallingford:Natural Antimicrobials in Food Safety and Quality,2011:349-358.
[40] CARSON D A,BARKEMA H W,NAUSHAD S,et al.Bacteriocins of non-aureus Staphylococci isolated from bovine milk[J].Applied and Environmental Microbiology,2017,83(17):e01015-17.
[41] 郭行.鼠李糖乳杆菌LS-8 中新型细菌素的挖掘及抑菌机制的研究[D].杨凌:西北农林科技大学,2020.GUO X.Excavation of novel bacteriocins in Lactobacillus rhamnosus LS-8 and study of the mechanism of bacterial inhibition[D].Yangling:Northwest A&F University,2020.
[42] 瞿茜楠,彭传涛,黄天,等.副干酪乳酪杆菌PC-724 蛋白类抑菌活性代谢产物的研究[J/OL].中国食品学报,(2023-04-08)[2023-06-25].https://kns.cnki.net/kcms2/article/abstract?v=3uoqIhG8C45S0n9fL2su RadTyEVl2pW9UrhTDCdPD64bFQGXWSZy43nINXKTrk6hwwzsJstBa7_O2OZUKYI -Es4zHMm4fNUh&uniplatform=NZKPT.QU Q N,PENG C T,HUANG T,et al.Study of the protein-like antibacterial activity metabolites of Lactobacillus paracasei PC-724[J/OL].Journal of Chinese Institute of Food Science and Technology,(2023 -04 -08)[2023 -06 -25].https://kns.cnki.net/kcms2/article/abstract?v=3uoqIhG8C45S0n9fL2suRad-TyEVl2pW9UrhTDCdPD64bFQGXWSZy43nINXKTrk6hwwzsJstBa7_O2OZUKYI -Es4zHMm4fNUh&uniplatform=NZKPT.
[43] QU H,XIN Y,DONG X,et al.An rmlA gene encoding D -glucose -1 -phosphate thymidylyltransferase is essential for mycobacterial growth[J].FEMS Microbiology Letters,2007,275(2):237-243.
[44] 丛峰松.神奇的小分子活性肽[M].上海:上海交通大学出版社,2015:18.CONG F S.Miraculous small molecule active peptide[M].Shanghai:Shanghai Jiao Tong University Press,2015:18.
[45] CÁRDENAS N,LAIÑO J E,DELGADO S,et al.Relationships between the genome and some phenotypical properties of Lactobacillus fermentum CECT 5716,a probiotic strain isolated from human milk[J].Applied Microbiology and Biotechnology,2015,99:4343-4353.