生物活性肽是一种蛋白质降解成氨基酸的中间产物,它除了具有营养价值外,还会对机体健康产生有益的生理作用,与传统药物相比,它无毒、副作用小且利于机体利用,目前许多生物活性肽已被应用于医学领域[1]。生物活性肽具有多种生物活性,主要包括抗氧化、抗菌、降血压及降血糖等[2]。目前研究报道,生物活性肽主要来源于乳制品、动物和植物,这是因为它们含有特定生物活性片段的蛋白质前体[3]。乳源蛋白质被视为生物活性肽的重要来源,目前越来越多的生物活性肽在乳蛋白水解物和发酵乳制品中被发现。乳清蛋白来源于奶酪生产加工中的副产物乳清中,蛋白组成包括β-乳球蛋白、α-乳白蛋白、免疫球蛋白、血清白蛋白、乳铁蛋白、乳过氧化物酶、蛋白胨和糖巨肽,含有大量必需氨基酸和支链氨基酸,如异亮氨酸、亮氨酸和缬氨酸,具有调节不同代谢通路、血糖稳态及含硫氨基酸平衡的功能[4]。它本身也具有优良的抗氧化性,可清除机体自由基,螯合金属离子及修复体内蛋白质中的游离巯基[5]。研究表明,乳清蛋白在水解过程中会释放出具有高抗氧化能力的抗氧化肽[6-7]。Peng 等[8]发现,与未水解的乳清分离蛋白相比,乳清分离蛋白的水解物具有更强的自由基清除能力、铜离子螯合能力和总还原能力。Daliri 等[9]研究表明,乳清蛋白经Corolase 酶或其它商业蛋白酶水解后得到的乳清蛋白肽具有较高的清除自由基能力。Corrochano 等[10]通过模拟胃肠道消化的方式研究乳清蛋白源抗氧化肽,发现乳清蛋白经消化后产生的多肽(ALMP、GDLE、TKIPA、VEELKPT、VGIN 和AVEGPK)通过Caco-2/HT-29 细胞中的肠屏障传输,从而抑制肌肉和肝细胞中自由基的形成。
人体产生氧自由基含量的多少,受机体内部和外部环境影响,比如自然生长发育的过程、外部环境接触的过程及不良饮食习惯的影响。在代谢正常的条件下,机体产生的自由基会被其内部的抗氧化系统中和,如果机体代谢紊乱就会导致自由基和抗氧化系统之间的平衡紊乱,从而导致细胞内积累过量的自由基。氧自由基在生物体内过量往往与活性氧种类和抗氧化物质之间的不平衡有关,这种自由基的平衡紊乱称为氧化应激[11]。自由基反应的加剧会引起强烈且持久的氧化应激,并可能导致DNA、蛋白质和脂质结构的永久性变化,这个过程会破坏细胞结构和基因,从而导致代谢紊乱和肿瘤转化[12]。研究表明,许多现代非传染性疾病与氧化应激有关,如肿瘤、神经退行性疾病、循环系统及神经系统的紊乱[13]。虽然合成药物可用于治疗这些疾病,但是其往往伴随有不良副作用,因此天然食物来源的抗氧化成分受到科学界的广泛关注。
传统的抗氧化肽筛选方法是通过特定酶对蛋白进行水解,然后对产生的肽段进行鉴定、合成,并进行体外和体内的抗氧化验证试验,因此抗氧化肽序列的试验研究往往既价格昂贵又耗费时间[14]。采用生物信息学对肽段进行电子分析来筛选抗氧化肽序列,可以克服这些缺点,对抗氧化肽的研究主要包括电子预测和分子模拟技术。生物活性肽的电子预测是基于机器学习方法创立的,在机器学习推理中通常需要正数据集和负数据集来训练预测模型,通过这种方式可以训练机器学习算法,从给定数据集中学习复杂的基本模式,并利用它们的物理化学性质和氨基酸组成预测生物活性肽的生物活性[15]。分子模拟技术是从生命科学中蛋白质、多肽等结构出发,在分子水平上解析生物反应过程,在分子作用机理上补充体外和体内试验的理论,由于其可对模拟粒子进行原子细节分析,因此在分子相互作用方面提高了对试验数据的理解和解释的新水平[16-17]。
综上所述,本研究利用虚拟酶解、电子预测、分子对接及分子动力学等生物信息学方法,以牛乳清蛋白为目标蛋白筛选抗氧化肽。旨在为筛选乳清蛋白源抗氧化肽提供一种高效、低成本的方法,为天然食源性抗氧化肽的快速筛选提供理论依据。
1 材料与方法
1.1 材料与试剂
牛乳清蛋白氨基酸序列,来自Universal Protein数据库(https://www.uniprot.org/);Keap1 蛋白的天然晶体结构,来自RCSB 蛋白质数据库(https://www.rcsb.org/)。
1.2 仪器与设备
Discovery studio(DS)V2019 软件,美国BIOVIA 公司。
1.3 牛乳清蛋白序列的获得
牛乳清蛋白主要包括β-乳球蛋白(1.3 g/L)、α-乳白蛋白(1.2 g/L)、免疫球蛋白(0.5~1.0 g/L)、牛血清白蛋白(0.4 g/L)、牛乳铁蛋白(0.1 g/L)[18],其蛋白序列来源于Universal Protein(https://www.uniprot.org/),5 种蛋白的编号和氨基酸数目分别为:Beta-lactoglobulin,P02754,178;Alpha-lactalbumin,P00711,142;Polymeric immunoglobulin receptor,P81265,757;Albumin,P02769,607 和Lactotransferrin,P24627,708。
1.4 计算机虚拟消化
采用BIOPEP-UWM(http://www.uwm.edu.pl/biochemia/index.php/pl/biopep)[19]中 的“enzyme(s)action”程序对5 种牛乳清蛋白源蛋白质进行虚拟消化,使用的水解酶为胃蛋白酶(EC 3.4.23.1)和胰蛋白酶(EC 3.4.21.4),识别和切割位点分别为:(胃蛋白酶)、(胰蛋白酶),且2 种蛋白酶均可作用于C-末端。
1.5 抗氧化肽的筛选
将虚拟消化得到的牛乳清蛋白源肽段在PeptideRanker(http://distilldeep.ucd.ie/PeptideRanker/)预测网站中进行潜在生物活性评分,生物活性评分越高代表预测肽的生物活性越大,通常选择不小于0.5 或0.4 作为筛选生物活性肽的基准[20]。本研究为了扩大筛选抗氧化肽的范围,因此选择不小于0.4 的生物活性评分的肽作为后续研究对象。
通过BIOPEP-UWM 和SATPdb[21](http://crdd.osdd.net/raghava/satpdb/)数据库,将上述筛选后的所有肽段与两数据库中已被报道的生物活性肽进行比对,筛选出未被报道过的肽进行后续研究。
1.6 毒性、过敏性及水溶性分析
通过ToxinPred[22]程序(http://crdd.osdd.net/raghava/toxinpred/)预测肽的毒性,设定SVM 阈值为0.0,采用两种预测方法:SVM(Swiss Prot)+Motif 和SVM(TrEMBL)+Motif 进行测定,只有当2 两种方法预测结果均为无毒时才可被视为无毒的肽序列;通过AllerTOP v.2.0 [23]程序(https://www.ddg-pharmfac.net/AllerTOP/index.html)预测肽的过敏性;通过Innovagen 在线网站(http://www.innovagen.com)预测肽的水溶性优劣,此网站可以将肽区分为“Good water solubility”和“Poor water solubility”。筛选出预测结果为无毒、无致敏性、良好水溶性的肽用于后续研究。
1.7 物理化学性质分析
通过Expasy-compute[24]程序(http://web.expasy.org/compute_pi/)的pI/Mw tool 功能预测肽的相对分子质量和等电点;通过在线网站Pepdraw(http://www.tulane.edu/~biochem/WW/PepDraw)预测肽的净电荷和疏水性。
1.8 药物代谢动力学分析
通过Discovery studio(DS)V2019(Dassault Systemes Biovia,San Diego,CA,USA)软件对多肽进行药物代谢动力学(Absorption distribution metabolism excretion toxicity,ADMET)分析,分析多肽作为相关药物的吸收与毒性,进一步筛选更加合理、健康的肽段。采用DS 软件的ADMET Descriptors 程序,分析多肽的人体肠道吸收(Human intestinal absorption,HIA)[25]、25 ℃水溶性(Aqueous solubility)[26]、血脑屏障通透性(Blood brain barrier,BBB)[27]、细胞色素P450 2D6 抑制性(Cytochrome P4502D6 inhibition)[28-29]、肝毒性(Hepatotoxicity)[29-30]及血浆蛋白结合率(Plasma protein binding,PPB)[31];采用DS 软件的Toxicity Prediction 程序对多肽进行毒理性质的预测,依据乳清蛋白源多肽的应用功能选定9 个预测模型:1)埃姆斯致突变性模型、2)NTP 数据集下的啮齿动物致癌性模型(NTP Rodent Carcinogenicity)、3)FDA 数据集下的啮齿动物致癌性模型(FDA Rodent Carcinogenicity)、4)啮齿动物致癌性证据权重(Weight-of-evidence rodent carcinogenicity)、5)致癌潜能TD50(Carcinogenic potency TD50)、6)潜在发育毒性(Developmental toxicity potential)、7)大鼠口服LD50(Rat oral LD50)、8)大鼠最大耐受剂量(Rat maximum tolerated dose)、9)大鼠长期口服最低毒副反应水平(Rat chronic oral lowest observed adverse effectlevel,LOAEL)。
1.9 分子对接
1.9.1 配体结构优化 使用DS 软件的“Macromolecules” 模块下的 “Build and Edit Protein”工具,将筛选出的多肽序列按照特定顺序输入到程序中;利用“Small Molecules”模块下的“Minimize Ligands”和“Prepare Ligands”工具对肽的结构在CHARMm 力场下进行能量优化。
1.9.2 准备受体蛋白 从RCSB 蛋白质数据库(https://www.rcsb.org/)获取Keap1 蛋白的天然晶体结构(PDB 代码:2FLU)。使用DS 软件打开PDB文件,删除水分子、氢原子及原有受体所携带的配体,并对蛋白进行加氢处理,再使用“Macromolecules”模块下的“Clean Protein”工具除去蛋白多构象,使Keap1 蛋白补充完整的氨基酸残基。通过DS 软件的“Receptor-Ligand Interactions”模块下的“Define and Edit Binding site”工具定义受体靶标活性位点,活性中心坐标(X:5;Y:9;Z:1),对接半径为15Å[32]。
1.9.3 半柔性分子对接 使用DS 软件的“Receptor-Ligand Interactions” 模块下的“Dock Ligands(CDOCKER)”,在“Input Ligands”栏选择全部优化后的配体,其余参数默认不变。将受体定义为刚性,配体定义为半柔性,进行半柔性分子对接,根据“-CDOCKER Energey”值评定分子对接效果。
1.10 分子动力学模拟
使用DS 软件的“Simulation”模块进行分子动力学模拟,将最终所筛选出的CDEF 肽序列-Keap1 最优结合复合物结构在“Macromolecules”模块下的 “Prepare protein” 进行蛋白质预处理,在“Simulation”模块下的“Apply forcefield”为蛋白复合物添加charmm36 力场,用“Solvation”对其进行溶剂化处理,在“Run simulation”中的“Standard Dynamics Cascade”进行分子动力学模拟,参数设置如下:升温阶段由50 K 逐渐加热至300 K,模拟加热时间4 ps,平衡阶段模拟时间设置为20 ps,模拟采样阶段设置为0.2 ns[33]。
2 结果与分析
2.1 牛乳清蛋白的虚拟酶解及抗氧化肽的筛选
为了在人体内发挥生物效应,生物活性肽必须经过胃肠道消化,然后在胃肠道吸收后以活性分子形式到达其目标部位[34]。因此本研究将牛乳清蛋白源的主要5 种蛋白成分用胃蛋白酶(EC 3.4.23.1)和胰蛋白酶(EC 3.4.21.4)虚拟消化,通过虚拟消化程序,计算出β-乳球蛋白、α-乳白蛋白、免疫球蛋白、牛血清白蛋白及牛乳铁蛋白的水解度分别为28.25%,24.82%,21.43%,29.87%及27.16%,结果表明牛血清白蛋白被两种酶水解程度最大,可能会消化产生相对更多的肽段。然后将消化后得到的多肽用PeptideRanker 程序进行生物活性肽评分预测,并将PeptideRanker 评分>0.4的生物活性肽筛选出来,最终与BIOPEP-UWM 和SATPdb 数据库中已报道的活性肽序列进行对比,具体筛选评分结果见表1。
表1 虚拟酶解及数据库分析得到的肽
Table 1 Peptides obtained by virtual enzymatic hydrolysis and database analysis

(续表1)

注:a.已被报道的具有抗氧化性质的肽;b.已被报道的具有降血压性质的肽;c.已被报道的具有降血糖性质的肽。
研究表明抗氧化肽具有以下特征:1)抗氧化肽一般含有0~20 个氨基酸;2)肽序列中含有Pro、Gly、Ala、Val 和Leu 的蛋白肽具有潜在的抗氧化活性;3)带有芳香环(Tyr、Trp、Phe)、咪唑基(His)和含硫基(Met、Cys)的肽链直接淬灭自由基[35]。由表1 可知,5 种蛋白质(β-乳球蛋白、α-乳白蛋白、免疫球蛋白、牛血清白蛋白及牛乳铁蛋白)分别筛选出11,8,38,36,44 条生物活性评分大于0.4 的肽段,一共137 条多肽,大多数筛选出的多肽符合抗氧化肽的特征性。接下来对比BIOPEP-UWM 和SATPdb 数据库,对未报道过的多肽进行毒性、过敏性及水溶性分析,通过3 项评价进一步筛选出无毒、无过敏性及水溶性良好的抗氧化肽,详细结果见表2。
表2 肽的毒性、过敏性及水溶性分析
Table 2 Toxicity,allergy and water solubility analysis of peptides

(续表2)

(续表2)

采用ToxinPred 程序对多肽进行毒性预测,结果显示绝大多数肽在此程序设定的2 种模型下表现为无毒。乳蛋白的过敏性是乳制品中不可忽视的重要因素,乳清蛋白中β-乳球蛋白和α-乳白蛋白是除酪蛋白外的婴儿牛乳过敏症的主要致敏因子[36],因此需要对乳清蛋白源抗氧化肽进行过敏性预测。通过AllerTOP v.2.0 程序预测肽的过敏性,结果显示大约50%的的多肽被预测为具有过敏性,说明牛乳清蛋白在经过胃肠道消化后依然会有许多过敏性多肽产生。多肽的水溶性是其在机体内转运分布的基础,也是应用食品、生物及材料化学研究时需要考虑的重要指标[37]。通过Innovagen 网站预测肽的水溶性,结果显示有近一半多肽的水溶性被判定为差。综合分析毒性、过敏性及水溶性3 项性质,可以发现α-乳白蛋白源的多肽没有同时符合无毒、无过敏性及水溶性良好的抗氧化肽,而β-乳球蛋白源的多肽只有PMHIR和DAQSAPL 序列同时符合这3 个条件。将同时符合3 个条件下的多肽进行相关物理化学分析,详细结果见表3。
表3 肽的分子质量、等电点及pH=7 时的净电荷分析
Table 3 Molecular weight,isoelectric point and net charge analysis at pH=7 of peptide

注:“-”表示没有同时符合无毒、无过敏性及水溶性良好的抗氧化肽,故不作后续分析。
2.2 药物代谢动力学分析
药物代谢动力学是一门传统上的生命科学学科,可以研究药物或候选药物在药理过程中的可用性,并表征其进入机体体内吸收、转移及代谢的生物转化过程[38]。研究多肽的药物代谢动力学行为可有效地指导多肽的结构优化与系统筛选,对药物的深入开发及应用具有重大意义[39]。
2.2.1 多肽的ADMET 分析 ADMET 是一种基于机器学习的有助于预测和筛选良好候选药物的方法,可以减少对药物进行传统试验方法的筛选,同时减少应用于临床治疗时的失败[40-41]。多肽的ADMET 分析如表4、5 所示,所有乳清蛋白源的多肽序列的人体肠道吸收(HIA)均很差,这是因为本研究选用的是生物体胃肠道中存在的胃蛋白酶和胰蛋白酶,获得的抗氧化肽会抵御胃肠道的消化吸收,这并不意味着其抗氧化能力无法在机体中表现出来,一些抗氧化肽的抗氧化作用的表达往往需要能够抵御胃肠道的消化作用[42]。除了免疫球蛋白源的YWCK 和牛血清白蛋白源的QNCDQF 肽序列在25 ℃水溶性表现为非常高,其余多肽在25 ℃水溶性都表现为极高,这说明所有多肽都具有良好的水溶性,同时也印证了前文通过Innovagen 网站预测肽的水溶性的准确性。多肽的血脑屏障通透性(BBB)均表现为未定义,通过打分数字的命名规律以及肠道吸收率可以预测出其对血脑屏障的穿透性也很低[43]。所有多肽均无细胞色素P450 2D6 抑制性。除β-乳球蛋白源的DAQSAPL、免疫球蛋白源的CPR 及牛乳铁蛋白源的CAGDDQGL、AAPR 肽序列有肝毒性,其余多肽均无肝毒性。血浆蛋白与药物分子的结合会影响药物的效率,因为结合部分会暂时避免代谢,所有多肽的血浆蛋白结合率(PPB)均小于90%,表明其均不会影响药物结合的效率。
表4 Admet 评分参考表
Table 4 Reference table of ADMET score

表5 肽的ADMET 分析
Table 5 ADMET analysis of peptides
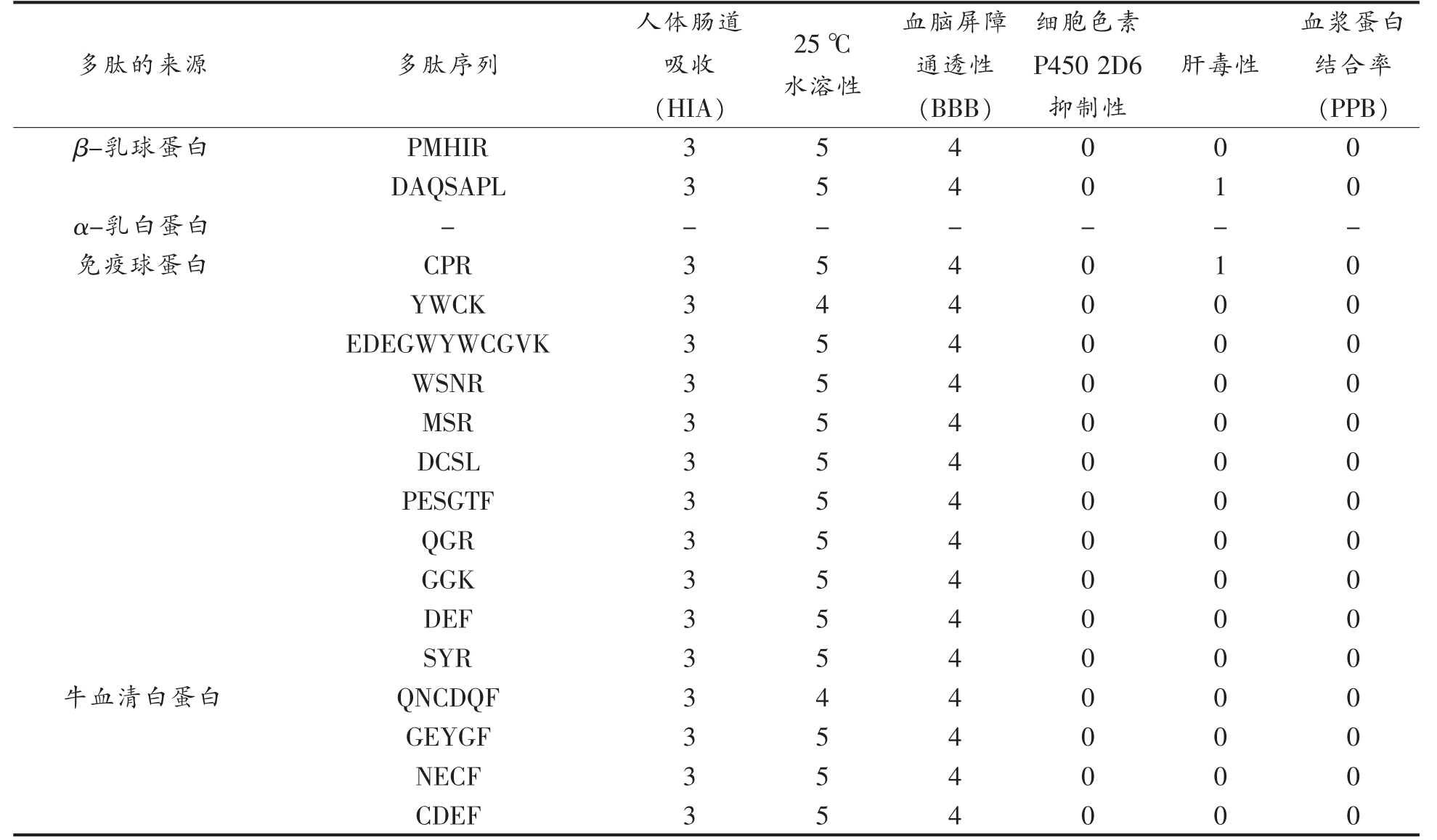
(续表5)
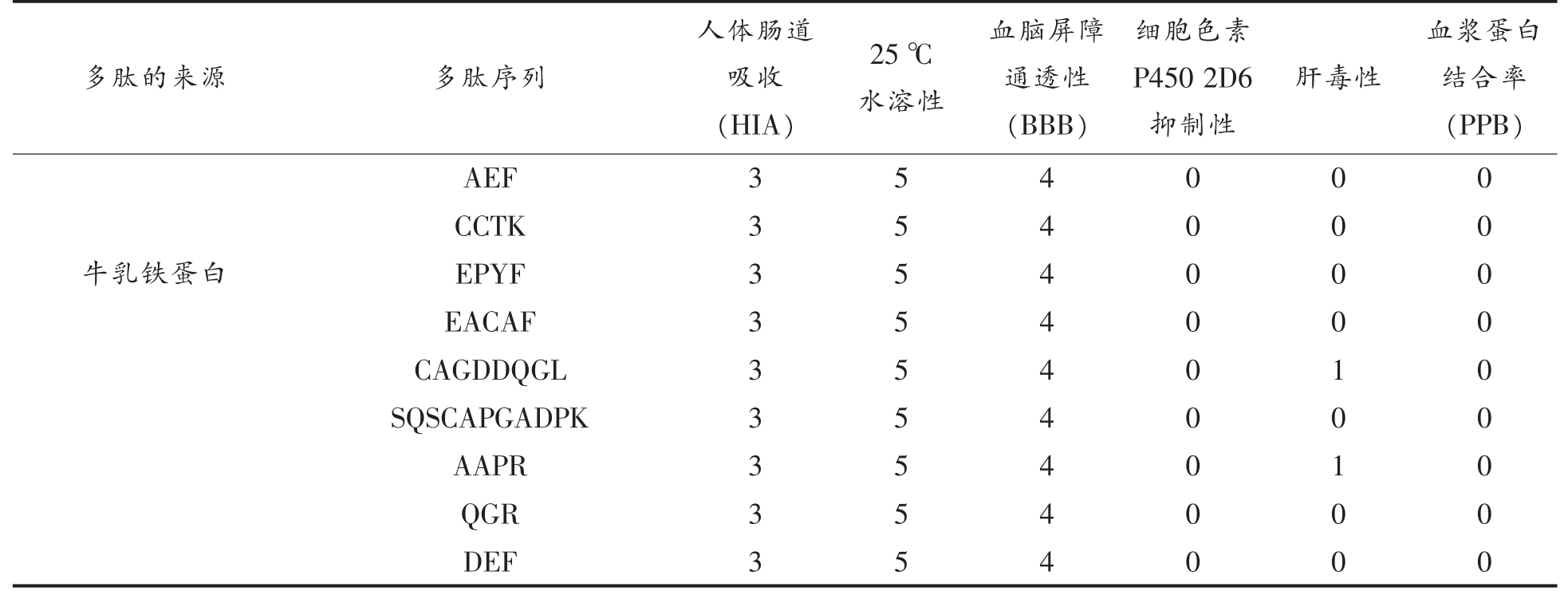
注:“-”表示没有同时符合无毒、无过敏性及水溶性良好的抗氧化肽,故不作后续分析;表5 数值含义参考表4 释义。
2.2.2 多肽的TOPKAT 分析 TOPKAT 是根据其二维分子结构来准确、快速地评估化学品毒性的预测手段,DS 软件中使用了一系列稳定的、交叉验证的、定量的结构-毒性关系(QSTR)模型来评估化合物特定的毒理学性质。肽的Topkat 毒性分析详情见表6、7。
表6 Topkat 评分参考表
Table 6 Topkat scoring reference table

表7 肽的Topkat 毒性分析
Table 7 Topkat toxicity analysis of peptides

注:“-”表示没有同时符合无毒、无过敏性及水溶性良好的抗氧化肽,故不作后续分析;表7 数值含义参考表6 释义。
埃姆斯致突变性模型是根据美国环保署基因毒素协议检测的化合物开发出来的,结果显示所有多肽序列均无致突变性。美国国家毒理学计划(National toxicology program,NTP)和美国食品药品监督管理局(Food and Drug Administration,FDA)数据集下的啮齿动物致癌性模型均是具有统计学意义和交叉验证的定量结构-毒性关系(QSTR)模型,结果显示所有多肽在NTP 数据集下的啮齿动物致癌性模型(包含雌性大小鼠和雄性大小鼠)中均无致癌性;在FDA 数据集下的啮齿动物致癌性模型中,β-乳球蛋白源的PMHIR 肽序列在雌性小鼠模型中表现为单剂量致癌性,在雄性小鼠模型中表现为多剂量致癌性,其次免疫球蛋白源的CPR 和YWCK、牛乳铁蛋白源SQSCAPGADPK 和AAPR 肽序列在雌性大鼠模型中表现为单剂量致癌性,而免疫球蛋白源(牛乳铁蛋白源)的QGR 和GGK、牛血清白蛋白源的CCTK 及牛乳铁蛋白源的肽序列在雄性小鼠模型中表现为多剂量致癌性,在雌性大鼠模型中表现为单剂量致癌性,最后牛乳铁蛋白源的CAGDDQGL 肽序列在雄性小鼠模型中表现为多剂量致癌性。啮齿动物致癌性证据权重模型是使用美国食品和药物管理局药物评估和研究中心证据权重协议所制定的QSTR 模型,结果显示所有多肽在此模型下均无致癌性。潜在发育毒性模型是来源于374 篇开放文献中开发得到的,此模型仅包括大鼠口服的数据,结果显示免疫球蛋白源(牛乳铁蛋白源)的QGR、牛血清白蛋白源的GEYGF 在此模型下表现为有毒,其余多肽均表现为无毒。
DS 软件中关于大鼠耐受剂量预测主要包括致癌潜能TD50、大鼠口服LD50、大鼠最大耐受剂量及大鼠长期口服最低毒副反应水平,耐受剂量多少可为后续多肽应用于食品、生物及医疗方面提供理论数据支持,详细结果见表8。
表8 Topkat 预测相关大鼠耐受剂量表
Table 8 Tolerance dose table of Topkat prediction related rats
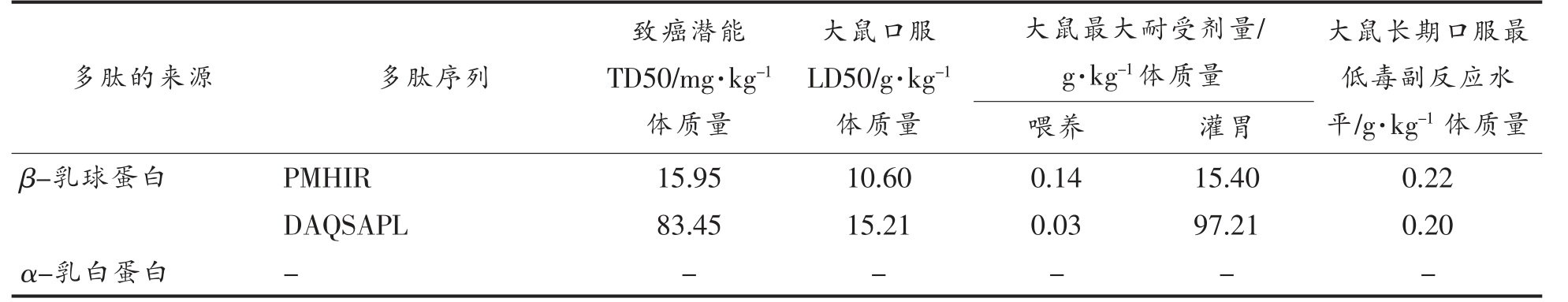
(续表8)

注:“-”表示没有同时符合无毒、无过敏性及水溶性良好的抗氧化肽,故不作后续分析。
综合分析以上药物代谢动力学结果,将在所有ADMET 和TOPKAT 数据模型下无毒、无致突变性及无致癌性的多肽序列筛选出来用于分子对接分析,可以发现β-乳球蛋白源的序列在此环节中无符合全部条件的多肽。
2.3 分子对接
在生物活性肽研究领域,分子对接可以基于结构构象表征肽在受体蛋白结合位点的行为,分子对接过程包括预测受体内配体的分子方向,然后使用结合亲和力评分函数计算受体-配体的互补相互作用[44-45]。虽然蛋白质水解物和肽的抗氧化作用的潜在机制尚未完全研究清楚,但据报道Keap1-Nrf2 通路是消除体内氧化应激的最可能机制,在Keap1-Nrf2 通路中,Nrf2 因子在迁移到细胞核后可与抗氧化反应元件ARE 序列结合,并启动血红素加氧酶1 的表达,从而激活抵抗氧化应激的防御系统[46]。当机体内细胞处于氧化应激条件下,外来的抗氧化剂会干扰Keap1-Nrf2 相互作用,从而最终激活ARE 序列调控细胞内一系列具有抗氧化功能的酶(如过氧化氢酶、超氧化物歧化酶和谷胱甘肽过氧化物酶等)的基因表达,从而促进机体的抗氧化能力[47]。为了进一步筛选乳清蛋白源抗氧化肽,并解释抗氧化肽的在Keap1-Nrf2抗氧化通路的分子机制,对乳清蛋白源抗氧化肽进行分子对接分析。准备好的受体蛋白见图1 所示。

图1 原始蛋白(a)及准备好的受体蛋白(b)
Fig. 1 Original protein(a)and prepared receptor protein(b)
2.3.1 分子对接评分 乳清蛋白源生物活性肽可以通过分子对接的方式研究其生物活性,如Li等[48] 借助分子对接研究了乳清蛋白水解物中的4种多肽(PQVSTPTL、MPGP、PMHIR、PPLT)的ACE抑制机理,结果显示乳清蛋白水解物可能是获得新型ACE 抑制肽的合适来源。对接结果中“-CDOCKER Energy” 值是受体与配体相互作用力或结合亲和力的评分,其值相对越高,表示受体与配体结合的越紧密、越容易[49]。本研究通过将前步筛选得到的13 条乳清蛋白源抗氧化肽与Keap1 受体进行分子对接,并选择文献中报道过的乳清蛋白源抗氧化肽WYSL[50]作为阳性对照也与Keap1 受体进行分子对接,具体对接结果见表9。
表9 抗氧化肽与Keap1 的分子对接评分
Table 9 Molecular docking scores of antioxidant peptides and Keap1
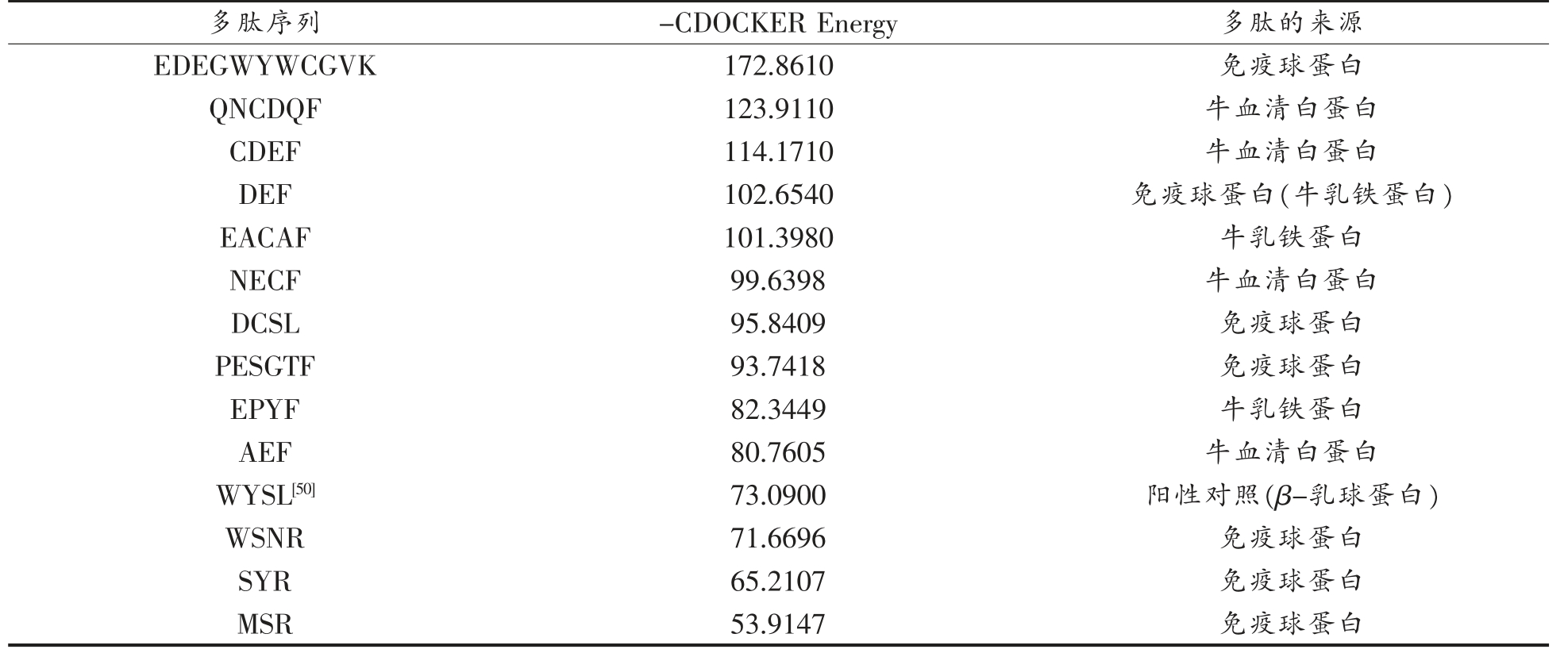
分子对接结果显示,相较阳性对照抗氧化肽WYSL,本研究筛选出的乳清蛋白源抗氧化肽的“-CDOCKER Energy”大多数高于WYSL 序列,说明筛选出的抗氧化肽能够较好地与Keap1 蛋白结合从而发挥抗氧化性,同时也印证了本研究前面筛选步骤的真实性与可靠性。选取“-CDOCKER Energy” 值排名前4 位的多肽以及阳性对照(WYSL 序列)与Keap1 受体进行相互作用分析。
2.3.2 分子对接相互作用分析 Keap1 的分子突变试验证明,ETGE 基序是与Keap1 的Kelch 结构域结合时关键基序[51]。研究表明,接触ETGE 基序相互作用的残基分别是3 个Arg 残基(Arg380、Arg415 和Arg483)、4 个Ser 残基(Ser363、Ser508、Ser555 和Ser602),以及Kelch 结构域中Tyr334、Asn382 和Gln530 的残基,此外Asn387、His436、Tyr525、Tyr572 和Phe577 的残基在Keap1-Nrf2蛋白质-蛋白质相互作用中也起着关键作用[52]。图2~6 表示的是阳性对照(WYSL 序列)、选定的4 个多肽分别与Keap1 活性位点分子对接相互作用的结果图(包括3D 和2D 模式图)。阳性对照(WYSL序列)为已报道过的乳清蛋白源抗氧化肽,在体外试验验证中表现出良好的抗氧化性[50]。图2 对接结果显示WYSL 序列与Keap1 分子的氨基酸残基Arg415 和Arg483 之间形成盐桥和电荷吸引力,与氨基酸Ser508、Arg483 之间形成常规氢键,与氨基酸残基Tyr572 之间形成碳氢键和π-供体氢键,与氨基酸残基Phe577、Tyr572 之间形成π-π相互作用,与氨基酸残基Tyr525、Tyr572 之间疏水相互作用,与氨基酸残基Tyr334、Arg380、Asn414、Ile461、Phe478、Ala556、Ser555、Gln530 之间形成范德华力。其中9 个氨基酸残基Arg415、Arg483、Ser508、Tyr572、Tyr525、Tyr334、Arg380、Ser555、Gln530 是影响Keap1-Nrf2 相互作用的关键残基,因此阳性对照(WYSL 序列)具有在体内发挥抗氧化性的潜力,验证了前人的结论。图3 对接结果显示EDEGWYWCGVK 序列与Keap1 分子的氨基酸残基Arg483、Arg380、Arg415 之间形成盐桥、电荷吸引力及π-阳离子作用,与氨基酸残基Arg415、Asn414 之间形成常规氢键,与氨基酸残基Tyr334、Gly462、Gly511、Leu557、Ala366、Val418、Gly603 之间形成碳氢键和π-供体氢键,与氨基酸残基 Gly574、Tyr572、Asn382、Phe577、Ser363、Gly509、Ile416、Ala510、Val463、Gly464、Gly558、Gly417、Gln530、Tyr525、Ser555、Ser602、Gly364、Ala556、Leu365、Gly605、Ile461、Ile559、Val604、Val606、Val512、Val465 之间形成范德华力。其中11 个氨基酸残基Arg483、Arg380、Tyr334、Tyr572、Asn382、Phe577、Ser363、Gln530、Tyr525、Ser555、Ser602 是影响Keap1-Nrf2 相互作用的关键残基,说明EDEGWYWCGVK 序列也具有在体内发挥抗氧化性的潜力。图4 对接结果显示QNCDQF 序列与Keap1 分子的氨基酸残基Arg483、Arg415 之间形成盐桥,与氨基酸残基Arg380、Gln530 形成常规氢键,与氨基酸残基Tyr334 形成π-π 相互作用,而同时也与氨基酸残基Tyr334 之间形成不利的供体-供体相互作用,与氨基酸残基Asn382、Ser602、Ser363、Gly603、Gly364、Tyr572、Phe577、Tyr525、Ala556、Asn414、Ser555 之间形成范德华力。其中11 个氨基酸残基Arg483、Arg415、Arg380、Tyr334、Asn382、Ser602、Ser363、Tyr572、Phe577、Tyr525、Ser555 是影响Keap1-Nrf2 相互作用的关键残基,说明QNCDQF 序列也具有在体内发挥抗氧化性的潜力。图5 对接结果显示CDEF 序列与Keap1 分子的氨基酸残基Arg483、Arg415、Arg380 之间形成盐桥、电荷吸引力及π-阴离子作用,氨基酸残基Ser508、Asn414、Tyr334、Asn382、Arg483、Arg380 之间形成常规氢键,与氨基酸残基Arg415 之间形成碳氢键,而与氨基酸残基Arg415 之间形成不利的阳性-阳性相互作用,与氨基酸残基Tyr525 之间形成π-π 相互作用,与氨基酸残基Gln530、Ser555、Gly509、Ile461、Ala556、Ser602、Ser363、Gly364、Gly603、Tyr572 之间形成范德华力。其中12 个氨基酸残基Arg483、Arg415、Arg380、Ser508、Tyr334、Tyr334、Tyr525、Gln530、Ser555、Ser602、Ser363、Tyr572 是影响Keap1-Nrf2 相互作用的关键残基,说明CDEF 序列也具有在体内发挥抗氧化性的潜力。图6 对接结果显示DEF 序列与Keap1 分子的氨基酸残基Arg483、Arg415、Arg380、Tyr334 之间形成盐桥和电荷吸引力,与氨基酸残基Ser508、Arg415、Asn414、Arg380、Asn382、Tyr334 之间形成常规氢键,与氨基酸残基Arg415 之间形成碳氢键,而与氨基酸Arg380 之间形成不利的阳性-阳性相互作用,与氨基酸残基Tyr572 之间形成π-π 相互作用,与氨基酸残基Ala556 之间形成疏水相互作用,与氨基酸残基Phe577、Ser602、Ser555、Tyr525、Gln530、Ile461 之间形成范德华力。其中11 个氨基酸残基Arg483、Arg415、Arg380、Tyr334、Ser508、Tyr334、Phe577、Ser602、Ser555、Tyr525、Gln530 是影响Keap1-Nrf2 相互作用的关键残基,说明DEF序列也具有在体内发挥抗氧化性的潜力。
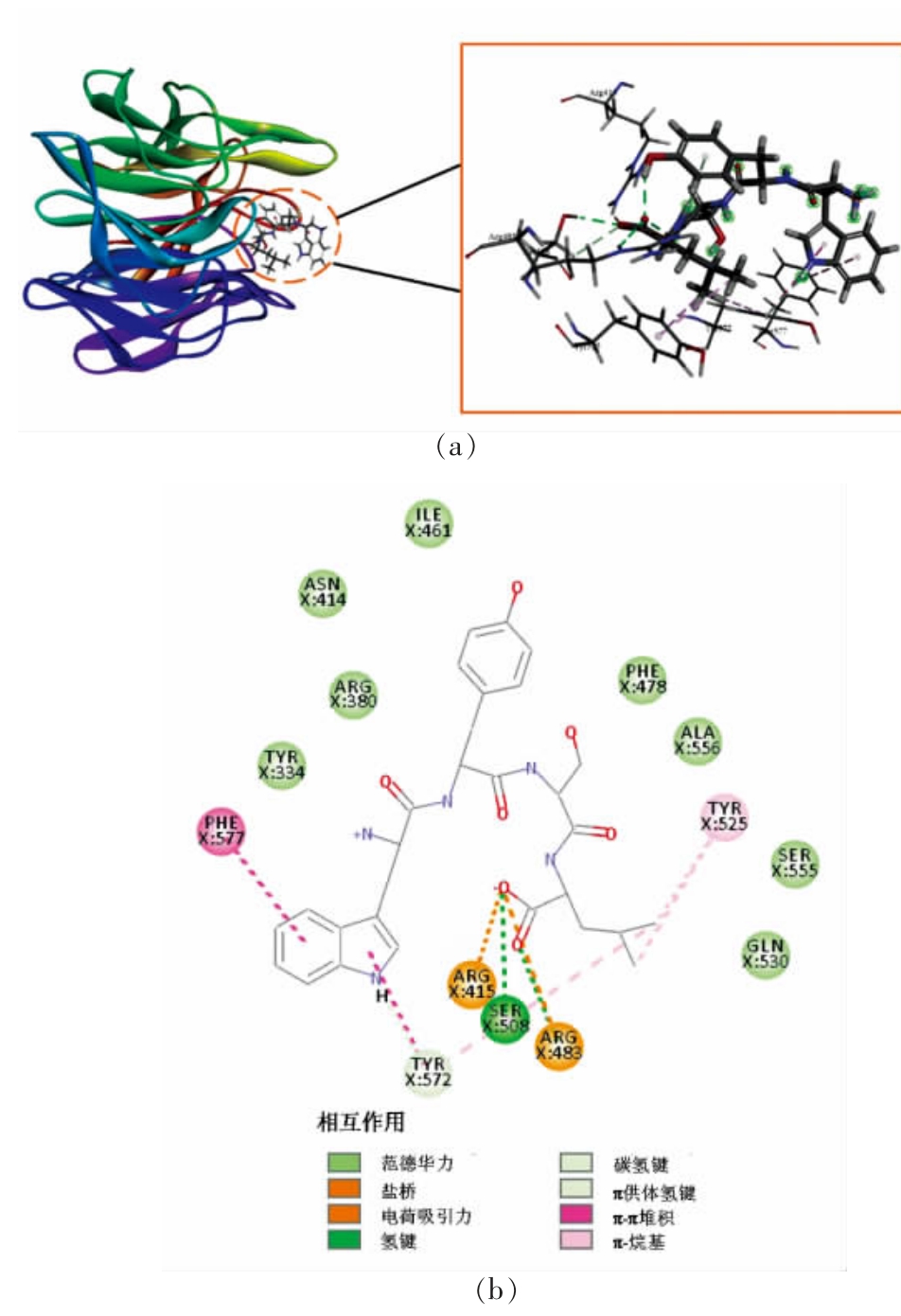
图2 阳性对照(WYSL 序列)与Keap1 活性位点相互作用的3D(a)和2D(b)模式图
Fig. 2 3D(a)and 2D(b)pattern of interaction between positive control(WYSL sequence)and Keap1 active site

图3 EDEGWYWCGVK 与Keap1 活性位点相互作用的3D(a)和2D(b)模式图
Fig. 3 3D(a)and 2D(b)mode diagram of the interaction between EDEGWYWCGVK and Keap1 active site

图4 QNCDQF 与Keap1 活性位点相互作用的3D(a)和2D(b)模式图
Fig. 4 3D(a)and 2D(b)pattern of interaction between QNCDQF and Keap1 active site
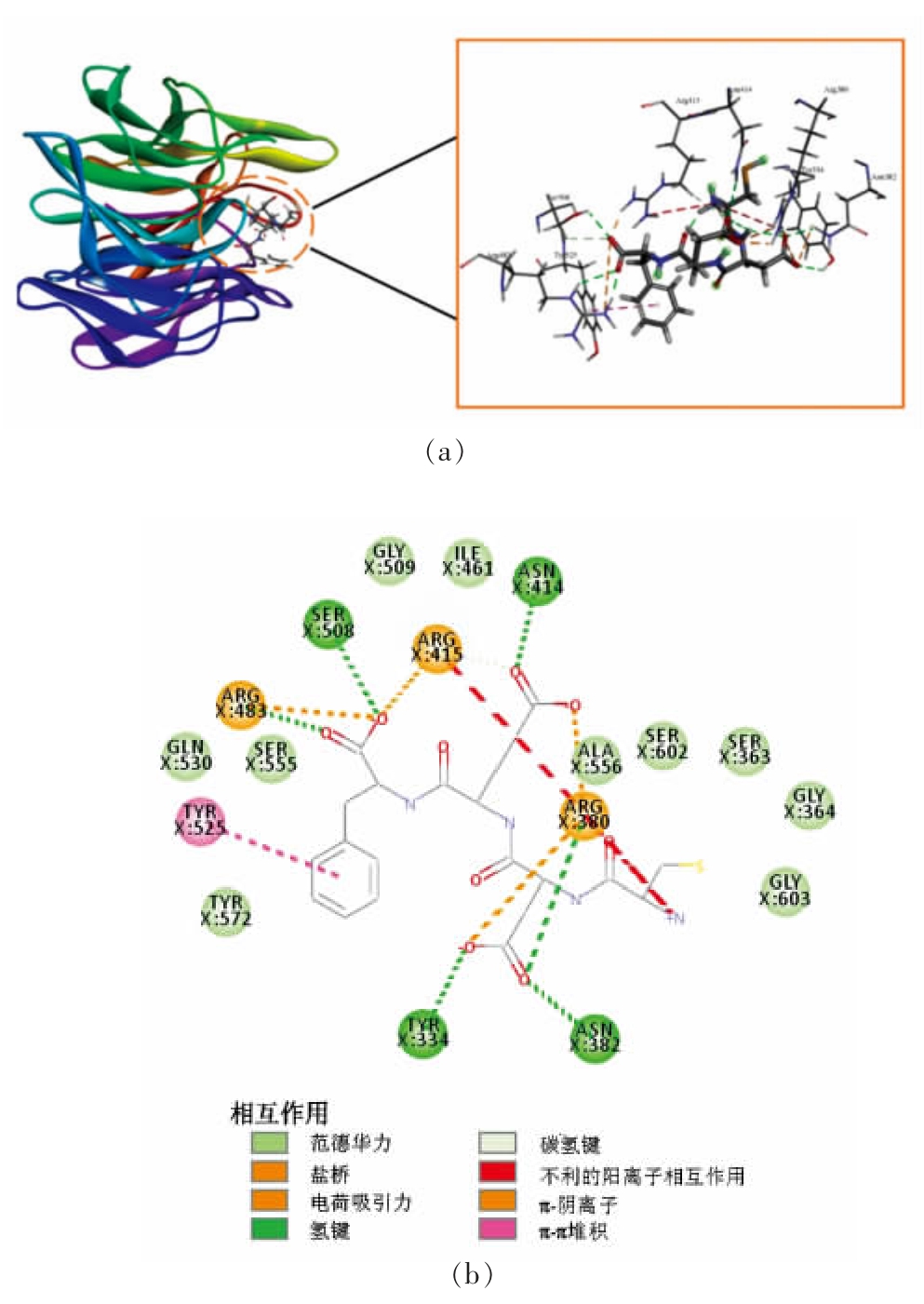
图5 CDEF 与Keap1 活性位点相互作用的3D(a)和2D(b)模式图
Fig. 5 3D(a)and 2D(b)pattern of interaction between CDEF and Keap1 active site
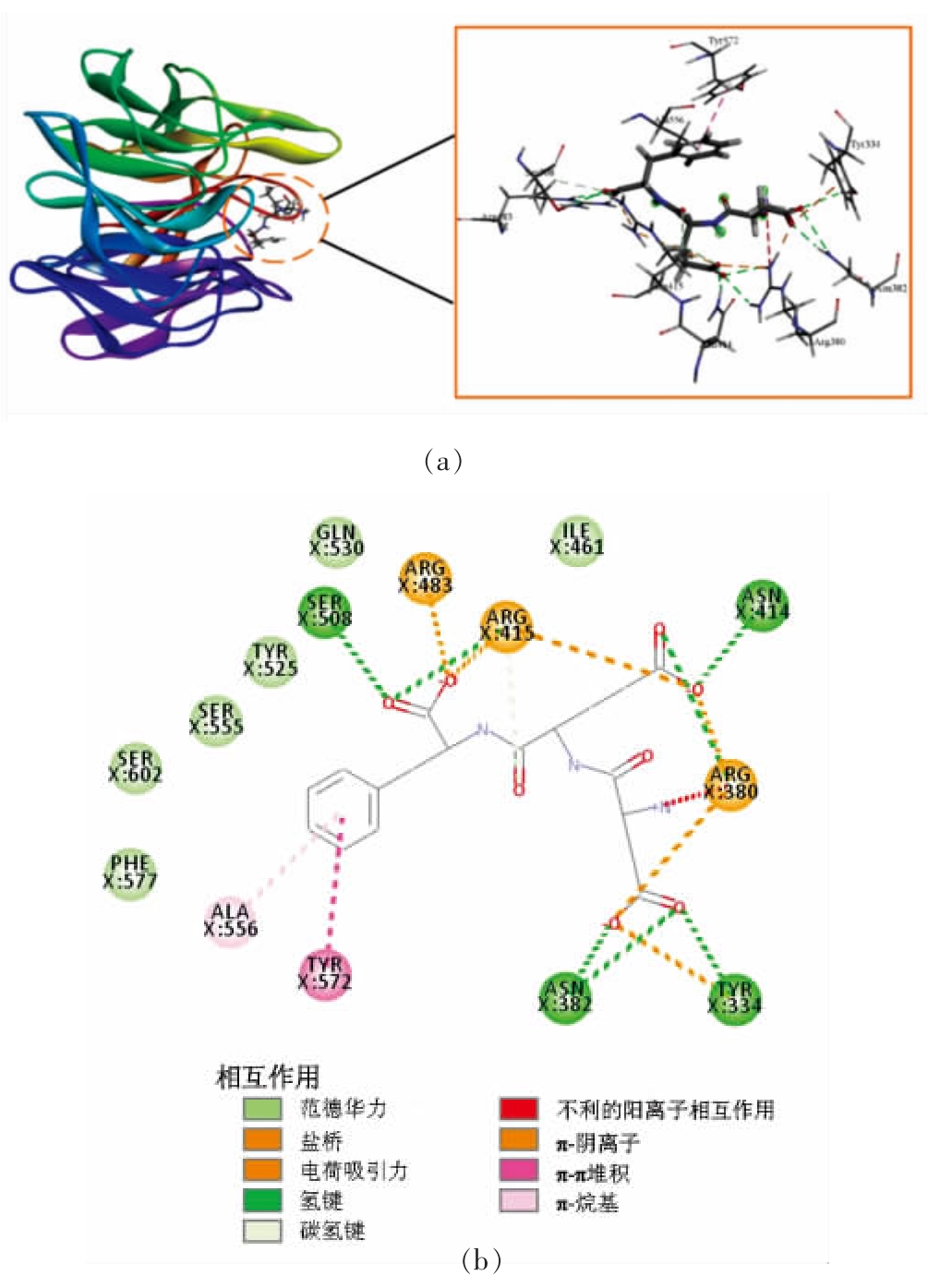
图6 DEF 与Keap1 活性位点相互作用的3D(a)和2D(b)模式图
Fig. 6 3D(a)and 2D(b)pattern of interaction between DEF and Keap1 active site
综合分析上述结果,4 种所选多肽相较阳性对照(WYSL 序列)均具有更多影响Keap1-Nrf2相互作用的重要残基,说明本研究前期抗氧化肽筛选过程的合理性与严谨性。同时可以发现所选4 种多肽中的CDEF 序列与Keap1 分子间存在最多影响Keap1-Nrf2 相互作用的关键残基(12 个),同时也存在最多的氢键作用力(6 个),因此CDEF序列可能是所选肽段中具有最高抗氧化活性的多肽序列,它可以与Keap1 分子竞争性结合并释放Nrf2 因子,并通过激活Keap1-Nrf2-ARE 途径在体内显示抗氧化活性。与本研究结果相似的是,Wei 等[53]从发酵鲁宾奶酪中获得的YPFPGPIH 肽序列也可以激活Keap1-Nrf2 途径激活抗氧化机制。
2.4 分子动力学模拟
分子对接是从结构的角度出发去评估对接结合的紧密程度,而药物的设计不能单从结构出发,往往需要考虑溶剂、温度及反应动力学的因素,分子动力学模拟是量化局部和全局蛋白质动力学的强大计算工具[54]。从分子对接中筛选出的最具抗氧化活性潜力的CDEF 序列,将其与Keap1 分子最优结合复合物进行分子动力学模拟,具体模拟结果见图7。结果显示,CDEF-Keap1 复合物在分子动力学模拟过程中,RSMD 在所产生的100 个构象中逐渐稳定,最终在200 ps 左右稳定时结合自由能约为210.87 kJ/mol,说明复合物在分子动力学模拟过程中结构变化相对平稳,氢键热图说明了复合物在模拟过程中与较多小分子形成氢键作用力,在前80 个构象氢键变化并不显著,因此可判断出复合物可能没有经历较大的构象变化[55]。进一步说明了筛选出的抗氧化肽CDEF 可以在人体中较好地发挥其抗氧化活性。

图7 CDEF-Keap1 复合物的分子动力学模拟RMSD 图(a)、能量变化图(b)及氢键热图(c)
Fig. 7 Molecular dynamics simulation RMSD diagram(a),energy change diagram(b)and hydrogen bond heat diagram(c)of CDEF-keap1 complex
3 结论
本研究通过虚拟消化牛乳清蛋白,将能够抵御胃肠道消化的肽段进行初步酶解筛选,并通过相关活性预测及药物代谢动力学对肽段进行细致筛选,将无毒、无致敏性、无致突变性、无致癌性及水溶性良好的肽段挑选出来,并对符合条件的肽段与Keap1 受体进行半柔性分子对接,通过分子对接评分和作用机理分析对肽段的抗氧化活性潜力进行评估,最终筛选出了最具抗氧化活性潜力的CDEF 肽序列,其与Keap1 受体的氨基酸残基Arg483、Arg415、Arg380、Ser508、Tyr334、Tyr334、Tyr525、Gln530、Ser555、Ser602、Ser363、Tyr572 之间形成的盐桥、电荷吸引力、π-阴离子作用、常规氢键、碳氢键、π-π 相互作用及范德华力,可能有助于激活Keap1-Nrf2-ARE 途径使其在体内显示抗氧化活性。分子动力学模拟分析表明CDEF 肽序列具有良好的结合能力与稳定性,可能成为抗氧化的候选药物。本研究基于生物信息学的手段详细阐述了如何从牛乳清蛋白源筛选出抗氧化肽,研究结果对进一步认识肽的抗氧化作用机制有一定的指导意义,为后续筛选天然食品源抗氧化肽提供了一定的理论基础。
[1] ABEYRATHNE E D N S,NAM K,HUANG X,et al.Plant-and animal-based antioxidants' structure,efficacy,mechanisms,and applications:A review[J].Antioxidants,2022,11(5):1025.
[2] TOLDRA F,GALLEGO M,REIG M,et al.Recent progress in enzymatic release of peptides in foods of animal origin and assessment of bioactivity[J].J Agric Food Chem,2020,68(46):12842-12855.
[3] ABD-TALIB N,YAJI E L A,ABD WAHAB N S,et al.Bioactive peptides and its alternative processes:A review[J].Biotechnology and Bioprocess Engineering,2022,27(3):306-335.
[4] LEON -LOPEZ A,PEREZ -MARROQUIN X A,ESTRADA-FERNANDEZ A G,et al.Milk whey hydrolysates as high value-added natural polymers:Functional properties and applications[J].Polymers,2022,14(6):1258.
[5] BIELECKA M,CICHOSZ G,CZECZOT H.Antioxidant,antimicrobial and anticarcinogenic activities of bovine milk proteins and their hydrolysates -A review[J].International Dairy Journal,2022,127(1):105208.
[6] PIHLANTO A.Antioxidative peptides derived from milk proteins[J].International Dairy Journal,2006,16(11):1306-1314.
[7] POWER O,JAKEMAN P,FITZGERALD R J.Antioxidative peptides:Enzymatic production,in vitro and in vivo antioxidant activity and potential applications of milk -derived antioxidative peptides [J].Amino Acids,2013,44(3):797-820.
[8] PENG X Y,KONG B H,XIA X F,et al.Reducing and radical-scavenging activities of whey protein hydrolysates prepared with Alcalase[J].International Dairy Journal,2010,20(5):360-365.
[9] DALIRI E B,LEE B H,PARK B J,et al.Antihypertensive peptides from whey proteins fermented by lactic acid bacteria [J].Food Sci Biotechnol,2018,27(6):1781-1789.
[10] CORROCHANO A R,FERRARETTO A,ARRANZ E,et al.Bovine whey peptides transit the intestinal barrier to reduce oxidative stress in muscle cells[J].Food Chem,2019,288(1):306-314.
[11] STOBIECKA M,KROL J,BRODZIAK A.Antioxidant activity of milk and dairy products[J].Animals,2022,12(3):245.
[12] GEORGE S,ABRAHAMSE H.Redox potential of antioxidants in cancer progression and prevention[J].Antioxidants(Basel),2020,9(11):1156.
[13] KARASAHIN T,ALKAN H,SATILMIS F,et al.Relationship between total antioxidant/oxidant status,and oxidative stress index and superovulation response in donor cows[J].Livest Sci,2021,244(1):104340.
[14] MADRAZO A L,CAMPOS M R S.In silico prediction of peptide variants from chia(S.hispanica L.)with antimicrobial,antibiofilm,and antioxidant potential[J].Computational Biology and Chemistry,2022,98(1):107695.
[15] OLSEN T H,YESILTAS B,MARIN F I,et al.AnOxPePred:Using deep learning for the prediction of antioxidative properties of peptides[J].Sci Rep-Uk,2020,10(1):21471.
[16] DROR R O,DIRKS R M,GROSSMAN J P,et al.Biomolecular simulation:A computational microscope for molecular biology[J].Annu Rev Biophys,2012,41(1):429-452.
[17] HOLLINGSWORTH S A,DROR R O.Molecular dynamics simulation for all[J].Neuron,2018,99(6):1129-1143.
[18] MADUREIRA A R,PEREIRA C I,GOMES A M P,et al.Bovine whey proteins -Overview on their main biological properties[J].Food Research International,2007,40(10):1197-1211.
[19] MINKIEWICZ P,IWANIAK A,DAREWICZ M.BIOPEP-UWM database of bioactive peptides:Current opportunities[J].International Journal of Molecular Sciences,2019,20(23):5978.
[20] AHMED A S,EL-BASSIONY T,ELMALT L M,et al.Identification of potent antioxidant bioactive peptides from goat milk proteins[J].Food Research International,2015,74(1):80-88.
[21] SINGH S,CHAUDHARY K,DHANDA S K,et al.SATPdb:A database of structurally annotated therapeutic peptides[J].Nucleic Acids Research,2015,44(D1):D1119-D1126.
[22] GUPTA S,KAPOOR P,CHAUDHARY K,et al.In silico approach for predicting toxicity of peptides and proteins[J].PLoS One,2013,8(9):e73957.
[23] DIMITROV I,BANGOV I,FLOWER D R,et al.AllerTOP v.2 -A server for in silico prediction of allergens[J].Journal of Molecular Modeling,2014,20(6):2278.
[24] WILKINS M R,GASTEIGER E,BAIROCH A,et al.Protein identification and analysis tools in the ExPASy server [J].Methods in Molecular Biology(Clifton,NJ),1999,112(1):531-552.
[25] EGAN W J,MERZ K M J R,BALDWIN J J.Prediction of drug absorption using multivariate statistics[J].J Med Chem,2000,43(21):3867-3677.
[26] CHENG A,MERZ K M J R.Prediction of aqueous solubility of a diverse set of compounds using quan titative structure-property relationships[J].J Med Chem,2003,46(17):3572-3580.
[27] EGAN W J,LAURI G.Prediction of intestinal per meability[J].Advanced Drug Delivery Reviews,2002,54(3):273-289.
[28] SUSNOW R G,DIXON S L.Use of robust classification techniques for the prediction of human cytochrome P450 2D6 inhibition[J].J Chem Inf Comp Sci,2003,43(4):1308-1315.
[29] XIA X Y,MALISKI E G,GALLANT P,et al.Classification of kinase inhibitors using a Bayesian model[J].Journal of Medicinal Chemistry,2004,47(18):4463-4470.
[30] CHENG A,DIXON S L.In silico models for the prediction of dose-dependent human hepatotoxicity[J].J Comput Aided Mol Des,2003,17(12):811-823.
[31] VOTANO J R,PARHAM M,HALL L M,et al.QSAR modeling of human serum protein binding with several modeling techniques utilizing structureinformation representation [J].Journal of Medicinal Chemistry,2006,49(24):7169-7181.
[32] 李良煜.基于Keap1-Nrf2 通路的蛋清源抗氧化小肽筛选及其作用机制研究[D].长春:吉林大学,2017.LI L Y.Screening of small antioxidant peptides from egg albumin based on Keap1 -Nrf2 pathway and their mechanism of action[D].Changchun:Jilin University,2017.
[33] WANG Y Q,GUO H Q,FENG Z W,et al.PD-1-targeted discovery of peptide inhibitors by virtual screening,molecular dynamics simulation,and surface plasmon resonance[J].Molecules,2019,24(20):3784.
[34] SONTAKKE S B,JUNG J H,PIAO Z,et al.Orally available collagen tripeptide:Enzymatic stability,intestinal permeability,and absorption of Gly-Pro-Hyp and Pro-Hyp[J].J Agric Food Chem,2016,64(38):7127-7133.
[35] ABDELKARIM B,SABRINE S,MARWA H,et al.The brown seaweed Cystoseira schiffneri as a source of sodium alginate:Chemical and structural characterization,and antioxidant activities[J].Food Biosci,2020,40(1):100873.
[36] 池桂良,战鑫.乳清蛋白肽的营养及其在食品中的应用[J].中国食品工业,2021,2021(14):114-118.CHI G L,ZHAN X.Nutrition of whey protein peptide and its application in food[J].China Food Industry,2021,2021(14):114-118.
[37] 陈艳楠,邱智军,刘学强,等.紫贻贝(Mytilus edulis)蛋白计算机模拟消化物活性的生物信息学分析[J].食品与发酵工业,2022,48(15):185-192.CHEN Y N,QIU Z J,LIU X Q,et al.Bioinformatics analysis of computer simulated digestive activity of Mytilus edulis protein[J].Food and Fermentation Industry,2022,48(15):185-192.
[38] LAI Y R,CHU X Y,DI L,et al.Recent advances in the translation of drug metabolism and pharmacokinetics science for drug discovery and development[J].Acta Pharm Sin B,2022,12(6):2751-2577.
[39] 丁海波.新型多肽AWRK6 在SD 大鼠体内的药代动力学研究[D].沈阳:辽宁大学,2020.DING H B.Pharmacokinetic study of a novel polypeptide AWRK6 in SD rats[D].Shenyang:Liaoning University,2020.
[40] JIA C Y,LI J Y,HAO G F,et al.A drug-likeness toolbox facilitates ADMET study in drug discovery[J].Drug Discov Today,2020,25(1):248-258.
[41] LEE J W,MARIA-SOLANO M A,VU T N L,et al.Big data and artificial intelligence(AI)methodologies for computer-aided drug design(CADD)[J].Biochem Soc Trans,2022,50(1):241-252.
[42] YOU L J,ZHAO M,REGENSTEIN J M,et al.Changes in the antioxidant activity of loach(Misgurnus anguillicaudatus)protein hydrolysates during a simulated gastrointestinal digestion[J].Food Chemistry,2010,120(3):810-816.
[43] 赵贵琴,李婷婷,宋敏杰,等.分子对接技术筛选鲈鱼肌球蛋白中黄嘌呤氧化酶抑制肽[J].中国食品学报,2021,21(6):81-91.ZHAO G Q,LI T T,SONG M J,et al.molecular docking technology for screening xanthine oxidase inhibitory peptides in bass myosin[J].Chinese Journal of Food,2021,21(6):81-91.
[44] MENG X Y,ZHANG H X,MEZEI M,et al.Molecular docking:A powerful approach for structure-based drug discovery[J].Curr Comput Aided Drug Des,2011,7(2):146-157.
[45] PINZI L,RASTELLI G.Molecular docking:Shifting paradigms in drug discovery [J].Int J Mol Sci,2019,20(18):4331.
[46] ZHU L J,XIONG H G,HUANG X,et al.Identification and molecular mechanisms of novel antioxidant peptides from two sources of eggshell membrane hydrolysates showing cytoprotection against oxidative stress:A combined in silico and in vitro study[J].Food Res Int,2022,157(1):111266.
[47] CHAPPLE S J,SIOW R C M,MANN G E.Crosstalk between Nrf2 and the proteasome:Therapeutic potential of Nrf2 inducers in vascular disease and aging[J].Int J Biochem Cell B,2012,44(8):1315-1320.
[48] LI X Y,FENG C S,HONG H,et al.Novel ACE inhibitory peptides derived from whey protein hydrolysates:Identification and molecular docking analysis[J].Food Biosci,2022,48(1):101737.
[49] YU Z P,KAN R T,WU S J,et al.Xanthine oxidase inhibitory peptides derived from tuna protein:Virtual screening,inhibitory activity,and molecular mechanisms[J].J Sci Food Agric,2021,101(4):1349-1354.
[50] ZHANG Q X,WU H,LING Y F,et al.Isolation and identification of antioxidant peptides derived from whey protein enzymatic hydrolysate by consecutive chromatography and Q-TOF MS[J].J Dairy Res,2013,80(3):367-373.
[51] I T K,YASUTAKE K,HIDEKI K,et al.Keap1 recruits Neh2 through binding to ETGE and DLG motifs:Characterization of the two-site molecular recognition model[J].Molecular and Cellular Biology,2006,26(8):2887-2900.
[52] SHIH-CHING L,XUCHU L,T H M,et al.Structure of the Keap1:Nrf2 interface provides mechanistic insight into Nrf2 signaling[J].The EMBO journal,2006,25(15):3605-3617.
[53] WEI G Q,ZHAO Q,WANG D D,et al.Novel ACE inhibitory,antioxidant and α-glucosidase inhibitory peptides identified from fermented rubing cheese through peptidomic and molecular docking[J].LWT-Food Sci Technol,2022,159(1):113196.
[54] GONZALEZ N A,LI B A,MCCULLY M E.The stability and dynamics of computationally designed proteins[J].Protein Eng Des Sel,2022,35(1):1093.
[55] 唐彪,王浩翔,付晓丽,等.基于Discovery Studio平台对TGF-β1 中药小分子抑制剂的虚拟筛选[J].中华中医药杂志,2021,36(11):6669-6673.TANG B,WANG H X,FU X L,et al.Virtual screening of TGF-β1 inhibitors of Chinese materia medica small molecule based on Discovery Studio platform[J].Chinese Journal of traditional Chinese medicine,2021,36(11):6669-6673.