随着社会的不断发展,乳酸菌制品逐渐走进大众的视野,并且乳酸菌已经广泛用于食品、医药健康、农牧业、工业制造等领域[1]。副干酪乳酪杆菌(Lacticaseibacillus paracasei)是一种兼性厌氧的革兰氏阳性菌,同时也是重要的乳酸菌菌种之一。副干酪乳酪杆菌分布广泛,可以从人体肠道、口腔、发酵制品以及青贮饲料中分离。Marctto等[2]研究表明,副干酪乳杆菌能够抵抗胃酸和肠道胆盐,顺利抵达婴儿肠道,并逐渐成为人体乳杆菌的优势菌群。Dang等[3]发现副干酪乳杆菌TD062 具有较高的α-酶抑制剂活性,良好的抗糖尿病能力,可作为一种新型的降血糖剂。本研究使用的副干酪乳酪杆菌PC-01 不仅可以调节肠道菌群平衡,具有良好的耐酸、耐胆盐特性[4],还具备预防高血压和降低胆固醇等益生特性。
随着测序技术的发展,全基因组测序技术被广泛应用于菌株的分类鉴定[5]。比较基因组学是在全基因组测序的基础上,分析种内或种间基因功能的相似性和差异性,以及物种的进化关系[6]。Smokvina等[7]对34 株副干酪乳杆菌进行基因组的比较分析,得到其泛基因组包含4 200 个同源基因,并从核心基因集中发现了与动物宿主相关的因子。Salvetti等[8]基于系统发育距离和保守蛋白质比例,将269 株乳杆菌重新分类,建立系统发育标记,为其遗传多样性的探究提供基础。Broadbent等[9]通过比较基因组学研究揭示干酪乳杆菌基因组的共线性较高,具有几个高度保守基因含量的大块。局部共线区域的数量随着基因组数量的增加而增加,基于相似性谱高度,检测到干酪乳杆菌之间明显的总体序列同源性,体现了局部多态性。Ghattargi等[10]通过全基因组测序和比较基因组学分析,研究菌株中与益生菌特性相关的关键遗传因素。
本研究通过全基因组测序技术结合比较基因组学的方法,以经过高密度发酵的10 株具有不同表型特征的副干酪乳酪杆菌PC-01 分离株为研究对象,采用比较基因组学和表型观察,从基因组水平上揭示同一菌株不同菌落间的差异性,为菌株在高密度发酵上的应用提供遗传学参考。
1 材料与方法
1.1 菌株来源
副干酪乳酪杆菌PC-01 菌株来自内蒙古农业大学乳酸菌菌种资源库(Lactic Acid Bacteria Collection Center,LABCC),菌株编号为 IMAU60230,副干酪 乳酪杆 菌 PC -01(IMAU60230)是2007 年分离自西藏拉萨地区酸牦牛奶的一株具有优良益生特性的菌株。
2022 年3 月14 日从NCBI(National Center for Biotechnology Information,https://www.ncbi.nlm.nih.gov/)RefSeq 数据库下载1 株副干酪乳酪杆菌PC-01 和副干酪乳酪杆菌Zhang 基因组完成图。
1.2 主要试剂和仪器
主要试剂包括MRS 培养基,广州环凯微生物科技有限公司;Wizard Genomic DNA Purifcation Kit、5×TBE 电泳缓冲液、琼脂糖、核酸染 料GELVIEW,北京百泰 克;DL2000 DNA Marker(Takara)、6×DNA Loading Buffer,Trans Gen。
Genomic DNA Purifcation Kit、5×TBE 电泳缓冲液、琼脂糖、核酸染 料GELVIEW,北京百泰 克;DL2000 DNA Marker(Takara)、6×DNA Loading Buffer,Trans Gen。
主要仪器包括ZHJH-C 型智能安全型超净工作台,上海智城分析仪器有限公司;HWS28 型水浴锅,天津知署科技有限公司;恒温培养箱,上海恒科技仪器有限公司;超微量紫外分光光度计,NanoDrop 公司;光学显微镜,日本奥林巴斯(OLYMPUS);漩涡振荡器,美国Scientific Industries 公司;电泳仪,北京六一仪器厂;凝胶成像仪,美国UVP 公司;测序仪,美国Illumina 公司。
1.3 菌株的获取
1.3.1 菌种活化及增殖培养 将冷冻保存(-80℃)的副干酪乳酪杆菌PC-01 接种于MRS 液体培养基中进行活化,32.5 ℃恒温需氧培养24 h,再以2%(体积分数)的比例接种于种子培养基中,32.5℃恒温需氧培养24 h,并且每次传代均需进行革兰氏染色镜检,确保在传代过程中没有被其它微生物污染。最后按5%(体积分数)接种量接种于发酵培养基中进行高密度发酵,32.5 ℃恒温需氧培养至对数期结束,将发酵终点的发酵液,在添加2%琼脂的MRS 平板上涂布,32.5 ℃需氧培养48 h,观察并记录菌落形态。
1.3.2 菌株DNA 提取 将上述挑取的单菌落,加入PBS 缓冲液,振荡30 s 混合均匀,然后以4 500 r/min,10 min 离心弃上清液,保留菌泥,使用Wizard  Genomic DNA Purification Kit 试剂盒提取DNA,具体试验步骤参考说明书,并使用Nano Drop 微量紫外分光光度计及0.8%的琼脂糖凝胶电泳对所提取的DNA 进行纯度和完整度的检测。
Genomic DNA Purification Kit 试剂盒提取DNA,具体试验步骤参考说明书,并使用Nano Drop 微量紫外分光光度计及0.8%的琼脂糖凝胶电泳对所提取的DNA 进行纯度和完整度的检测。
1.4 全基因组的测序与组装
将质量合格的DNA 样品低温运输至诺禾致源生物信息科技有限公司,基于第2 代高通量测序平台IlluminaNovaSeq 6000[11]进行全基因组序列测定。
对得到的数据进行过滤,获得高质量的cleandata 后,再运用软件包SOAPdenovo v2[12]对高质量的reads 进行拼接,并调试不同的kmer 值。最后进行Soap[13]验证和比对,经过校验后,调用Gap Closer 软件填补contigs 间的内部空白[14],过滤去除500 bp 以下的片段,最终获得完整序列。
1.5 比较基因组学分析
1.5.1 平均核苷酸一致性(average nucleotide identity,ANI)值的计算 本研究使用自制的Perl脚本计算副干酪乳酪杆菌PC-01 不同菌株的ANI值。计算方法参考Goris等[15]和Chen等[16]方法。
1.5.2 构建核心基因和泛基因集 首先使用Prokka v1.12 软件 [17] 对10 株副干酪乳酪杆菌PC-01 基因组分别进行基因预测及注释,以获得GFF 格式文件进行后续分析(用于计算核心基因集和泛基因集)。最后使用Roary 软件[18]构建核心基因集和泛基因集。
1.5.3 构建系统发育树 基于核心基因使用ITOL(https://itol.embl.de/login.cgi)网站进行系统发育树的构建。
1.5.4 功能基因分析 通过CAZyme(Carbohydrate Active Enzymes)在线注释数据库(https://bcb.unl.edu/dbCAN2/blast.php);对基因组序列进行注释。
通过RAST 数据库(http://rast.nmpdr.org/)对核苷酸序列进行在线注释[19-20]。
通过eggNOG 数据库(http://eggnog5.embl.de/)使用eggNOG-mapperV2 在线注释工具将基因组序列与COG 数据库比对,从而获得功能注释结果。
1.5.5 单核苷酸多态性(single nucleotide polymophism,SNP)分析 以副干酪乳酪杆菌PC-01原始全基因组序列为参考,使用mummer 软件对10 株副干酪乳酪杆菌PC-01 进行SNP 分析。
1.6 数据作图
使用TBtools 及Origin 2021 等软件进行图表的绘制。
2 结果与分析
2.1 菌落形态
经高密度发酵后涂布培养的副干酪乳酪杆菌PC-01 在同一培养基上出现5 种不同的菌落形态,将这5 种不同形态的菌落依次编号为PC-01-1、PC-01-2、PC-01-3、PC-01-4 和PC-01-5,选取相同菌落形态的两株菌编号为PC-01-1-1 和PC-01-1-2,PC-01-2-1 和PC-01-2-2,PC-01-3-1 和PC-01-3-2,PC-01-4-1 和PC-01-4-2,PC-01-5-1 和PC-01-5-2。通过观察菌落特征(表1)发现,菌落PC-01-3 和PC-01-4,PC-01-2 和PC-01-5 较为相似。
表1 菌落的基本特征
Table 1 Basic characteristics of colony

2.2 菌落全基因组测序的基本信息
通过对10 株具有不同表型特征的分离株的基因组进行二代测序,并与副干酪乳酪杆菌PC-01 原始基因组序列比较分析,基因组统计结果如表2 所示。由表2 可知,10 株分离株的基因组大小为2.69~2.83 Mb,GC 含量为46.50%~46.64%。每株分离株识别到3 050±197 个基因编码区(Coding sequences,CDs ),其中PC-01-2-1 识别到的CDs 最多,为3 406 个,PC-01-4-1 的最少,为2 793 个。结果表明,不同菌落的基因数量、基因组大小和GC 含量等指标存在较小差异。
表2 基因组的基本特征
Table 2 Basic characteristics of genomes
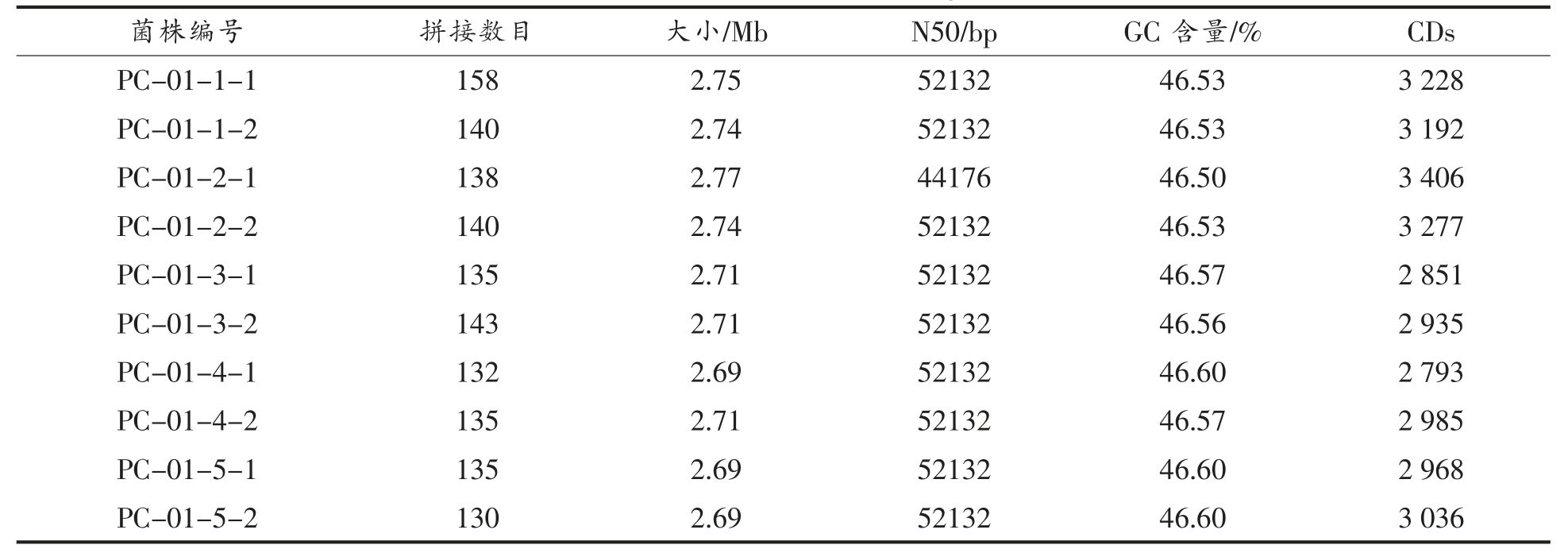
2.3 平均核苷酸一致性(ANI)分析
平均核苷酸一致性(ANI),是用来鉴定微生物基因组种内关系的黄金方法[21],可以用于在基因组水平评估物种内的亲缘关系。ANI 可以通过对任何两个菌株的同源序列进行比较计算,当ANI值>95%时,可视为同一物种[22]。结果表明,副干酪乳酪杆菌PC-01 原始基因组与10 株具有不同表型特征的副干酪乳酪杆菌 PC-01 分离株间ANI值均大于99.88%(图1),表明本试验的副干酪乳酪杆菌PC-01 分离株与原始菌株具有较高的一致性。
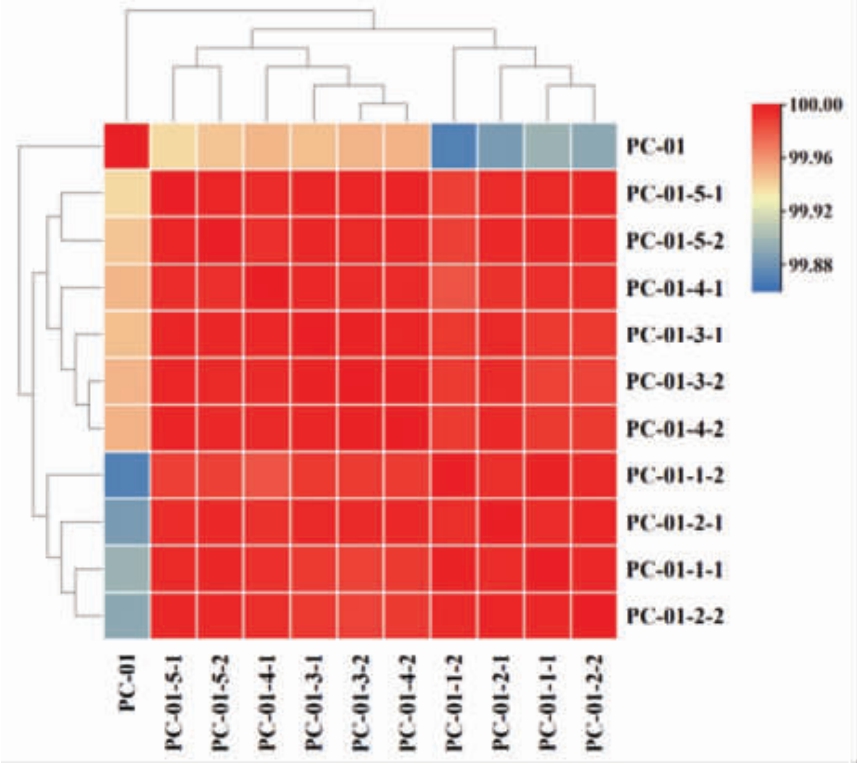
图1 平均核苷酸一致性
Fig.1 Average nucleotide identity
2.4 泛-核心基因集分析
利用Roary 软件计算11 株菌的泛基因集,得到的泛基因集(pan genes)包含2 869 个基因,其中核心基因(core genes)为2 571 个,非核心基因集为298 个,包含122 个附属基因(accessory gene)和176 个特异基因(unique gene)。由图 2a可知,随着基因组数量的增加,泛基因组数量逐渐增加,而核心基因的个数逐渐趋于稳定。对于泛基因存在与缺失的结果绘制热图,通过图2b 可知,核心基因占泛基因集的89.61%,表明11 株副干酪乳酪杆菌核心基因组高度保守。
图2 泛-核心基因集变化趋势图以及泛基因存在与缺失热图
Fig.2 The trend chart of the size of core-pan genes and pan genes presence and absence heat map
2.5 CAZy 功能注释
已有研究表明不同环境的副干酪乳酪杆菌的进化基因的衰变主要体现在碳水化合物[23]。碳水化合物 活性酶(Carbohydrate-active enzymes,CAZyme)的家族分类以及对组装序列进行注释,主要用于分析、理解和比较菌株组装和分解复杂的碳水化合物的能力[24]。因此,基于CAZy 数据库,对副干酪乳酪杆菌PC-01 原始基因组及高密度发酵后的10 株分离株在碳水化合物方面的代谢能力进行基因注释。结果表明(图3),共注释到6 大类,36 小类的碳水化合物活性酶,包括19 个糖苷水解酶(Glycoside Hydrolases,GHs)家族、8个糖基转移酶(GlycosylTransferases,GTs)家族、3个碳水化合物酯酶(Carbohydrate Esterases,CEs)家族、2 个碳水化合物结合模块(Carbohydrate-Binding Modules,CBMs)家族、3 个辅助模块酶类(Auxiliary Activities,AAs)家族和1 个多糖裂解酶(Polysaccharide Lyases,PLs)家族。11 株菌中主要以GHs 和GTs 酶类的含量最为丰富,并且不同菌株编码功能的基因数量及种类均存在差异,其差异主要体现在编码GH 家族中的GH1、GH13_29和GH13_31 酶的基因数量不同,这类酶主要与α-葡萄糖苷酶、6-磷酸-β-半乳糖苷酶等酶类生物合成相关,以及编码GT 家族中的GT2 酶的基因数量不同,这类酶主要与半乳糖、蔗糖等二糖以及低聚糖和多糖的生物合成相关。
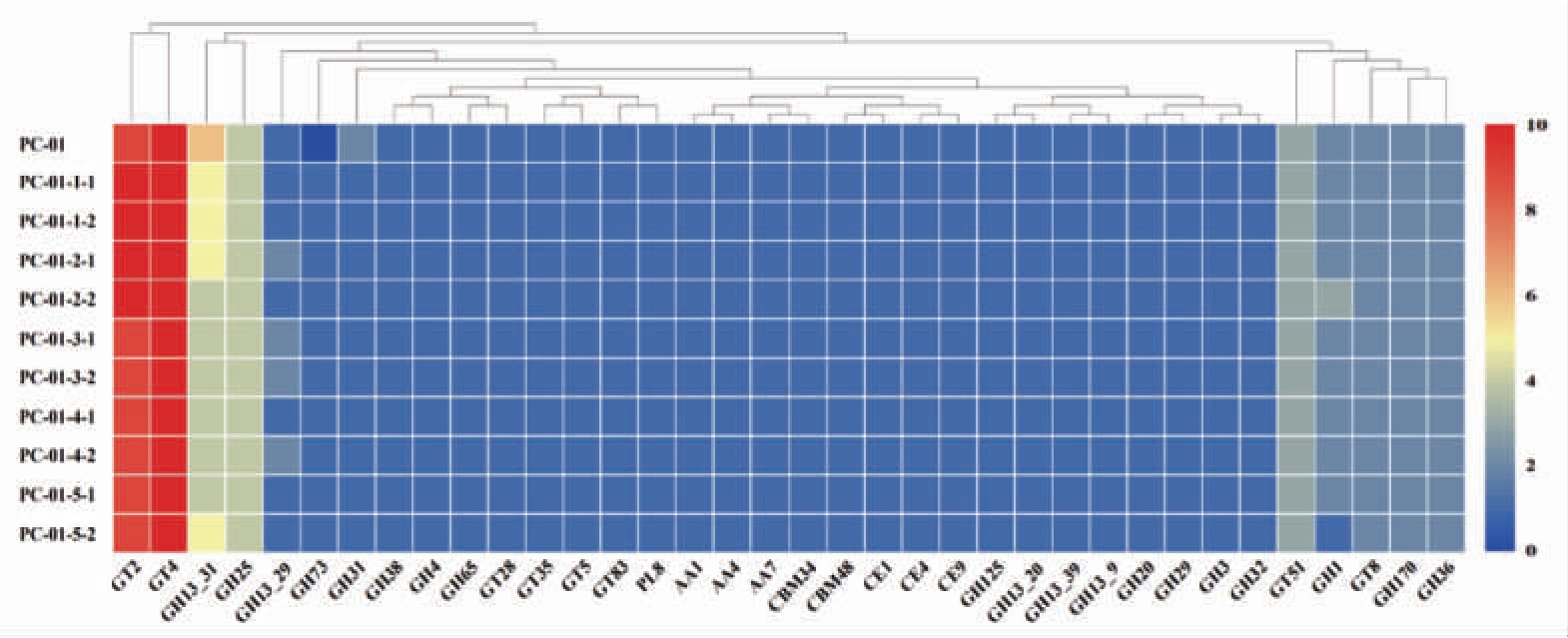
图3 CAZy 注释结果
Fig.3 CAZy annotated result
2.6 RAST 功能注释
利用RAST 在线工具对11 株副干酪乳酪杆菌PC-01 基因组进行更全面的功能基因注释,共注释得到23 个功能大类(图4),功能注释结果显示,11 株副干酪乳酪杆菌PC-01 中参与碳水化合物代谢(Carbohydrates)的基因数量最多,其次是参与蛋白代谢(Protein Metabolism)以及氨基酸及其衍生物合成及降解(Amino acids and derivatives)的基因。并且参与这些功能的基因种类及数目存在差异,碳水化合物代谢中存在差异的主要是参与乳糖和半乳糖的摄取和利用的α-半乳糖苷酶(GalA)和6-磷酸-β-半乳糖苷酶(LacG)。蛋白代谢中存在差异的主要是参与蛋白质生物合成的rRNA。氨基酸及其衍生物合成及降解中存在差异的主要是参与蛋氨酸降解和生物合成以及半胱氨酸生物合成的胱硫氨酸γ 裂解酶、胱硫氨酸β-合酶和半胱氨酸合酶等。

图4 RAST 注释结果
Fig.4 Result of RAST annotation
2.7 系统发育树分析
系统发育分析可以了解同一群体内不同个体间的亲缘关系,因此构建系统发育树是研究种群结构和物种进化的必要前提[25]。为了探究这11 株副干酪乳酪杆菌PC-01 之间的进化关系,选择与副干酪乳酪杆菌遗传距离较近的副干酪乳酪杆菌Zhang(Lacticaseibacillus paracasei Zhang)作为外群菌株,使用核心基因构建系统发育树,如图5 所示,系统发育树主要分为2 个分支,且11 株副干酪乳酪杆菌PC-01 与副干酪乳酪杆菌Zhang 处于两大分支,10 株副干酪乳酪杆菌PC-01 与副干酪乳酪杆菌PC-01 原始菌株聚在同一分支(BranchII)。BranchII 又分为3 个进化支(Clade),CladeI 中的PC-01-1-1、PC-01-1-2 与PC-01的遗传距离最短,表明其亲缘关系最接近。CladeⅡ分支包含PC-01-3-1、PC-01-3-2、PC-01-4-1 和PC-01-4-2。CladeⅢ分支包含PC-01-2-1、PC-01-2-2、PC-01-5-1 和PC-01-5-2。并且通过菌落形态统计结果发现PC-01-3 和PC-01-4 呈半透明、表面不光滑、边缘不整齐的菌落形态,PC-01-2 和PC-01-5 呈透明、表面光滑、边缘整齐、扁平的菌落形态,均存在较为相似的菌落形态。这表明菌株的遗传距离会对菌落形态产生影响。

图5 基于核心基因序列构建系统发育树
Fig.5 Construct a phylogenetic tree based on core gene sequences
2.8 SNPs 位点分析
以副干酪乳酪杆菌PC-01 原始菌株基因组作为参考,对10 株分离菌株进行SNP 分析。共识别到121 个SNPs 位点,包括81 个非同义突变和20 个同义突变,其余突变位于基因间区。非同义突变位点中,除去编码假定蛋白的基因,共有62个突变位点位于编码区。通过比较不同菌株的SNP 位点差异,发现PC-01-1、PC-01-2、PC-01-3、PC-01-4、PC-01-5 5 种不同菌落形态的菌株中存在不同的SNPs 位点(图6);且具有相同菌落形态的两株菌,例如PC-01-1-1 和PC-01-1-2 存在19 个相同的突变位点,其中非同义突变为11个,同义突变为8 个。除此之外,PC-01-3 和PC-01-4 具有相似的菌落形态,存在8 个相同的突变位点,其中非同义突变为5 个,同义突变为3 个,且由表1 可知菌落形态与其它菌落不同。这表明菌株的变异与菌落形态存在关联。

图6 基于121 个SNP 位点绘制UpSet图
Fig.6 UpSet plot based on 121 SNP sites
通过COG 进行功能注释,可以对其进行分析及归类[26],并预测不同分离株中相关蛋白质的主要功能。因此,对81 个非同义突变位点所对应的基因进行COG 注释,由注释结果(图7)可知,除功能未知(S)的基因外,与复制、重组和修复(L)相关的功能基因最多,其次是参与碳水化合物转运和代谢(G)及核苷酸转运和代谢(F)基因。
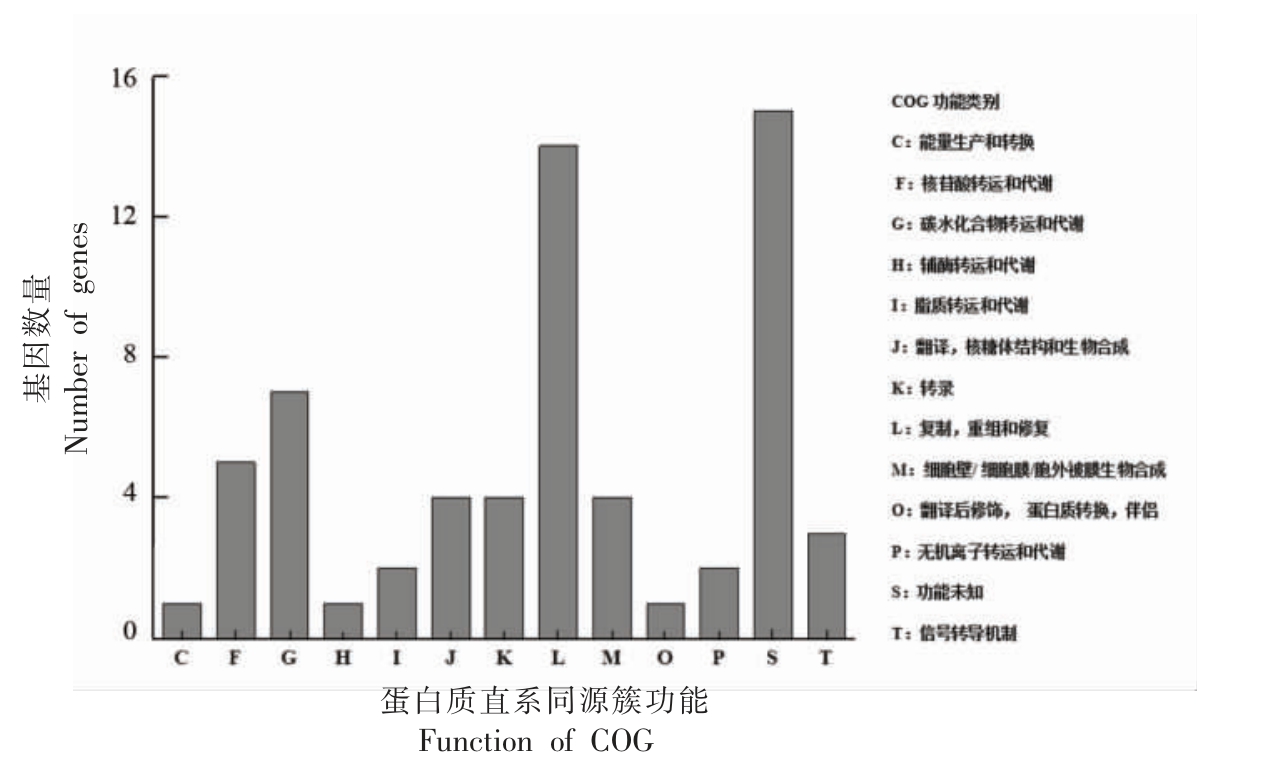
图7 COG 注释结果
Fig.7 COG annotation results
进一步分析发现,10 株分离株中均存在与糖基转移酶和ABC 蛋白家族相关的突变位点。非同义突变中包含的GT8 家族蛋白以及epsJ、pbpF、ponA 等基因均与糖基转移酶相关,糖基转移酶是合成乳酸菌胞外多糖(EPS)的关键酶,参与生物膜形成、细胞黏附等过程[27]。在细菌的生命活动中,菌体还可以通过产生大量胞外多糖,来保护细菌细胞免受干燥缺水、温度过高等各种极端条件影响,从而维持菌体形态。与ABC 蛋白家族相关突变位点,包括UVrABC 系统蛋白A、UVrABC 系统蛋白B、甜菜碱/肉碱运输渗透酶蛋白GbuB、推定运输蛋白HsrA、柔红霉素/阿霉素耐药ATP 结合蛋白DrrA、推定的ABC 转运蛋白ATP 结合蛋白YheS 和杆菌肽转运ATP 结合蛋白BceA 等,并且ABC 蛋白家族是一类家族庞大、功能多样的跨膜转运蛋白,普遍存在于所有生物体中,主要参与初级和次级代谢物质的转运[28],包括一些小分子,例如糖类、核苷酸等,还有一些大分子如蛋白质、多肽等。另外还参与代谢解毒、信号转导、脂质稳态平衡和病毒防御等重要生理过程[29~30],从而调节微生物的生命活动[31]。这也可能是在高密度发酵过程中,菌株能更好的适应环境,从而存活下来的原因。
另一方面,菌落PC-01-3、PC-01-4 中均存在ltaS 基因发生了非同义突变,ltaS 基因编码的脂磷壁酸合酶(LtaS)[32]是革兰氏染色阳性细菌细胞壁生物合成的关键酶。缺乏LtaS 的革兰氏阳性细菌表现出细胞分裂受损和生长缺陷,这可能是菌落PC-01-3、PC-01-4 形成半透明、表面不光滑、边缘不整齐的菌落形态的原因。
除此之外,在菌落PC-01-1 和PC-01-2 中还存在磷酸转移酶、D-半乳糖脱水酶(dgoD)非同义突变位点属于磷酸转移酶系统(PTS),其调控碳水化合物的运输和代谢,在调控分解代谢基因和操纵子的表达中发挥重要作用[33]。并且还存在较多参与其它代谢途径(脂质代谢,维生素代谢等),遗传信息处理(DNA 修复和重组)相关的突变位点。而菌落PC-01-5 中存在的突变位点大多与遗传信息处理相关,与PC-01-1 和PC-01-2 相比缺少了代谢等功能相关的非同义突变,这也可能是PC-01-5 的菌落形态相对较小的原因。综上所述,这些突变的发生可能是菌落形态存在差异的重要原因。
3 结论
本研究以副干酪乳酪杆菌PC-01 原始基因序列作为参考菌株,对高密度发酵后的副干酪乳酪杆菌PC-01 的10 个不同分离株进行全基因组测序、组装得到11 株菌的基因组基本信息,基因组分析表明,不同菌落的基因数目、大小、GC 含量等指标存在较小差异。通过CAZyme、RAST 功能注释,发现编码不同功能的基因种类及数目不同。基于核心基因构建系统发育树证明遗传距离会对菌落形态产生影响,同时对SNPs 位点进行分析进一步证实了菌落形态与基因型相关联。综上所述,本研究从全基因组水平完成了副干酪乳酪杆菌PC-01 的10 个不同株的比较分析,结果表明同一菌株具有不同表型的菌落在基因组水平上的差异,这也为副干酪乳酪杆菌PC-01 的后续研究提供了理论依据和数据支持。
[1] 陈卫.乳酸菌科学与技术[M].北京:科学出版社,2018:1-19.CHEN W.Science and technology of lactic acid Bacteria[M].Beijing:Science Press,2018:1-19.
[2] MARZOTTO M,MAFFEIS C,PATERNOSTER T,et al.Lactobacillus paracasei A survives gastrointestinal passage and affects the fecal microbiota of healthy infants[J].Research in Microbiology,2006,157(9):857-866.
[3] DANG F F,JIANG Y J,PAN R L,et al.Administration of Lactobacillus paracasei ameliorates type 2 diabetes in mice[J].Food and Function,2018,9(7):3630-3639.
[4] 刘凯龙,康小红,张哲,等.副干酪乳杆菌PC-01益生特性和安全性研究[J].中国食品学报,2021,21(11):47-52.LIU K L,KANG X H,ZHANG Z,et al.Studies on probiotic properties and safety of Lactobacillus paracasei PC-01[J].Journal of Chinese Institute of Food Science and Technology,2021,21(11):47-52.
[5] PRADHAN D,MALLAPPA R H,GROVER S,et al.Comprehensive approaches for assessing the safety of probiotic bacteria[J].Food Control,2020(108):106872.
[6] WEI L,LIU Y,DUBCHAK I,et al.Comparative genomics approaches to study organism similarities and differences[J].Journal of Biomedical Informatics,2002,35(2):142-150.
[7] SMOKVINA T,WELS M,POLKA J,et al.Lactobacillus paracasei comparative genomics:towards species pan-genome definition and exploitation of diversity[J].PloS One,2013,8(7):1-18.
[8] SALVETTI E,HARRIS H M B,FELIS G E,et al.Comparative genomics reveals robust phylogroups in the genus Lactobacillus as the basis for reclassification[J].Applied and Environmental Microbiology,2018,84(17):1-15.
[9] BROADBENT J R,NEENO -ECKWALL E C,STAHL B,et al.Analysis of the Lactobacillus casei supragenome and its influence in species evolution and lifestyle adaptation[J].Bmc Genomics,2012,13(1):533.
[10] GHATTARGI V C,GAIKWAD M A,METI B S,et al.Comparative genome analysis reveals key genetic factors associated with probiotic property in Enterococcus faecium strains[J].BMC Genom,2018,19(1):1-16.
[11] MODI A,VAI S,CARAMELLI D,et al.The Illumina sequencing protocol and the NovaSeq 6000 system[J].Methods in Molecular Biology(Clifton,N.J.),2021,2242(5):15-42.
[12] LUO R B,LIU B H,XIE Y L,et al.SOAPdenovo2:an empirically improved memory -efficient short-read de novo assembler[J].Giga Science,2012,1(1):18.
[13] LI R Q,YU C,LI Y R,et al.SOAP2:an improved ultrafast tool for short read alignment [J].Bioinformatics,2009,25(15):1966-1967.
[14] KURTZ S,PHILLIPPY A,DELCHER A L,et al.Versatile and open software for comparing large genomes[J].Genome Biology,2004,5(2):1-9.
[15] GORIS J,KONSTANTINIDIS K T,KLAPPENBACH J A,et al.DNA-DNA hybridization values and their relationship to whole-genome sequence similarities[J].International Journal of Systematic and Evolutionary Microbiology,2007,57:81-91.
[16] CHEN J P,YANG X W,CHEN J W,et al.SISP:a fast species identification system for prokaryotes based on total nucleotide identity of whole genome sequences[J].Infectious Diseases &Translational Medicine,2015(1):30-55.
[17] SEEMANN T.Prokka:Rapid prokaryotic genome annotation[J].Bioinformatics,2014,30(14):2068-2069.
[18] PAGE A J,CUMMINS C A,MARTIN H,et al.Roary:rapid large -scale prokaryote pan genome analysis[J].Bioinformatics,2015(22):3691-3693.
[19] AZIZ R K,BARTELS D,BEST A A,et al.The RAST Server:rapid annotations using subsystems technology[J].Bmc Genomics,2008,9(1):75-75.
[20] OVERBEEK R,OLSON R,PUSCH G D,et al.The SEED and the rapid annotation of microbial genomes using subsystems technology(RAST)[J].Nucleic Acids Research,2013,42(1):206-214.
[21] CHIRAG J,LUIS M R R,ADAM M P,et al.High throughput ANI analysis of 90K prokaryotic genomes reveals clear species boundaries[J].Nature Communications,2018,9(1):5114.
[22] ARAHAL D.Whole -genome analyses:Average nucleotide identity[J].Methods in Microbiology,2014,41(12):103-122.
[23] FONTANA A,ZACCONI C,MORELLI L.Genetic signatures of dairy Lactobacillus casei group [J].Frontiers in Microbiology,2018,9(1):1-9.
[24] LOMBARD V,GOLACONDA R H,DRULA E,et al.The carbohydrate -active enzymes database(CAZy)in 2013[J].Nucl Acids Res,2014,42(1):490-495.
[25] 宋宇琴.德氏乳杆菌保加利亚亚种的群体遗传学和功能基因组学研究[D].呼和浩特:内蒙古农业大学,2018.SONG Y Q.Study on population genetics and functional genomics of Lactobacillus delbrueckii subsp.bulgaricus[D].Hohhot:Inner Mongolia Agricultural University,2018.
[26] GALPERIN M Y,MAKAROVA K S,WOLF Y I,et al.Expanded microbial genome coverage and improved protein family annotation in the COG database[J].Nucl Acids Res,2015,43(1):261-269.
[27] CAGGIANIELLO G,KLEEREBEZEM M,SPANO G.Exopolysaccharides produced by lactic acid bacteria:from health-promoting benefits to stress toler ance mechanisms[J].Applied Microbiology &Biotechnology,2016,100(9):3877-3886.
[28] 刘姣,高志鹏,周有祥,等.古细菌、细菌和真核生物中ATP 结合蛋白家族的全基因组分析[J].基因组学与应用生物学,2021,40(Z1):2100-2108.LIU J,GAO Z P,ZHOU Y X,et al.Genomewide analysis of ATP-binding cassette family across archaea,bacteria,and eukaryotes[J].Genomics and Applied Biology,2021,40(Z1):2100-2108.
[29] WILKENS S.Structure and mechanism of ABC transporters[J].F1000Prime Reports,2015,7(2):14.
[30] 王晓珠,孙万梅,马义峰,等.拟南芥ABC 转运蛋白研究进展[J].植物生理学报,2017,53(2):133-144.WANG X Z,SUN W M,MA Y F,et al.Research progress of ABC transporters in Arabidopsis thaliana[J].Plant Physiology Journal,2017,53(2):133-144.
[31] 孙铭优,吴照晨,王斌,等.ABC 转运蛋白及其相关的多药抗性研究现状[J].植物保护学报,2022,49(1):374-382.SUN M Y,WU Z C,WANG B,et al.Research status of ATP-binding cassette transporters and related multidrug resistance[J].Journal of Plant Protection,2022,49(1):374-382.
[32] WEZEN X C,CHANDRAN A,EAPEN R S,et al.Structure-based discovery of lipoteichoic acid synthase inhibitors[J].Journal of Chemical Information and Modeling,2022,62(10):2586-2599.
[33] JECKELMANN J M,ERNI B.Transporters of glucose and other carbohydrates in bacteria[J].Pflügers Archiv-European Journal of Physiology,2020,472(9):1129-1153.