食品工业是关系国计民生的基础性产业,是衡量一个国家或地区经济发展水平和人民生活质量的重要标志,是国民经济的支柱产业。食品产业的转型升级和高质量发展,在保障民生、拉动内需、助力脱贫攻坚等方面,发挥着举足轻重的支撑作用。酶是一种具有生物催化功能的蛋白质,其特点包括高催化活性、条件温和以及副产物少。随着蛋白质工程和基因工程的不断发展,大量新型酶制剂被不断发掘,其催化类型逐步多样化,在食品领域得到广泛应用。根据Markets and Markets 的数据,2020 年世界工业酶市场价值为59 亿美元,预计2026 年将达到87 亿美元,年复合增长率为6.5%;预计到2027 年,全球蛋白酶市场的规模将达到27 亿美元,我国的市场规模预计将达到5.505 亿美元,年复合增长率为7.6%。随着酶制剂在食品行业应用的发展,预计到2026 年,食品领域将占领酶应用市场的最大份额[1]。
优质食品酶的制备、催化与转化是食品绿色制造不可或缺的组成部分。国外大型酶制剂企业对食品酶制剂研究和开发起步较早,经过数十年的资金投入和系统研究,已建立了一系列标准化、高生产强度、高产酶水平的微生物表达体系。然而,目前我国批准使用的食品酶制剂仅60 余种,且绝大部分为国际公司申请,受专利保护,70%以上的酶制剂市场被国际公司垄断。国内食品酶领域的科技创新不足,相关技术研究的力量比较薄弱,现有产品大多为低端,附加值低,国际竞争能力弱,是生产大国而非强国(表1),技术积累远远不能满足新经济模式下食品领域需要。这一现状不仅束缚了我国相关酶制剂生产企业的发展,而且对我国食品行业的健康、快速发展形成了极其不利的影响。开发具有自主知识产权、高效、安全的食品级表达系统,开发更多自主知识产权的优质食品酶品种,对于突破国际跨国企业的束缚,促进我国食品酶制剂行业转型升级和食品产业高质量发展具有重要的意义。
表1 国内外几种主要酶制剂产酶水平比较
Table 1 Comparison of enzyme production levels of several main enzyme preparations in China and at abroad

酶工程作为现代生物技术的重要组成部分,是酶学与工程学科相融合,主要研究酶及其应用的综合性科学技术,而基因组学、蛋白质组学、生物信息学、酶的定向进化技术、高通量筛选技术等的快速发展为食品酶工程研究提供了更多有效的技术手段。为了满足食品领域和环境可持续发展的要求,研究和开发优质食品酶已成为酶工程领域的热点课题,采用优质食品酶创制高端配料也已成为一种发展趋势。鉴于此,本文综述优质食品酶的催化机制及其在高端配料创制方面的应用基础,重点关注新型食品酶高效筛选和分泌表达的生物学基础,高端高值食品酶的催化机制,食品酶催化性能改造的结构基础,食品酶创制高端配料的内在规律及作用机制等关键科学问题,以期为相关的科研工作和生产企业提供参考。
1 新型食品酶高效筛选和分泌表达的生物学基础
1.1 发展现状及需求分析
目前随着食品绿色加工产业的发展和环境资源压力的推动,人们对于新型食品酶的应用和需求迅速增加,因此新型食品酶的挖掘和开发具有重要意义。新型食品酶主要来源于自然界中的微生物,其在漫长的进化过程中需适应各种各样的环境,逐步进化成为丰富的酶资源库。对微生物资源进行挖掘和筛选,有利于开发新型酶或酶的新功能。传统的酶挖掘方法是从自然界中分离筛选产酶菌株,包括产酶菌株的富集培养、纯种分离、初筛和复筛等步骤,已经鉴定和表征了多种生物催化剂用于工业生产。然而,自然界中可培养的微生物所占比例不足微生物总量的1%,限制了新型食品酶的发现[2]。宏基因组技术具有无需微生物培养,操作步骤简单等优势,已经成为挖掘新酶常用且成熟的方法。随着结构生物学以及生物信息学的发展,大大增加了数据库中的酶基因序列与蛋白结构信息。基于宏基因组和数据库的挖掘,结合高通量筛选方法,在新型食品酶的发现和筛选方面具有极大的发展潜力。
从天然环境中或借助宏基因组技术筛选出新型食品酶基因,借助高效表达菌株对该酶基因进行表达,可有效提高酶的产量,为获得高性能的新型食品酶奠定基础(图1)。目前,许多革兰氏阳性菌被用于构建高效的蛋白表达系统。这些菌包括枯草芽孢杆菌、谷氨酸棒状杆菌、地衣芽孢杆菌和短芽孢杆菌等[3]。此外,一些真菌,如解脂耶氏酵母、巴斯德毕赤酵母、李氏木霉等,也是蛋白异源表达的理想宿主,被广泛应用于食品酶的高效生产。在使用时应注意,生产宿主必须是经GB 2760-2041 《食品安全国家标准 食品添加剂使用标准》批准使用的微生物。许多工业微生物经过改造,以高效分泌目标食品酶蛋白,并减少副产物的产生。理想情况下,微生物能够在较短时间内大量产生目标酶蛋白,酶由微生物细胞分泌至胞外,回收和纯化过程比细胞内酶的生产过程简单。因此,有必要阐明新型食品酶高效筛选和分泌表达的生物学基础,为后续高效筛选新型食品酶和构建高效表达菌株提供理论依据。
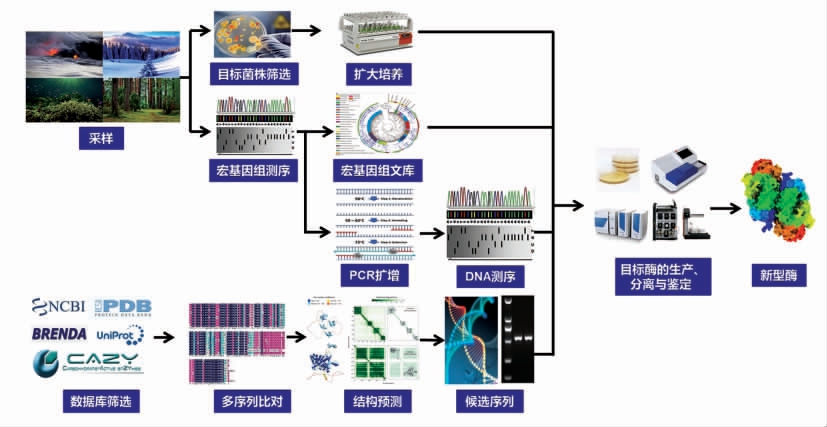
图1 新酶挖掘与筛选方法示意图
Fig.1 Schematic diagram of the mining and screening methods for novel enzymes
1.2 主要任务
1.2.1 基于微生物培养筛选的生物学基础 从自然界中分离筛选产酶菌株,是最早和最常用的新型食品酶基因挖掘方法。为了获得具有特殊生长特性的微生物,通过对于采集的混合微生物样品进行富集培养,分离筛选出具有较高催化活性的菌株,再从菌株中获得目标酶基因。菌种的筛选目的决定了样品采集的环境,需根据所需酶的性质与微生物表型的关系,考虑目标产酶菌株可能存在的环境。微生物的富集培养是指通过控制培养基的营养成分(碳源、氮源、维生素和无机盐类等),在可控的培养条件下(温度、pH 值、空气、光照等)对采集的样品进行一次或多次培养,使具有所需特征的微生物得到大量繁殖,同时使其余微生物的生长受到抑制,从而提高目标产酶菌株的比例。例如,使用淀粉作为唯一碳源,可以有效富集产淀粉酶类的微生物。
一些微生物在极端pH 值、温度、酸度、碱度的环境下,进化出具有独特催化性能的酶。从极端微生物中开发新型食品酶,有利于拓宽酶的应用范围,实现新型食品酶的工业化应用。近年来,极端环境(如火山口、深海、含硫温泉和冰川土壤等)中的微生物成为开发新型食品酶的一类重要资源。在极端环境下,酶的稳定性常因原始来源菌株的种类及其生存条件的不同而具有较大差异。了解与分析极端微生物酶的特殊性质与其结构-功能之间的关系,对于开发具有理想性质的新型食品酶尤为重要。例如,在工业应用中,酯酶需要具备有机溶剂耐受性以发挥生物活性,而酶的这一功能特性与蛋白结构表面带有大量负电荷的氨基酸残基有关,和嗜盐菌耐受高盐环境的机制类似,因此有潜力从此类微生物中挖掘有机溶剂耐受酶[4]。
目前,挖掘极端微生物新酶可对工业应用潜力较大的酶,如淀粉酶、蛋白酶、水解酶等进行筛选,其主要方法是传统的培养筛选方法,然而,许多极端微生物不适于标准化培养方式,通常需要依靠宏基因组技术以促进极端微生物中新型食品酶的筛选[5]。
1.2.2 基于宏基因组和数据库筛选的生物学基础
宏基因组技术可直接从环境中分离宏基因组DNA(全部微小生物基因组DNA 的总和),而不需要微生物的培养,能有效弥补传统新酶筛选方法的不足[6],主要包括以下步骤:1)从环境样本提取DNA,构建到合适的表达载体;2)转化易于培养的细菌,获得宏基因组文库;3)分离筛选,筛选方法包括基于功能的筛选和基于序列的筛选[7]。
基于功能的宏基因组文库筛选依赖于基因产物在宿主机体中的成功异源表达,根据酶的活性进行筛选。该方法的优点是不需要了解序列相关信息,直接进行筛选,可以获得与现有基因序列无任何相似性的新基因序列。基于活性功能的筛选策略,对于准确检测阳性克隆至关重要。常用的筛查方法包括基于表型的检测,基于宏基因组文库克隆的异源互补检测和基于诱导报告基因表达的检测。其中,表型检测是指利用与目标酶的底物相关的显色物质筛查酶的活性,属于经典的平板检出方法。此外,在液体培养基中添加与底物耦联的荧光物质,基于高通量筛选技术可以通过荧光信号进行筛选。异源互补策略是指向宏基因组插入的基因包含表达宿主生存生长所需要的必须基因,在选择条件下,只有具有互补能力基因的宿主才能够存活下来。基于诱导报告基因表达的检测属于对酶活性的间接筛选,在宏基因组文库表达的基础上,诱导报告基因在宿主细胞表达(底物或产物诱导)。此外,不同的筛选方法包括使用底物的种类以及检测步骤,对检测结果会有较大影响。因此,根据所需的酶学性质选择底物和检出方法的最佳组合至关重要,这也是高效、准确鉴定新型食品酶的基础[8]。
基于序列的筛选方法是指从宏基因组文库中利用简并引物PCR 或DNA 探针获得目标基因。该策略利用宏基因组文库或数据库获取酶的同源序列,将其克隆到合适的表达载体中,最后进行酶的活性筛选[9]。例如,Santana-Pereira 等[10]使用土壤微生物构建了一个大型插入的宏基因组文库,基于PCR 或下一代测序(Next-generation sequencing,NGS)技术的多重策略,结合生物信息学分析,从19 200 个克隆中鉴定出223 个阳性克隆(1.2%),发现两种新型酶的基因簇,分别是聚酮合成酶和非核糖体肽合成酶[10]。此外,通过此种方法已发现多种酶,如双加氧酶、几丁质酶、醇脱氢酶、甘油脱水酶、羰基还原酶等。基于序列的筛选方法中,使用的杂交DNA 探针是根据已知蛋白质家族的保守区域设计的。因此基于同源序列的方法是依赖基因组数据的准确性和完整性,且该策略局限于筛选与已知蛋白序列同源性较高的酶家族,可能不易发现新型食品酶基因。
随着NGS 技术与宏基因组技术的快速发展,基因组序列和蛋白结构的数据规模以指数增长的形式膨胀,为发现新型食品酶基因序列提供了基础。目前,针对酶的基因序列、蛋白功能和蛋白结构信息,研究人员设计了各种类型的蛋白资源数据库,极大地促进了新型食品酶基因的挖掘和筛选工作。其中UniProt 是信息最为丰富的蛋白数据库,该数据库中通过与其它蛋白数据库中结构域的序列进行同源比对获得功能注释。为了提高筛选的成功率,研究人员进一步开发了新的算法,对候选序列进行酶蛋白聚类分析和优先化排序,从而利用信息资源整合对未知蛋白进行序列、进化、功能和结构多方面综合分析,为序列空间内未知区域的探索提供了理论指导[11-12]。对于初步获得的大量候选序列,通常需要开展大量试验进行验证。因此,基于酶的可溶性表达和底物选择性等性质开发的优先化排序标准与算法,有助于提高新型食品酶的筛选效率,加速新型食品酶制剂的开发和应用。未来,基于计算机的数据库筛选方法,也将进一步促进新型食品酶的挖掘工作。
1.2.3 重组酶高效表达体系构建的生物学基础微生物体内的蛋白质合成遵循“中心法则”,通过调控细胞代谢的高效、有序进行,可控制酶蛋白的丰度和种类。基于这一原理,研究者通过对一些性能优良的天然菌株进行改造,设计一系列能够高水平表达优质食品酶蛋白的微生物细胞工厂,以满足不同食品加工场景的应用需求。
1)表达元件优化 重组酶基因的表达元件优化主要包括启动子、分泌信号肽、重组蛋白基因和转录终止子等策略,不同策略的合理使用,可以显著提高重组酶蛋白的表达量。例如,通过挖掘或改造启动子,使其调控转录能力增强,可显著提升目的基因的mRNA 转录水平和相关蛋白表达量。此外,不同宿主细胞基因密码子偏好性直接影响异源基因的表达量,通过优化重组酶基因的密码子,可以提高优质食品酶在大肠杆菌、枯草芽孢杆菌、毕赤酵母等宿主中的蛋白表达量[13-15]。转录终止子直接影响mRNA 的转录和降解效率,通过人工设计合成的转录终止子,可以有效提高重组蛋白在酿酒酵母等微生物中的表达水平[16]。重组蛋白的N 端分泌信号肽序列的主要作用是引导蛋白在折叠完成后被识别并转运至细胞外,这对于实现重组蛋白的分泌表达至关重要。有研究发现,删除信号肽的部分氨基酸序列可以促进重组酶在毕赤酵母中的异源表达和胞外分泌[17]。
2)提高重组酶基因表达量 增加重组酶基因的拷贝数是促进优质食品酶蛋白表达有效策略,总体上可分为两类方法,即体外构建法和体内构建法。在一定的拷贝数范围,重组酶蛋白的分泌表达水平与拷贝数呈线性正相关的关系,然而过高的拷贝数可能会增加微生物细胞的代谢负担,限制细胞加工通量和分泌运输通量,导致重组酶蛋白的表达量降低[18]。因此,有必要充分考虑不同食品酶蛋白基因的特征,据此选择合适的拷贝数。
3)优化蛋白分泌运输途径 蛋白的分泌表达是一个涉及跨膜运输的复杂过程,在这一过程中,蛋白与细胞膜发生相互作用。在明确相关作用机制的基础上,利用此特性进一步增强细胞膜的通透性,对于促进蛋白的可溶性表达具有重要意义。以大肠杆菌为例,外源蛋白在大肠杆菌内的表达过程中容易受到宿主细胞蛋白酶降解或形成包涵体的影响。为了促进蛋白分泌,可以采取一些策略来提高大肠杆菌的细胞膜通透性,如在发酵培养基中添加甘氨酸、表面活性剂和金属离子等外源添加剂。此外,通过促进酶分泌途径的关键蛋白质过量表达,合理增加分泌运输通量,能够有效提高重组酶蛋白在宿主细胞中的分泌表达水平[19]。
2 高端高值食品酶的催化机制
2.1 发展现状及需求分析
随着生命科学与技术的不断发展,研究者对生物酶的认识更加深入,酶的分离纯化手段也更加高效和丰富,使得应用的酶种类大幅增加。根据国际生物化学与分子生物学联盟对酶催化反应类型的分类,酶可分为氧化水解酶、异构酶、还原酶、裂解酶、转移酶、连接酶6 大类[20]。大多数工业酶制剂主要催化水解反应,用于降解各种天然组分,具有高效、专一、条件温和以及催化活性可控等优势,被广泛应用于各类食品的生产和加工过程中。例如,蛋白酶主要在乳制品行业中广泛用于水解蛋白质,淀粉酶则主要应用于淀粉的酶解转化等场景。
酶促反应属于特定的催化作用,每种酶只能对一类或者一种结构相似的特定底物进行催化作用,例如蛋白质水解反应主要由蛋白酶催化。在食品工业实际应用中,选择酶制剂需要考虑其底物偏好性、产物特异性、反应速率、热稳定性、pH 值稳定性以及对底物的亲和力等催化特性。近年来,结构生物学的快速发展为食品酶结构及催化机制的解析奠定了坚实基础。例如:大多数脂肪酶属于α/β 水解酶,其活性位点是由Ser、His 和Asp 残基组成的催化三联体构成,周围存在Gly-X-Ser-XGly 组成的五肽结构,具有一个α 螺旋形成的“盖子”结构,其外表面相对亲水,内表面则相对疏水,该结构直接影响酶活性和酶对底物的特异性和稳定性[21]。褐藻胶裂解酶具有β 果冻卷、(α/α)n 桶状结构、β 螺旋结构3 种类型的结构,通过β 消除的方式来降解褐藻胶,根据催化过程中有无金属离子的辅助,可分为金属离子辅助型β 消除和His(Tyr)/Tyr 型β 消除两类催化机制,分别具有各自的应用场景[22]。淀粉酶主要由结构域A、结构域B、结构域C 组成核心结构,还具有数量可变的氨基端结构域或羧基端端结构域,各个结构域在酶催化过协同工作,进而完成酶对底物催化的整个过程,并决定酶的催化特性[23]。因此,明确食品酶,尤其是高端高值食品酶的结构和催化机制,有助于深入了解酶的使用性能,并为其性能改造提供理论依据。
2.2 主要任务
2.2.1 高端高值食品酶的蛋白结构解析 随着结构生物学的发展,蛋白结构的解析技术也得到极大的进步,不仅提供了一个展示高端高值食品酶三维结构获得晶体结构数据的方法,也为高端高值食品酶的改造提供了分子层面的信息。目前获得脂肪酶蛋白三维结构的方法包括冷冻电镜三维重构技术、X-射线衍射晶体学、核磁共振光谱成像、AlphaFold2 和RoseTTAFold 同源建模等[24-27]。
其中,通过冷冻电镜获得蛋白结构的优点是:所需的蛋白样品较少,无需获得蛋白的晶体结构,适合解析展示处于不同功能时的蛋白结构,然而对蛋白样品的制备要求较高。通过核磁共振可以获得蛋白在其生理环境中时的一系列动态的晶体结构,其分辨率可达到X-射线级别,然而主要适合分子质量相对小的蛋白,应用范围较小。AlphaFold2 和RoseTTAFold 主要是利用人工智能计算蛋白的结构,其最大优点是仅需氨基酸序列即可预测蛋白结构[28],是最近发展起来的解析蛋白结构的新技术,然而所获得的结果有时存在一定偏差。
X-射线衍射是发展最早也是最成熟的技术,其主要优点是极高的空间分辨率,是获得蛋白结构使用最普遍的技术,在蛋白结构数据库中约90%以上的蛋白结构是由X-射线衍射获得[29]。X-射线衍射获得蛋白结构的过程主要包括蛋白晶体的制备,X-射线衍射和数据收集,相位解析等,其难点在于尺寸适合的晶体的获得和相位解析,需要在获得晶体的前提下才能获得其晶体数据并解析蛋白结构。由于X-射线的高分辨率,因此理论上X-射线衍射适合于解析任何分子质量大小的蛋白[30]。到目前为止,蛋白质结构数据库中通过X-射线衍射获得的蛋白结构数目呈指数生长,多于冷冻电镜和核磁共振获得的蛋白结构,是解析高端高值食品酶的蛋白结构的重要技术。
2.2.2 高端高值食品酶的功能和催化机制解析酶蛋白的结构可分为活性中心、远活性区和外层,活性中心决定了酶的催化特性和应用价值,活性区口袋的形状决定了底物选择性和结合模式;远活性区的氨基酸虽不直接与底物发生作用,但可能间接影响底物结合区的性质;外层的区域与溶剂或者其它蛋白质接触,决定了热稳定性等酶学性质[31]。因此,酶的结构功能研究是生命科学领域中的核心问题,许多重大科学问题的揭示和酶应用场景的挖掘都依赖于结构-功能构效关系和酶催化机制的认知。通过计算机辅助方法,如同源比对、分子对接以及动力学模拟等,可以深入研究高端高值食品酶的结构特征和催化过程中的构象变化,有助于了解其催化机制和结构-功能关系,为高端高值食品酶的性能优化和工业应用奠定基础。
1)生物信息同源比对 随着Genbank 数据库基因序列和蛋白质结构数据库PDB(Protein data bank)中的结构信息不断丰富,生物信息同源比对为高端高值食品酶的结构与功能关系研究提供了有效策略,主要方法包括多序列比对、系统进化分析和结构比对分析。基于高端高值食品酶家族的系统进化分析,可以通过同源序列或结构的比较来分析进化上的远近,鉴定保守性氨基酸以及共同进化的重要氨基酸,成为研究高端高值食品酶蛋白序列保守性和活性位点的标准方法之一。例如,有研究者对糖苷水解酶5(Glycoside hydrolase 5,GH5)家族的芦丁糖苷酶AnRut 及其同源酶进行系统进化分析,发现具有较高同源性的糖苷酶,通过进一步的多序列比对确定了AnRut 在底物结合和催化功能发挥中关键活性位点的参与[32]。此外,结构比对分析也是高端高值食品酶结构研究中的重要手段,通过叠合比较同源蛋白的三维结构,可系统分析并进一步揭示引起同源蛋白功能差异的结构基础。例如,Schiano-Di-Cola 等[33]对GH7 家族超嗜热内切葡聚糖酶Re-Cel7B 与同源蛋白的结构进行比对分析,结果显示ReCel7B 结构表面的loop 环具有较高的刚度,并且存在较多的糖基化位点,这些特征是ReCel7B具有耐高温性质的结构基础。
2)结构预测与分子对接 计算机辅助结构预测主要是利用已知目标氨基酸序列,构建蛋白质的空间结构模型,主要包括同源建模法、从头计算法、穿线法等方法。在尚未获得高端高值食品酶的晶体结构时,研究者通常采用已解析蛋白质的同源结构作为模板,进行三维结构模型的模拟预测,进而在此基础上,分析高端高值食品酶的结构-功能关系及催化机制[34-35]。这一方法可弥补蛋白晶体结构解析周期长、成本高等不足。针对从结构模拟获取的三维结构,可利用分子对接技术,进一步分析高端高值食品酶的结构特征,并通过受体结构和配体结构间的几何互补、能量匹配来实现酶-底物复合物的对接,预测高端高值食品酶发挥催化功能的关键位点[36]。例如,Zhang 等[37]对大肠杆菌β-葡萄糖醛酸苷酶进行分子对接和结构分析,发现559 位甘氨酸在催化过程中起着关键作用,并据此设计突变体G559S,其活性比野生型提高2~6 倍。
3)分子动力学模拟辅助催化机制解析 高端高值食品酶的催化功能与其蛋白结构的构象变化密切相关,然而其蛋白结构具有柔性,这增加了深入了解高端高值食品酶催化机制的难度。分子动力学模拟技术是解决这一问题的有效手段,其基于经典分子力学力场模拟生物大分子的原子运动,通过模拟高端高值食品酶在识别和催化底物过程中的构象变化,预测决定高端高值食品酶催化分子机制的关键位点,可实现配体结合影响高端高值食品酶蛋白构象变化的微观过程可视化,有助于从原子水平上明确酶-配体识别和结合机制。尤其是近年来,随着计算机硬件和人工智能的迅速发展,分子动力学模拟的功能变得更加强大,操作也更加便捷,进一步巩固了其在高端高值食品酶蛋白质结构、功能和催化机制等研究中的重要地位[38]。
4)多尺度组合模拟辅助催化机制解析 近年来,研究人员在酶工程领域的研究中引入量子力学(Quantum mechanics,QM)及量子-分子力学(Quantum mechanics/molecular mechanics,QM/MM)组合模型化学计算方法,为高端高值食品酶的催化理论研究提供了精准的预测和验证工具[39]。在全酶环境下进行的QM/MM 模拟体系,通常需要对活性中心区域进行QM 计算,并对整个酶进行分子力场MM 计算,这有助于明确底物糖环构象动力学对高端高值食品酶催化机制的影响[40]。通过跟踪高端高值食品酶催化过程中细微的原子运动,可揭示传统试验技术难以捕捉到的机理细节[41],进而有助于从原子水平认知高端高值食品酶的结构功能特征和催化机制,为高端高值食品酶的功能解析和设计改造提供更为精确的计算指导[42]。
随着计算机运算能力的不断提升和新算法在食品酶工程研究中的广泛应用,计算模拟技术将更加理性和准确地用于高端高值食品酶的结构预测、催化机制解析、构象动力学模拟以及分子设计和理性改造。此外,机器学习算法也为酶工程研究提供了全新手段。随着人工智能技术不断渗入酶工程研究领域,高端高值食品酶的功能和催化机制解析将变得更加高效和便捷。
3 食品酶催化性能改造的结构基础
3.1 发展现状及需求分析
大多数天然酶具有稳定性差,催化效率低,选择性不足等缺陷,限制了其在食品中的工业化应用。利用生物技术手段设计并改造食品酶的结构,从而改善食品酶的使用性能,成为近年来食品酶工程领域的研究热点。随着计算机模拟、序列分析、基因突变与合成以及蛋白质工程等技术交叉融合,研究人员对食品酶结构和催化性能的改造更具有针对性,使食品酶使用性能调控进入了主动时代,有助于克服天然酶性能和应用的局限性。
酶蛋白的分子改造方法主要包括理性设计和非理性设计。其中,理性设计是在充分了解食品酶蛋白的结构信息以及结构-功能关系的基础上,准确鉴定出潜在的与稳定性密切相关的残基或者结构区域,再通过基因改造的手段,如定点突变、片段插入敲除等方法对这些残基和结构进行精确的设计,从而改造食品酶的催化性能(图2)。理性设计虽然所针对的点较少,筛选方便,效率高,但是需要对酶蛋白结构和功能的关系有着清晰的了解。晶体解析技术和生物信息学的飞速发展,也为酶理性设计提供了新思路、新工具。
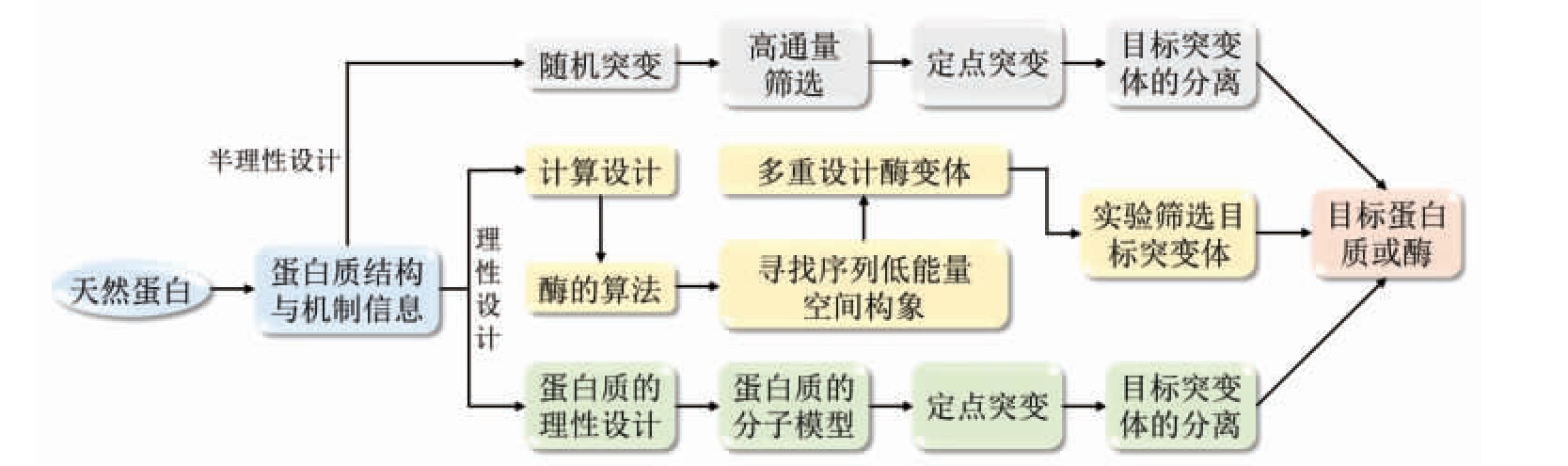
图2 酶分子改造的方法示意图[43]
Fig.2 Schematic diagram of the method for the modification of the enzyme molecule[43]
3.2 主要任务
3.2.1 基于序列的理性设计 相比于蛋白质三维结构的解析,目前对于蛋白质一级序列的测定技术手段更为成熟和高效,易于获得有效结果。因此,研究人员通常将酶蛋白序列信息和其相应的功能结合分析,建立序列-功能关系数据库,从而对一级序列信息相似的酶功能进行预测和设计,这种对酶的理性设计方法即基于序列的理性设计[44]。在设计具体某一种食品酶的特定理化性质时,需要首先挖掘关键序列区段与其功能性质之间的构效关系,进而通过搜索相应序列模式,获得具有目标性质的食品酶。
3.2.2 基于结构的理性设计 食品酶的蛋白结构是决定其催化活性、底物选择性、产物特异性和稳定性等性质的基础。其中,催化活性氨基酸和底物结合口袋(Substrate binding pocket)是食品酶催化底物的结构基础,通过深入研究并阐明这些关键氨基酸与底物相互作用的机制,有助于理性设计关键点和突变方向,从而有效改善食品酶的使用性能[45],甚至可以实现不同非活性蛋白的食品酶分子化和不同功能食品酶之间的转换。此外,食品酶分子中还存在其它结构区域,如小分子进入活性中心的底物通道,控制蛋白活性区域与溶剂接触的盖子区域等,与食品酶的催化性质也密切关联,可以作为理性设计和分子改造的目标,有助于调控底物和蛋白形成复合物的过程,从而提升酶的催化效率[46]。例如,Yang 等[47]通过对位于活性位点的催化核心中的甲硫氨酸残基进行基于结构的工程改造,使侧链尺寸和活性位点的空间位阻增加,提高了来自解淀粉碱单胞菌(Alkalimonas amylolytica)的碱性淀粉酶的氧化稳定性。
3.2.3 计算机模拟辅助的理性设计 通过计算机模拟食品酶与底物的结合过程,选择性调节酶-底物结合口袋的形状和大小,可有效调控食品酶对底物的专一性。这种计算机辅助的理性设计,通过引入一种或多种算法,对大量食品酶的氨基酸序列的结构信息进行分析计算和排序,相对于基于试验结果设计的传统方法,对于预测关键残基以及相关突变体的催化特性更加精准、可控,具有高效、经济的优势[48]。例如,李玉等[49]通过分子动力学模拟发现,野生型碱性蛋白酶结构中Gly97-Gly102 环状结构(loop)区域能够与底物分子接触并相互作用,从而影响其催化功能,进而运用Hot-Spot Wizard 3.0 设计了酶在Gly97-Gly102 loop 区域的突变库,最终筛选得到G97E 突变体,其胶原蛋白降解活力相比于野生型碱性蛋白酶显著降低。
3.2.4 深度学习辅助的理性设计 蛋白质一级序列测定技术的成熟和完善,加速了人类对蛋白质一级结构的探索和研究,这个涵盖了上亿条蛋白质一级序列的庞大数据库中,隐藏了复杂的蛋白质氨基酸排列和进化的规律,亟待深入挖掘。随着近年来计算机硬件的飞速发展,研究者能够运用深度学习网络提取其中的特征信息,以高效完成食品酶的功能结构预测、分子理性设计等下游任务[50]。在人工智能的辅助下,食品酶工程将实现相比于传统方法成本更小、试验周期更短的理性设计和改造策略。
3.2.5 酶蛋白的从头设计 蛋白质的从头设计是更加前沿的食品酶理性设计方法,其目的是创造出没有被人类所发现或自然界中不存在的食品酶蛋白,以完成特定的催化功能。计算机模拟技术和深度学习网络的发展,也为酶蛋白骨架的精确预测、模拟以及从头设计提供了坚实的技术保障。这种方法打破了传统蛋白质工程研究对象的限制,成为相关领域的研究热点。目前最大的蛋白质语言模型的参数量已达到150 亿,以应对大规模基因测序带来的蛋白质序列数据库的爆炸式增长[51]。然而,目前完全基于结构且设计出具有生物催化功能的蛋白质成功案例很少[52],针对食品酶的从头设计研究更为匮乏。食品酶工程领域的人工智能方法,仍然需要在预测精度和学习蛋白质上位性效应等方面作出突破。
4 食品酶创制高端配料的内在规律及作用机制
4.1 发展现状及需求分析
食品产业是民生产业,我国国民经济第一大产业,在过去10 余年虽稳健发展,但也面临着巨大发展压力。例如,我国食品配料行业的发展较发达国家落后,高端配料的自主率低,80%以上的婴幼儿乳蛋白、直投式发酵剂、母乳寡糖等高端食品配料被国外企业垄断,主要原因在于缺乏高端配料开发的核心技术。
酶催化技术具有绿色高效,低碳可持续,安全性高,易于控制的特点,目前正快速渗透到食品行业,在推动食品产业转型升级,培育新业态,打造产业高质量发展新动能等方面具有重要作用,有助于实现高端配料的创制和绿色高效生产,成为深入推进“制造强国”“健康中国”等战略的重要抓手。利用新型酶制剂以生物基原料生产高端食品配料,具有产量高,成本低,安全性高,节能减排等特点,且产品被视为“天然的”,是目前最具竞争力的生产方式之一。研究食品酶创制高端配料的内在规律及作用机制,已成为酶工程技术领域的热点课题。
4.2 主要任务
4.2.1 功能性油脂酶法制备的内在规律及机制天然油脂的组成和结构不能完美地满足未来食品工业及人民日益增长的美好生活需要对油脂功能或质构的所有需求,通常需要对油脂进行改性。近年来,通过对油脂进行改性,开发出易消化,调节能量代谢,具有功能性成分的结构脂质,已经成为油脂研究领域的新的热点[53]。其中酶法改性是实现功能性油脂的安全制备的有效技术解决方案之一。脂肪酶是油脂酶法改性工艺使用的主要酶类,其具有良好的特异性和异相反应能力,作用位点在甘油三酯的羧酸酯键,得到广泛应用。
不同的脂肪酶可以定向调整脂肪酸分子的迁移和骨架的构型变化,改性得到的功能性油脂具有特殊的分子结构与骨架,其结构不同,生理代谢功能不同。以甘油二酯为例,具有改善脂代谢功能,目前通常采用甘油酶解法和酯化法两种工艺,常存在产物中甘油二酯纯度低,副产物甘油三酯难于分离,需添加有机溶剂等问题[54]。有研究者通过探究脂肪酶催化特异性,创新水解-酯化两步酶法耦联的全酶法制备工艺,同时得到中等纯度(56.69%)和高纯度(98.11%)的甘油二酯,原料利用率高,副产物少[55]。再如中链脂肪酸甘油三酯,其是分子结构上的碳原子数介于短链脂肪酸和长链脂肪酸之间的一类脂肪酸,具有迅速为人体供能和不造成脂肪堆积的功能[56]。中链脂肪酸甘油三酯主要由脂肪酶催化合成,脂肪酶是一种界面催化酶,体系中需要存在少量的水来保持油-水界面,然而过量的水又会造成水解反应占主导地位,产生甘油、脂肪酸、单甘酯和甘油二酯等副产物[55],因此利用脂肪酶催化酯化反应时需要合理控制反应体系中的水分含量。
4.2.2 功能糖酶法制备的内在规律及机制 功能糖是一类不能被人体肠胃消化吸收且具有促进益生菌生长等特殊功效的碳水化合物,包括功能性稀少糖、功能性糖醇、功能性低聚糖、功能性多糖等,可以作为功能性食品添加剂或蔗糖的替代原料,满足糖尿病、肥胖等代谢异常人群的营养需求[57]。在功能性低聚糖的传统生产工艺中,多种酶制剂通常依次作用于原料,生产效率低下,且容易引起产物抑制,严重制约酶反应效率和效果[58]。此外,由于酶产物特异性差等原因,因此酶促水解生产功能性低聚糖的方法,通常获得多个聚合度的混合物,需要复杂的分离纯化工艺才能获得特定聚合度的功能性低聚糖,不仅增加了生产成本,而且造成副产物利用率低。
革新功能性低聚糖的酶法生产技术,提高多酶协同催化效率,降低产物抑制,实现酶解产物全利用,有必要分析不同种类食品酶在生产功能性低聚糖过程中的协同催化机制,利用酶结构解析、理性设计、高通量筛选等技术手段,对酶进行分子改造,优化多酶耦联应用的适应性和协同效应,从而构建高效的多酶耦联催化体系。此外,实现产物的定向调控,有必要明确酶促反应过程中的产物抑制机制,以及相应的调控策略,从而有效提高目标产品的得率和纯度。最终,明确糖链的吸附特性,针对不同低聚糖的结构和性质,开发定向分离纯化技术,实现不同类型、不同功能低聚糖的分级利用。
4.2.3 活性肽酶法制备的内在规律及机制 活性肽又称功能肽,可由蛋白酶酶解蛋白质制得。酶解法制备活性肽时,原料和蛋白酶种类的差异直接影响酶解产物,水解度的不同直接影响酶解产物的分子质量,不同原料以及不同酶解方式生产的活性肽的功能不同。研究表明,相比高分子质量的肽段,低分子质量的肽段活性更高,主要是由于小肽很容易通过肠道上皮细胞,进而被人体吸收,与目标物质结合发挥作用。活性肽的生物活性功能主要由其组成的氨基酸序列和结构特征决定,尤其与多肽链中疏水性氨基酸、芳香族氨基酸、酸性或碱性氨基酸以及含硫氨基酸的数量,在碳链中所处的位置密切相关[59],而活性肽的结构与活性之间的构效关系有待阐明。
在活性肽的酶法制备工艺中,蛋白酶的选取至关重要,不同蛋白酶的切割位点具有特异性,包括碱性蛋白酶、中性蛋白酶等微生物源蛋白酶,胃蛋白酶、胰蛋白酶等动物源蛋白酶,以及菠萝蛋白酶、木瓜蛋白酶等植物源蛋白酶[60]。因此,需要根据原料和目标产物的结构,选择合适蛋白酶,也可以针对不同的底物,采用具有不同特异性的蛋白酶复配,实现协同增效,有助于缩短酶解时间,提高水解度,进而实现特定氨基酸序列、特定聚合度、特定功能活性肽的设计和高效制备。
5 展望
酶制剂是食品产业的“芯片”。我国虽然是酶制剂生产和使用大国,但是高端高值的优质食品酶多被国际公司垄断,相关产业发展“受制于人”。今后一个时期,我国食品酶工程领域发展挑战和机遇并存。亟需组织科研力量,集体攻关从酶学理论到酶工程技术的卡脖子问题,形成完整的优质食品酶制剂创制链条,实现酶制剂的国产化替代,实现高端食品配料制造的源头创新,促进食品产业的高质量发展。
基于生物信息学、细胞生物学、结构生物学等多学科手段的交叉融合创新,深入探究高端高值食品酶的催化机制,明确酶分子改造的结构基础,为食品高端配料的酶法创制提供理论基础和实践指导,也是未来食品科技的核心竞争内容。相关科学问题和关键技术的突破,将大力推动高端配料创制和食品生物加工技术的快速发展,在食品产业提质升级、绿色、低碳可持续发展和未来食品发挥更大的作用!
[1] 周北雅,郭艳东,薛雅鞠,等.功能蛋白酶催化及应用进展[J].生物加工过程,2023,21(4):419-438.ZHOU B Y,GUO Y D,XUE Y J,et al.Advances in the catalysis and application of functional proteases[J].Chinese Journal of Bioprocess Engineering,2023,21(4):419-438.
[2] FERRER M,MARTÍNEZ-ABARCA F,GOLYSHIN P N.Mining genomes and ‘metagenomes’ for novel catalysts[J].Current Opinion in Biotechnology,2005,16(6):588-593.
[3] YAO D B,ZHANG K,WU J.Available strategies for improved expression of recombinant proteins in Brevibacillus expression system:A review[J].Critical Reviews in Biotechnology,2020,40(7):1044-1058.
[4] 王伟,姚从禹,孙晶晶,等.极地微生物酶资源开发研究进展[J].极地研究,2020,32(2):264-275.WANG W,YAO C Y,SUN J J,et al.A review of novel polar microbial enzymes for industrial applications [J].Chinese Journal of Polar Research,2020,32(2):264-275.
[5] DE SANTI C,ALTERMARK B,PIERECHOD M M,et al.Characterization of a cold-active and salt tolerant esterase identified by functional screening of Arctic metagenomic libraries[J].BMC Biochemistry,2016,17(1):1.
[6] HESS M,SCZYRBA A,EGAN R,et al.Metagenomic discovery of biomass -degrading genes and genomes from cow rumen[J].Science,2011,331(6016):463-467.
[7] YANG G,DING Y S.Recent advances in biocatalyst discovery,development and applications [J].Bioorganic &Medicinal Chemistry,2014,22(20):5604-5612.
[8] LEIS B,ANGELOV A,LIEBL W.Screening and expression of genes from metagenomes[M]// SARIASLANI S,GADD G M.Advances in applied microbiology.San Diego:Academic Press,2013:1-68.
[9] 张冬寒,曹明明,李延清,等.基于序列筛选发掘宏基因组文库中的新颖卤化酶基因[J].应用与环境生物学报,2018,24(6):1301-1306.ZHANG D H,CAO M M,LI Y Q,et al.Discovery of novel halogenase genes using sequence-driven metagenomics approach[J].Chinese Journal of Applied and Environmental Biology,2018,24(6):1301-1306.
[10] SANTANA-PEREIRA A L R,SANDOVAL-POWERS M,MONSMA S,et al.Discovery of novel biosynthetic gene cluster diversity from a soil metagenomic library [J].Frontiers in Microbiology,2020,11(1):585398.
[11] RODRÍGUEZ BENÍTEZ A,NARAYAN A R H.Frontiers in biocatalysis:Profiling function across sequence space[J].ACS Central Science,2019,5(11):1747-1749.
[12] 张建志,付立豪,唐婷,等.基于合成生物学策略的酶蛋白元件规模化挖掘[J].合成生物学,2020,1(3):319-336.ZHANG J Z,FU L H,TANG T,et al.Scalable mining of proteins for biocatalysis via synthetic biology[J].Synthetic Biology Journal,2020,1(3):319-336.
[13] SRIVIDYA D,SHYAM MOHAN A H,RAO S N.Expression and purification of codon-optimized cre recombinase in E.coli[J].Protein Expression and Purification,2020,167(1):105546.
[14] KIM M S,JANG J H,KIM Y W.Overproduction of a thermostable 4-α-glucanotransferase by codon optimization at N-terminus region[J].Journal of the Science of Food and Agriculture,2013,93(11):2683-2690.
[15] YANG Z L,ZHANG Z S.Codon-optimized expression and characterization of a pH stable fungal xylanase in Pichia pastoris[J].Process Biochemistry,2017,53(1):80-87.
[16] CURRAN K A,MORSE N J,MARKHAM K A,et al.Short synthetic terminators for improved heterologous gene expression in yeast[J].ACS Synthetic Biology,2015,4(7):824-832.
[17] LIN-CEREGHINO G P,STARK C M,KIM D,et al.The effect of α-mating factor secretion signal mutations on recombinant protein expression in Pichia pastoris[J].Gene,2013,519(2):311-317.
[18] CÁMARA E,LANDES N,ALBIOL J,et al.Increased dosage of AOX1 promoter-regulated expression cassettes leads to transcription attenuation of the methanol metabolism in Pichia pastoris[J].Scientific Reports,2017,7(1):44302.
[19] HOU J,TYO KEITH E J,LIU Z H,et al.Metabolic engineering of recombinant protein secretion by Saccharomyces cerevisiae[J].FEMS Yeast Research,2012,12(5):491-510.
[20] 赵琳,宋瑞瑞,吴希,等.工业酶制剂的发展与应用研究[J].应用化工,2021,50(5):1403-1408,1413.ZHAO L,SONG R R,WU X,et al.Study on development and application of industrial enzyme preparation[J].Applied Chemical Industry,2021,50(5):1403-1408,1413.
[21] 王新伟,成高民,李蕊,等.脂肪酶在面包和馒头中应用研究进展[J].食品与发酵工业,2022,48(11):332-337.WANG X W,CHENG G M,LI R,et al.Recent application of lipase in bread and steamed bread:A review[J].Food and Fermentation Industries,2022,48(11):332-337.
[22] 李谦,胡富,宁利敏,等.褐藻胶裂解酶的结构及催化机制研究进展[J].生物加工过程,2020,18(5):592-598.LI Q,HU F,NING L M,et al.Insight into the structure and catalytic mechanism of alginate lyases[J].Chinese Journal of Bioprocess Engineering,2020,18(5):592-598.
[23]  F,MACGREGOR E A,et al.Starch-binding domains as CBM families-history,occurrence,structure,function and evolution[J].Biotechnology Advances,2019,37(8):107451.
F,MACGREGOR E A,et al.Starch-binding domains as CBM families-history,occurrence,structure,function and evolution[J].Biotechnology Advances,2019,37(8):107451.
[24] SHENG J,JI X F,ZHENG Y,et al.Expression,purification,crystallization,and diffraction analysis of a selenomethionyl lipase Lip8 from Yarrowia lipolytica[J].Preparative Biochemistry &Biotechnology,2018,48(3):213-217.
[25] LIU S,WU K,CHEN C.Obtaining protein foldability information from computational models of AlphaFold2 and RoseTTAFold[J].Computational and Structural Biotechnology Journal,2022,20:4481-4489.
[26] MCPHERSON A,LARSON S B,KALASKY A.The crystal structures of Thermomyces(Humicola)lanuginosa lipase in complex with enzymatic reactants[J].Current Enzyme Inhibition,2020,16(3):199-213.
[27] MOHARANA T R,PAL B,RAO N M.X-ray structure and characterization of a thermostable lipase from Geobacillus thermoleovorans[J].Biochemical and Biophysical Research Communications,2019,508(1):145-151.
[28] DOBSON L,SZEKERES L I,GERDÁN C,et al.TmAlphaFold database:Membrane localization and evaluation of AlphaFold2 predicted alpha -helical transmembrane protein structures[J].Nucleic Acids Research,2022,51(D1):D517-D522.
[29] 岑一欣.工程化改造酶突变体的蛋白质晶体结构及其分子机制研究[D].杭州:浙江大学,2019.CEN Y X.Study on the crystal structure and molecular mechanism of engineered enzymes [D].Hangzhou:Zhejiang University,2019.
[30] 方亚坤.嗜热玫瑰红球菌来源嗜热脂肪酶的重组表达、结构解析及底物特异性改造[D].无锡:江南大学,2023.FANG Y K.Recombinant expression,structure analysis and substrate specific modification of thermophilic lipase from Thermomicrobium roseum [D].Wuxi:Jiangnan University,2023.
[31] 韩旭,李倩,韦泓丽,等.工业应用导向的蛋白质结构与功能研究进展[J].生物工程学报,2022,38(11):4050-4067.HAN X,LI Q,WEI H L,et al.Application-oriented structure and function study of proteins:A review[J].Chinese Journal of Biotechnology,2022,38(11):4050-4067.
[32] PACHL P,KAPESOVÁ J,BRYNDA J,et al.Rutinosidase from Aspergillus niger:Crystal structure and insight into the enzymatic activity [J].FEBS Journal,2020,287(15):3315-3327.
[33] SCHIANO -DI -COLA C,KOŁACZKOWSKI B,SØRENSEN T H,et al.Structural and biochemical characterization of a family 7 highly thermostable endoglucanase from the fungus Rasamsonia emersonii[J].The FEBS Journal,2020,287(12):2577-2596.
[34] XU Y H,FENG X D,JIA J T,et al.A novel βglucuronidase from Talaromyces pinophilus Li -93 precisely hydrolyzes glycyrrhizin into glycyrrhetinic acid 3-O-mono-β-D-glucuronide[J].Applied and Environmental Microbiology,2018,84(19):e00755-18.
[35] FENG X D,LIU X F,JIA J T,et al.Enhancing the thermostability of β -glucuronidase from T.pinophilus enables the biotransformation of glycyrrhizin at elevated temperature[J].Chemical Engineering Science,2019,204(1):91-98.
[36] DADHEECH T,JAKHESARA S,CHAUHAN P S,et al.Draft genome analysis of lignocellulolytic enzymes producing Aspergillus terreus with structural insight of β-glucosidases through molecular docking approach[J].International Journal of Biological Macromolecules,2019,125(1):181-190.
[37] ZHANG X L,SITASUWAN P,HORVATH G,et al.Increased activity of β -glucuronidase variants produced by site-directed mutagenesis[J].Enzyme and Microbial Technology,2018,109(1):20-24.
[38] COLLEPARDO-GUEVARA R,HALVORSEN LEIF A,JUNG S,et al.Biomolecular modeling and simulation:A field coming of age[J].Quarterly Reviews of Biophysics,2011,44(2):191-228.
[39] 程瑞琛,刘艳丽,戴大章.计算机辅助糖苷酶分子设计与改造研究进展[J].分子催化,2020,34(5):475-483.CHENG R C,LIU Y L,DAI D Z.Recent progress in computer-aided design and engineering of glycosidases[J].Journal of Molecular Catalysis(China),2020,34(5):475-483.
[40] ARDÈVOL A,ROVIRA C.Reaction mechanisms in carbohydrate -active enzymes:Glycoside hydrolases and glycosyltransferases.Insights from ab initio quantum mechanics/molecular mechanics dynamic simulations[J].Journal of the American Chemical Society,2015,137(24):7528-7547.
[41] RAICH L,BORODKIN V,FANG W X,et al.A trapped covalent intermediate of a glycoside hydrolase on the pathway to transglycosylation.Insights from experiments and quantum mechanics/molecular mechanics simulations[J].Journal of the American Chemical Society,2016,138(10):3325-3332.
[42] WANG B J,WALTON P H,ROVIRA C.Molecular mechanisms of oxygen activation and hydrogen peroxide formation in lytic polysaccharide monooxygenases[J].ACS Catalysis,2019,9(6):4958-4969.
[43] 李兆丰,顾正彪,陈坚.淀粉生物技术[M].北京:中国轻工业出版社,2023:55.LI Z F,GU Z B,CHEN J.Starch biotechnology[M].Beijing:China Light Industry Press Ltd.,2023:55.
[44] SUPLATOV D A,BESENMATTER W,ŠVEDAS V K,et al.Bioinformatic analysis of alpha/beta-hydrolase fold enzymes reveals subfamily-specific positions responsible for discrimination of amidase and lipase activities[J].Protein Engineering,Design and Selection,2012,25(11):689-697.
[45] EBERT M C,PELLETIER J N.Computational tools for enzyme improvement:Why everyone can -and should -use them[J].Current Opinion in Chemical Biology,2017,37(1):89-96.
[46] PADHI S K,FUJII R,LEGATT G A,et al.Switching from an esterase to a hydroxynitrile lyase mechanism requires only two amino acid substitutions[J].Chemistry &Biology,2010,17(8):863-871.
[47] YANG H Q,LIU L,WANG M X,et al.Structure-based engineering of methionine residues in the catalytic cores of alkaline amylase from Alkalimonas amylolytica for improved oxidative stability[J].Applied and Environmental Microbiology,2012,78(21):7519-7526.
[48] 路福平,黄爱岚,赵蕾,等.计算机模拟在食品工业用酶改造中的应用[J].中国食品学报,2020,20(11):1-10.LU F P,HUANG A L,ZHAO L,et al.Application of computer simulation in modification of enzymes used in food industry[J].Journal of Chinese Institute of Food Science and Technology,2020,20(11):1-10.
[49] 李玉,李家霖,朱宝悦,等.理性设计降低碱性蛋白酶的胶原降解活力[J].天津科技大学学报,2021,36(3):1-5,52.LI Y,LI J L,ZHU B Y,et al.Reducing collagen degradable activity of alkaline protease by rational design[J].Journal of Tianjin University of Science &Technology,2021,36(3):1-5,52.
[50] 康里奇,谈攀,洪亮.人工智能时代下的酶工程[J].合成生物学,2023,4(3):524-534.KANG L Q,TAN P,HONG L.Enzyme engineering in the age of artificial intelligence[J].Synthetic Biology Journal,2023,4(3):524-534.
[51] LIN Z M,AKIN H,RAO R S,et al.Evolutionary-scale prediction of atomic-level protein structure with a language model [J].Science,2023,379(6637):1123-1130.
[52] SIEGEL J B,ZANGHELLINI A,LOVICK H,et al.Computational design of an enzyme catalyst for a stereoselective bimolecular diels -alder reaction [J].Science,2010,329(5989):309-313.
[53] 季圣阳,鞠兴荣,徐斐然,等.功能性油脂-结构脂质酶法合成的研究进展[J].粮食科技与经济,2018,43(10):75-78.JI S Y,JU X R,XU F R,et al.Grain science and technology and economy[J].Grain Science and Technology and Economy,2018,43(10):75-78.
[54] 刘书成,鲁海龙,张林尚,等.酶在油脂制取、精炼与改性中的应用[J].中国油脂,2023,48(12):31-39.LIU S C,LU H L,ZHANG L S,et al.Application of enzymes in oil production,refining and modification[J].China Oils and Fats,2023,48(12):31-39.
[55] 连伟帅.甘油二酯、LML 型结构脂的酶法制备与应用研究[D].广州:华南理工大学,2019.LIAN W S.Study on the enzymatic preparation and application characteristics of diacylglycerols and LML-type structured lipids[D].Guangzhou:South China University of Technology,2019.
[56] 陈倩倩,王金荣,王朋,等.中链脂肪酸的生物学功能及其在动物生产中的应用研究进展[J].中国畜牧杂志,2022,58(2):41-47.CHEN Q Q,WANG J R,WANG P,et al.Research progress on biological functions of medium chain fatty acids and its applications in animal production [J].Chinese Jouranl of Animal Science,2022,58(2):41-47.
[57] 李德茂,童胜,曾艳,等.未来食品的低碳生物制造[J].生物工程学报,2022,38(11):4311-4328.LI D M,TONG S,ZENG Y,et al.Low carbon biomanufacturing for future food[J].Chinese Journal of Biotechnology,2022,38(11):4311-4328.
[58] YANG J G,ZHANG T,TIAN C Y,et al.Multienzyme systems and recombinant cells for synthesis of valuable saccharides:Advances and perspectives[J].Biotechnology Advances,2019,37(7):107406.
[59] CHEN H M,MURAMOTO K,YAMAUCHI F.Structural analysis of antioxidative peptides from soybean β-conglycinin[J].Journal of Agricultural and Food Chemistry,1995,43(3):574-578.
[60] 张昂,徐威,郭青松.酶解食品源蛋白质制备生物活性肽的研究进展[J].食品研究与开发,2023,44(24):208-215.ZHANG A,XU W,GUO Q S.Progress in the preparation of bioactive peptides by enzymatic digestion of food-derived proteins[J].Food Research and Development,2023,44(24):208-215.